聚类算法汇总
1、K-Means聚类
步骤:①初始化K个样本点作为初始聚类质心②根据欧氏距离将其他样本点划分为相应类别③计算新的质心作为聚类中心④重复二三步骤 终止条件:最终一次聚类的质心与上一次聚类的质心差值<设定的阈值
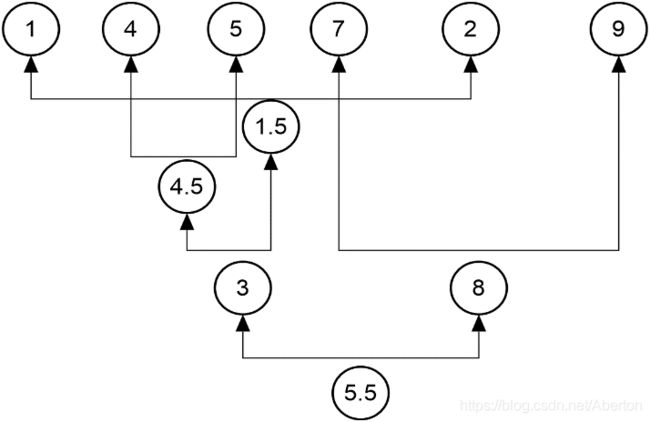
2、层次聚类(AgglomerativeClusting)
树结构聚类
聚类距离选择:最近single linkage,最远complete linkage,平均average linkage
3、密度聚类(DBSCAN)
主要的两个参数: eps半径 minpts最小样本点
备注:评价聚类算法 两个指标参数
轮廓系数:a(i)样本点到簇内中心的平均距离 b(i)样本点到其他簇中心的平均距离 取最小 系数r = (b(i)-a(i)) / max(a(i),b(i))
兰德系数; 聚类结果与实际结果相互比较 正确率
from sklearn import datasets
from sklearn.cluster import KMeans#KMeans聚类
from sklearn.cluster import AgglomerativeClustering#层次聚类
from sklearn.cluster import DBSCAN#密度聚类
import matplotlib.pyplot as plt
from sklearn import metrics
circles=datasets.make_circles(n_samples=100,noise=0.02)
data=circles[0]
label=circles[1]
km=KMeans(n_clusters=4)
km.fit(data)
pre_label=km.labels_
colors='bgrkcmy'
for p in range(len(data)):
plt.scatter(data[p,0],data[p,1],color=colors[pre_label[p]])
#plt.show()
#agg=AgglomerativeClustering(n_clusters=2,linkage='complete')
#agg.fit(data)
#pre_label=agg.labels_
#colors='bgrkcmy'
#for p in range(len(data)):
# plt.scatter(data[p,0],data[p,1],color=colors[pre_label[p]])
#plt.show()
#db=DBSCAN(eps=0.5,min_samples=3)
#db.fit(data)
#pre_label=db.labels_
#colors='bgrkcmy'
#for p in range(len(data)):
# plt.scatter(data[p,0],data[p,1],color=colors[pre_label[p]])
#plt.show()
print("轮廓系数:",metrics.silhouette_score(data,pre_label))
print("兰德系数:",metrics.adjusted_rand_score(label,pre_label))