【无标题】
【自用】nnUNet研究并努力魔改记录
目录清单
- nnUNet的安装
- nnUNet的输入数据
-
- 原始数据准备
- 数据预处理
-
- 图像裁剪
- 标准化
- 数据集的划分
- 加载数据集
-
- 数据扩增
- 模型训练
-
- 基本模型
- 训练策略
- 模型预测
- 数据后处理
nnUNet的安装
关于nnUNet的安装和使用其实网络上有很多,可以参考的链接有:
nnUNet最舒服的训练教程(让我的奶奶也会用nnUNet)link
nnUNet使用教程 link
保姆级教程:nnUnet在2维图像的训练和测试 link
在我自己使用的期间,我觉得最实用的其实就是官网的安装方式:https://github.com/MIC-DKFZ/nnUNet#installation,即:
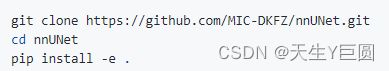 但是像我的话是在实验室的服务器上安装,并且我本人是没有root权限的,因此在使用这个命令之前,先要通过非root权限的方式去安装git,具体的方法可以参考这个链接。除此之外,由于可能存在的版本冲突,建议在新建的环境下搭建nnUNet。比如像我之前有过安装了较新的版本的一些包,结果发现最后安装完成以后居然运行报错了,再重新降低版本又安装,非常心力交瘁。
但是像我的话是在实验室的服务器上安装,并且我本人是没有root权限的,因此在使用这个命令之前,先要通过非root权限的方式去安装git,具体的方法可以参考这个链接。除此之外,由于可能存在的版本冲突,建议在新建的环境下搭建nnUNet。比如像我之前有过安装了较新的版本的一些包,结果发现最后安装完成以后居然运行报错了,再重新降低版本又安装,非常心力交瘁。
nnUNet的输入数据
原始数据准备
我的原始数据是numpy格式的,而nnUNet的输入数据要求必须是.nii.gz格式的文件,因此需要先将原始的文件转换成为nnUNet可以识别并处理的格式。具体的操作过程其实之前提到的链接中也都有涉及,就不一一赘述了,主要就是包括了以下的文件:
1. 处理好的文件放在 …/nnUNet_raw/nnUNet_raw_data/YourTaskName/ 下的 (1)其中图像的文件夹下,针对二维的图像来说,应该是(1, x, y)大小的.nii.gz文件,如果图像具有多个通道的话,则在图像名称后面加上从_0000,_0001,_0002,…等对应的后缀,并分别保存为.nii.gz格式的文件;
(1)其中图像的文件夹下,针对二维的图像来说,应该是(1, x, y)大小的.nii.gz文件,如果图像具有多个通道的话,则在图像名称后面加上从_0000,_0001,_0002,…等对应的后缀,并分别保存为.nii.gz格式的文件;
(2)对于labels文件来说,一个case就对应一个大小为(1,x,y)的mask;
2. 至于最后的dataset.json文件,其实可以通过 …/nnunet/experiment_planning/nnUNet_plan_and_preprocess.py 来生成,生成完了以后可以打开检查一下,这个文件夹下有一些标准的数据的生成文件,可以作为参考来生成或修改自己的json文件。
数据预处理
在输入了预处理命令以后(命令在文章头部分享的链接中都有,就不说了),nnUNet就会分别对原始数据进行裁剪和标准化的工作。
图像裁剪
- nnUNet针对输入的case,会首先统计一个non_zero_mask,其实就是统计了每一个通道中值为0的区域,然后对应生成一个只包含了非零区域的bounding box,再根据这个bbox对图像进行裁剪。可以认为去掉了一部分没有信息的区域;
- 裁剪后会在 …/nnUNet_raw/nnUNet_cropped_data/TaskName/下保存处理好的文件;
- 会生成如下的文件:
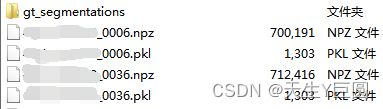
- 其中,gt_segmentations文件夹下就是所有的groundtruth的mask,并且是完全没有裁剪的,感觉就是搬运了过来;
- npz文件打开就是一个大小为(channel+1,x,y)的numpy文件
- 其中,前几层就对应了裁剪后的每个通道的图像;
- 最后一层则是case对应的一个mask,与 ground truth mask不同的是,这个mask会将那些在每个channel上都为0的像素位置赋值为-1,其他位置的像素值和ground truth的是一样的;
- 而pkl文件则是文件的信息清单:
| Information | Meaning |
|---|---|
| original_size_of_raw_data | 即图片原始的大小,value=[1, x, y] |
| original_spacing | 图片原始的像素间距,我的是 [1, 1, 1],当然你们的一般不是哈 |
| list_of_data_files | 就是对应case中所有通道对应的nii.gz文件的路径 |
| seg_file | 原始的ground truth mask存放的路径 |
| itk_origin | 不太清楚是啥,我的都是 (0.0, 0.0, 0.0) |
| itk_spacing | 应该是和original_spacing差不多,我的是 (1.0, 1.0, 1.0) |
| itk_direction | 方向,value=(1.0, 0.0, 0.0, 0.0, 1.0, 0.0, 0.0, 0.0, 1.0) |
| crop_bbox | 一个list,统计得到的非零区域的bounding box,对应了bounding box的三个端点,我的是二维数据,所以value=[[0, 1], [x1, y1], [x2, y2]] |
| classes | 一个array,图像中的类别,由于我做的是三种区域的分割,再加上了nnUNet统计了都为0的区域且赋值为-1,因此我的value=[-1. 0. 1. 2.] |
| size_after_cropping | 一个tuple,顾名思义就是裁剪了之后的尺寸,我的是二维的所以是(1, xx, yy) |
| use_nonzero_mask_for_norm | 看源码好像是和数据类别什么的有关,我的数据value=OrderedDict([(0, False), (1, False), (2, False)]) |
标准化
- 对于裁剪后的图像,进一步对其进行标准化的操作,其实就是对每个case做一个减均值除标准差;
- 做好标准化之后会在…/nnUNet_preprocessed/Task826_IVUS/下保存处理好的文件;
- 生成的文件如下:

- gt_segmentation:和 crop 之后的一样,都是搬运的原始的 ground truth mask;
- nnUNetData_plans_v2.1_2D_stage0:和 crop 之后的一样,下面都保存了 npz 文件和 pkl 文件,其中
- npz 文件保存的就是标准化之后的文件,因为是在crop的基础上进行标准化的,所以大小是和crop后的大小一样的;
- pkl 文件同样是保存了文件的相关信息,其中,crop 文件夹下的 pkl 文件内的信息全都包含了在内,除此之外还添加了新的信息,如下:
| Information | Meaning |
|---|---|
| size_after_resampling | 顾名思义是重采样后的大小,nnUNet会在crop之后将图像进行重采样,因为我的数据是各向同性的,所以 value 和 crop 后的大小是一样的 |
| spacing_after_resampling | 和上面的描述一致,我的反正是 [1. 1. 1.] |
| class_locations | 就是整理了一下每个class在图像中的位置 |
- dataset.json:保存了数据集的信息清单,如下
| Information | Meaning |
|---|---|
| description | 这我也不知道是啥,因为我的显示是一个空的string,那就不管了吧 |
| labels | 一个词典,value={‘0’ : ‘background’, ‘1’ : ‘object1’, ‘2’ : ‘object2’, …} |
| licence | 估计有些数据集会有的,不过这里是我们自己的数据集,所以value=‘nope’ |
| modality | 一个词典,用来指示每个通道的含义,比如value={‘0’ : ‘R’, ‘1’ : ‘G’, ‘2’ : ‘B’, …} |
| name | 数据集的名字(通常是疾病或者器官的名字) |
| numTest | 测试集的图像的数量(如果有多个通道的话 value=case数×通道数) |
| numTraining | 训练集的图像的数量(如果有多个通道的话 value=case数×通道数) |
| reference | 不知道是啥,我这里是一个空的字符串 |
| release | 我也不知道是啥,我这里 value = ‘0’ |
| tensorImageSize | 因为我做的是二维的多通道的数据,所以显示 value = ‘3D’,即图片是一个3D的tensor |
| test | 所有的测试图像的路径清单 |
| training | 所有的训练图像的路径清单 |
- dataset_properties.pkl保存了训练数据集的信息清单,如下:
| Information | Meaning |
|---|---|
| all_sizes | 保存了训练集所有 case 在 crop 之后的 mask 的 size |
| all_spacings | 保存了训练集所有 case 的像素间距 |
| all_classes | 类别清单(除去)了 background 后其他的类别,我的 value = [1, 2] |
| modalities | 和 dataset.json 文件中的一样 |
| intensityproperties | 不知道是啥,我的 value = None |
| size_reductions | [0, 1]的一个数值,表示了 crop前后图片尺寸减小的程度 |
- nnUNetPlansv2.1_plans_2D.pkl保存了实验的相关信息清单,如下:
| Information | Meaning |
|---|---|
| num_stages | 不知道是啥但是 value = 1 |
| num_modalities | 通道数目 |
| modalities | 一个词典,用来指示每个通道的含义,比如value={‘0’ : ‘R’, ‘1’ : ‘G’, ‘2’ : ‘B’, …} |
| normalization_schemes | 我的不在nnUNet里面常见的所有模态中,因此 value = OrderedDict([(0, ‘nonCT’), (1, ‘nonCT’), (2, ‘nonCT’)]) |
| dataset_properties | 一个词典,内容就是上面dataset_properties.pkl中的内容 |
| list_of_npz_files | crop 后的npz文件的存放路径清单 |
| original_spacings | 所有的case的原始的像素间距列表 |
| original_sizes | crop 后的所有 case 对应的 mask 的大小尺寸 |
| preprocessed_data_folder | 预处理后的文件的存放路径 |
| num_classes | 除去了background 后的类别的数量 |
| all_classes | 类别清单(除去)了 background 后其他的类别,我的 value = [1, 2] |
| base_num_features | 不知道是啥,我的 value = 32,不懂什么意思 |
| use_mask_for_norm | 不知道是啥,我的数据value=OrderedDict([(0, False), (1, False), (2, False)]) |
| keep_only_largest_region | 不知道是啥,我的 value = None |
| min_region_size_per_class | 不知道是啥,我的 value = None |
| min_size_per_class | 不知道是啥,我的 value = None |
| transpose_forward | 不知道是啥,我的 value = [0, 1, 2] |
| transpose_backward | 不知道是啥,我的 value = [0, 1, 2] |
| data_identifier | 要用什么 plan 来进行实验,和文件名一样 |
| plans_per_stage | 关于实验的一些设置 |
| preprocessor_name | 使用了那种预处理方式,我的是二维的,所以 value = ‘PreprocessorFor2D’ |
其中,对于我的数据而言,plans_per_stage的值为(单纯记录一下):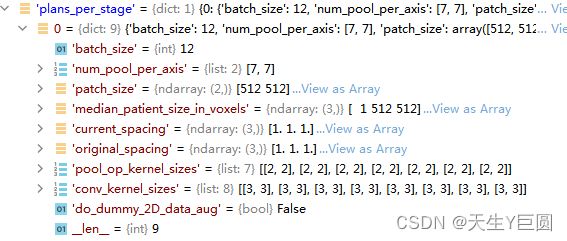
数据集的划分
- 我们知道,nnUNet在训练的过程中使用的是交叉验证的方式,因此在开始训练之前先需要对数据集进行划分。
- 原始的nnUNet是在对Trainer进行初始化的时候进行的数据集划分,代码的具体位置在
nnunet.training.network_training.network_trainer.py中的NetworkTrainer类下的 do_split 功能函数:
// do_split
splits_file = join(self.dataset_directory, "splits_final.pkl")
if not isfile(splits_file):
self.print_to_log_file("Creating new split...")
splits = []
all_keys_sorted = np.sort(list(self.dataset.keys()))
kfold = KFold(n_splits=5, shuffle=True, random_state=12345)
for i, (train_idx, test_idx) in enumerate(kfold.split(all_keys_sorted)):
train_keys = np.array(all_keys_sorted)[train_idx]
test_keys = np.array(all_keys_sorted)[test_idx]
splits.append(OrderedDict())
splits[-1]['train'] = train_keys
splits[-1]['val'] = test_keys
save_pickle(splits, splits_file)
splits = load_pickle(splits_file)
if self.fold == "all":
tr_keys = val_keys = list(self.dataset.keys())
else:
tr_keys = splits[self.fold]['train']
val_keys = splits[self.fold]['val']
tr_keys.sort()
val_keys.sort()
self.dataset_tr = OrderedDict()
for i in tr_keys:
self.dataset_tr[i] = self.dataset[i]
self.dataset_val = OrderedDict()
for i in val_keys:
self.dataset_val[i] = self.dataset[i]
- 可以看到,就是用的
sklearn.model_selection.KFold函数去生成的五折的数据; - 所以如果大家想要不采用5折的话,也可以根据自己的需求写相关的分折的代码,但是一定要记住保存生成的dataset的列表。
加载数据集
nnUNet使用的2D数据集加载的方法位于...\nnunet\training\dataloading\dataset_loading.py中的 DataLoader2D 函数类。
//nnunet\training\network_training\nnUNetTrainer.py
//get_basic_generators
dl_tr = DataLoader2D(self.dataset_tr, self.basic_generator_patch_size, self.patch_size, self.batch_size,
oversample_foreground_percent=self.oversample_foreground_percent,
pad_mode="constant", pad_sides=self.pad_all_sides, memmap_mode='r')
dl_val = DataLoader2D(self.dataset_val, self.patch_size, self.patch_size, self.batch_size,
oversample_foreground_percent=self.oversample_foreground_percent,
pad_mode="constant", pad_sides=self.pad_all_sides, memmap_mode='r')
对于我的图像来说,
self.basic_generator_patch_size:数据扩增时用到的尺寸,[602, 602];
self.patch_size:我的图像的原始尺寸,[512, 512]
self.batch_size:前面在生成 plan 的时候就指定了的 batch size, [12, 12]
oversample_foreground_percent:[0, 0.66]
- 在数据进行扩增之前,nnUNet 会对数据先进行 padding,生成 [602, 602] 大小的图像,然后进行各种数据扩增的手段,最后会通过 crop 得到最终的 [512, 512] 大小的输入图像;
- 至于 oversample 这个参数而言,表示在进行对图片进行的采样的过程中,要保证前景像素所占的最小比例。其实就是为了使得采样的图像不全是背景,让网络能够学习到更多有效的信息。
数据扩增
nnUNet数据扩增相关的内容也可以参考这篇文章 https://blog.csdn.net/weixin_44858814/article/details/124559924 具体来说,nnUNet 数据扩增部分主要有:
| Augmentation | Parameters |
|---|---|
| 弹性形变 | V2版本的 nnUNet 是不做弹性形变的 |
| 旋转 | 包含了各个方向上的旋转,在 V2 中,“rotation_x” = (-180°, 180°),“rotation_y”=“rotation_z” = (-0°, 0°) |
| 缩放 | 缩放的尺度是 (0.7, 1.4) |
| 加高斯噪声 | |
| 高斯模糊 | |
| 亮度变换 | |
| 对比度变换 | |
| 分辨率变换 | |
| gamma变换 | |
| 镜像变换 |
注:由于和尺寸相关的只有旋转和缩放,因此会在其他的操作之前有一个 crop 的操作将图片重新规范到 patch_size 的大小,而在所有的变换之后,则会将 pad 的的部分,即值为-1的部分都置0。
模型训练
基本模型
我们知道 nnUNet 顾名思义就是用 UNet 进行分割的,具体的网络结构大家可以通过查看论文原文
训练策略
结构上面就不多介绍了,论文附件中所有的相关结构都有详细的解释,这里我们主要关注模型的训练策略。
| 策略 | 设置 |
|---|---|
| 优化器 | SGD,momentum = 0.99 |
| 学习率调整策略 | poly_lr,poly_lr(ep, max_num_epochs=1000, initial_lr=1e-2, 0.9) |
其实也没啥特别的,然后存储模型的时候就会存储两个模型,一个是最优的模型,一个是最近的模型;当然如果你在参数的部分设置了每隔多少个epoch就保存一次的话,那么也会对模型进行保存;
模型预测
- 主程序位于
...\nnunet\inference\predict_simple.py - 推理的过程会运用到所有折的结果,由于我在实验中只使用一个2DUNet,所以不涉及多个模型之间的结果对比,然后选择最优的模型,而是直接使用每个折的 best model 进行一个预测;
- 在预测的过程中,使用每一折的 model 对输入图像进行预测,得到 softmax 的概率,然后对其进行平均得到最终图片的预测结果;
除此之外,nnUNet会在模型推理的过程中进行一个 sliding window 地预测,这种就是对于那些比较大的目标就比较有用,还记得 plan 中的 patch_size 吗,如果很大的话就会基于 patch 进行一个分割,当然普通的这种基于 patch 的分割常常会有棋盘状的伪影,所以 nnUNet 会在推理的时候给 patch 的中心和边缘不同的权重,而且在测试时使用镜像地数据增强
数据后处理
nnUNet 中有包含可选的后处理步骤;不过只有处理最大连通区域的操作;