【ML】主成分分析 PCA(Principal Component Analysis)原理 + 实践 (基于sklearn)
【ML】主成分分析 PCA(Principal Component Analysis)原理 + 实践 (基于sklearn)
- 原理简介
- 实践
-
- 数据集
- 数据处理
- 使用KNN模型进行分类预测(为了和PCA处理后做对比)
- 数据归一化
- 保持与原始数据维度数,观察维度方差占比
- 计算两个维度的PCA
- 再次用KNN计算PCA获取的新维度的准确度
- 总结
原理简介
PCA(Principal Component Analysis)主成分分析,顾名思义,找出一组更低维度的特征,可以代表原始特征(精度会些许降低,但计算速度会大大加快)【主要目的是降维】。注意:不是筛选出原始维度的某几个维度,新的特征维度不是之前的特征维度,而是计算出来的新的特征数据。
详细推导可以参考这篇文章:https://www.jianshu.com/p/73080c9de848
实践
数据集
鸢尾花数据集:https://www.kaggle.com/datasets/himanshunakrani/iris-dataset
数据处理
import numpy as np
import pandas as pd
# 数据处理
origin_data = pd.read_csv("/kaggle/input/iris-dataset/iris.csv")
origin_data.loc[:,'species'].value_counts()
data = origin_data.replace({'species':{'setosa':1,'versicolor':2,'virginica':3}})
X = data.drop(columns=['species'])
y = data.loc[:,'species']
使用KNN模型进行分类预测(为了和PCA处理后做对比)
# 使用knn模型进行
from sklearn.neighbors import KNeighborsClassifier
# 周围3个点作为判断条件
KNN = KNeighborsClassifier(n_neighbors=3)
KNN.fit(X,y)
y_predict = KNN.predict(X)
from sklearn.metrics import accuracy_score
accuracy = accuracy_score(y,y_predict)
print(accuracy)
输出:0.96
数据归一化
至于为什么要归一化:
- 是为了加快计算速度
- 因为需要距离计算,归一化后将各个维度量纲拍平
# 归一化
from sklearn.preprocessing import StandardScaler
X_norm = StandardScaler().fit_transform(X)
print(type(X_norm)) # 保持与原始数据维度数,观察维度方差占比
方差越小,说明数据变化越小,对结果的影响就越小
# 查看各个维度的方差
from sklearn.decomposition import PCA
# 四个维度都计算
pca = PCA(n_components=4)
x_pca = pca.fit_transform(X_norm)
print(pca.explained_variance_ratio_)
# 绘图更直观
fig2 = plt.figure(figsize=(10,5))
plt.bar([1,2,3,4],pca.explained_variance_ratio_)
# 注意已经不是原始的特征了,所以用PC1,2,3,4代替
plt.xticks([1,2,3,4],['PC1','PC2','PC3','PC4'])
plt.ylabel('pca variance ratio')
plt.show()
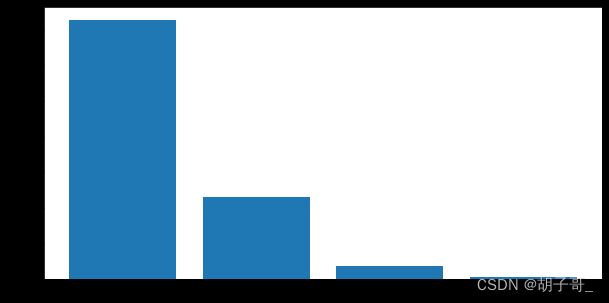
显然,有两个维度对结果影响很大,所以我们取两个维度作为特征维度。
计算两个维度的PCA
# 训练两个纬度的pca
pca_2 = PCA(n_components=2)
x_pca_2 = pca.fit_transform(X_norm)
print(type(x_pca_2))
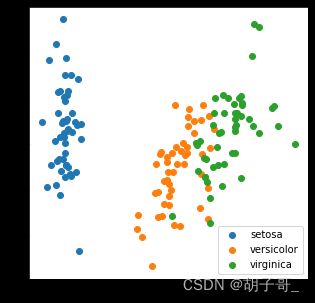
# 可视化两个维度
fig3 = plt.figure(figsize=(5,5))
setosa = plt.scatter(x_pca_2[:,0][y==1],x_pca_2[:,1][y==1])
versicolor = plt.scatter(x_pca_2[:,0][y==2],x_pca_2[:,1][y==2])
virginica = plt.scatter(x_pca_2[:,0][y==3],x_pca_2[:,1][y==3])
plt.legend((setosa,versicolor,virginica),('setosa','versicolor','virginica'))
plt.show()
再次用KNN计算PCA获取的新维度的准确度
# 再次训练,对比准确度变化
KNN_norm = KNeighborsClassifier(n_neighbors=3)
KNN_norm.fit(x_pca_2,y)
y_predict_norm = KNN_norm.predict(x_pca_2)
accuracy_norm = accuracy_score(y,y_predict_norm)
print(accuracy_norm)
输出:0.9533333333333334
总结
使用4个维度的准确率为:96%,使用PCA后的准确率是:95.3%,相差很小