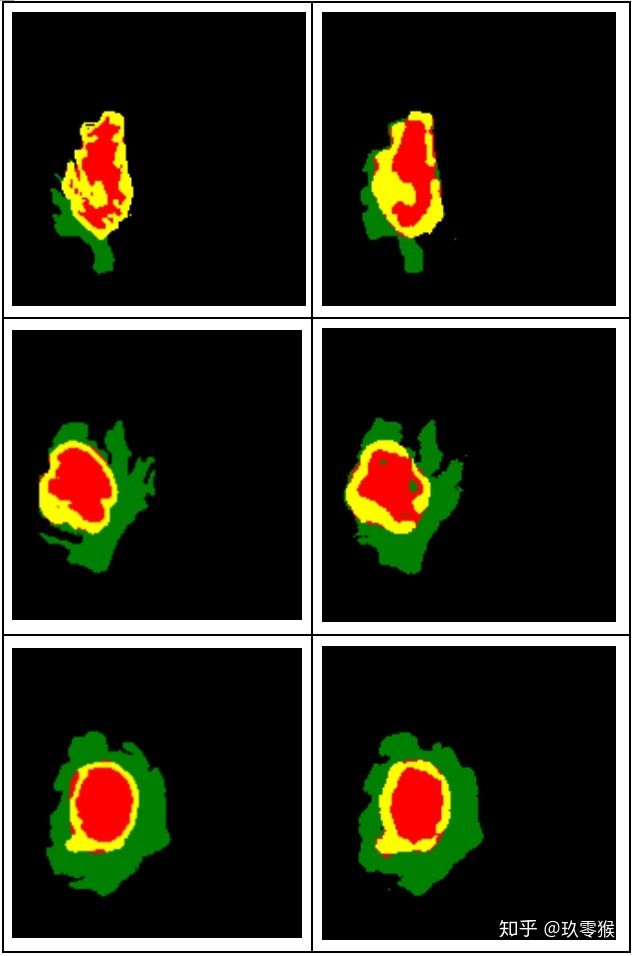
2D-FCN8s,FCN16s,FCN32s脑胶质瘤分割BraTs + Pytorch实现
FCN讲解
玖零猴:FCN+与CNN的区别+三大技术+网络结构
BraTs数据准备
数据来源
本文用的训练集和验证集均来自BraTs2018的训练集(其中HGG:210个病人,LGG:75个病人)
但由于BraTs只公开训练集数据,没有测试集数据,如果在训练集中再拆一部分用来作测试集的话,那训练集便少了许多,训练数据如果过少,容易出现过拟合现象,即在训练集中表现好,而在测试集中表现差,此时的网络泛化能力变差了.为了解决数据少的问题,灵机一动的我想出了一个办法.
因为BraTs2019的训练集在BraTs2018的基础上增多了,其中HGG增加了49例,LGG增加了1例,那么我就把这些新增的作为我的测试集
下面我提供百度云盘给大家下载,这是原始数据
BraTs18数据集下载地址(不包含测试集,提供的验证集无GT)
链接:https://pan.baidu.com/s/1Ry41OVl9VLOMzhQQR9qXuA 提取码:qvmo
BraTs19数据集下载地址如下(不包含测试集,提供的验证集无GT)
链接: https://pan.baidu.com/s/1S5XGTdHkwFnagKS-5vWYBg 提取码: 2333
数据的预处理以及实现代码
把上面两年的数据下下来,然后我对数据的预处理方法是链接
完整的实现代码(jupyter notebook打开)
下载:https://github.com/Merofine/BraTS2Dpreprocessinggithub.com
- GetTrainingSets.ipynb——>训练集和验证集
- GetTestingSetsFrom2019.ipynb-—>测试集
代码执行完后,获得npy数据
<如果大家嫌麻烦,我这里提供预处理好的npy数据>
链接:https://pan.baidu.com/s/1iIBvqrXIx2JAvoyt3FcuYw 密码:4qua
训练集、验证集和测试集——预处理之区别
它们的预处理除了是否要去除没有病灶切片外,别无区别
训练集是去除的,以缓解类别不均衡问题,类别不平衡(class-imbalance)就是指分类任务中不同类别的训练样例数目差别很大的情况,但若差别很大,则会对学习过程造成困扰.我们的任务是分割,分割是一种对像素级别的分类,一个切片假如病灶很少甚至没有,那么就会出现严重的类别不均衡,学习的时候网络就会偏向于多的那一类靠,为了缓解这种情况,应该剔除没有病灶的切片
而验证集我也是去除的,因为验证集其实在训练过程中扮演了另一角色,虽然并没有直接参与训练,可是却是为了防止过拟合现象,也就是说防止网络将这些有病灶的切片学得太过头了,这是个人理解,具体到底是否去除,还得通过实验证明
测试集当然是不用去除的,因为这个时候就是考验它的时候到了,让它自己判断是否有病灶
运行环境的安装
windows10 64 bits、nvidia驱动、CUDA8.0、cudnn、anaconda
打开命令窗口, 分别输入以下指令:
conda create -n jiu0Monkey python=3.6
conda activate jiu0Monkey
pip install simpleitk
pip install opencv-python==3.4.2.16
pip install scipy
pip install scikit-learn==0.20
pip install scikit-image==0.14
conda install numpy mkl cffi
安装pytorch,选择与cuda版本对应的进行安装,python版本也要对应
下载链接:https://mirrors.tuna.tsinghua.edu.cn/anaconda/cloud/pytorch/
我选择的是win-64 pytorch-0.4.0-py36_cuda80_cudnn7he774522_1.tar.bz2
下载完毕后进行安装,找到下载目录并执行:
conda install --offline .\pytorch-0.4.0-py36_cuda80_cudnn7he774522_1.tar.bz2
conda install torchvision -c pytorch
conda install Pillow=6.1
conda install tqdm
conda install pandas
pip install -U scikit-image
pip install -i https://pypi.tuna.tsinghua.edu.cn/simple numba
pip install hausdorff
代码下载链接
Merofine/FCN2D_For_BraTs
训练:
将train.py的img_paths和mask_paths修改为自己的trainImage和trainMask的路径
训练前会通过train_test_split函数将数据集划分为训练集和验证集,只要参数random_state一样,划分的结果就是一样的
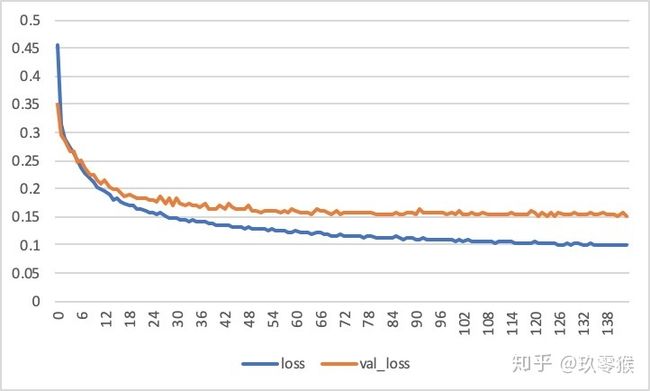
每一次epoch训练结束,都会对验证集进行测试Iou指标,如果比之前最好的还要好就保存本次训练模型,如果超过args.early_stop这个参数还没有训练更好的话,便结束训练,这个原理就是early_stop,主要还是防止网络训练过度,造成过拟合现象,这也就是验证集虽然没有直接参与训练,但是却在其中扮演了一个非常重要的角色!
如果要训练FCN8s,则运行下面指令
python .\train.py --arch="FCN8s" --dataset=“Jiu0Monkey”
此外还可以训练FCN16s、FCN32s
其它参数根据自己的情况进行配置
预训练好的模型下载:
链接:https://pan.baidu.com/s/1GvdimdO7WvjMVZJbk7Ie-w 密码:r8qo预测:
将test.py的img_paths和mask_paths修改为自己的testImage和testMask的路径
运行下面指令获得测试结果以及GT文件:
python .\test.py --name="Jiu0Monkey_FCN8s_woDS" --mode="GetPicture"
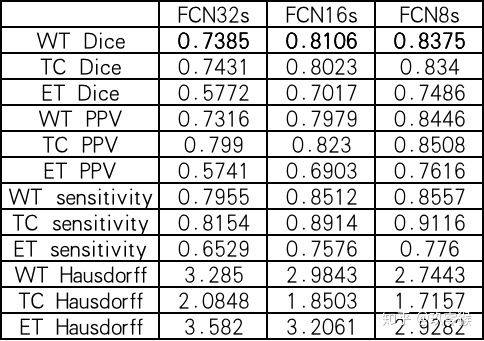
运行下面指令评价测试结果以及GT文件的指标,想了解更多指标的信息包括Dice、Hausdorff、IOU、PPV等,可以参考我这一篇(分割常用评价指标)
python .\test.py --name="Jiu0Monkey_FCN8s_woDS" --mode="Calculate"
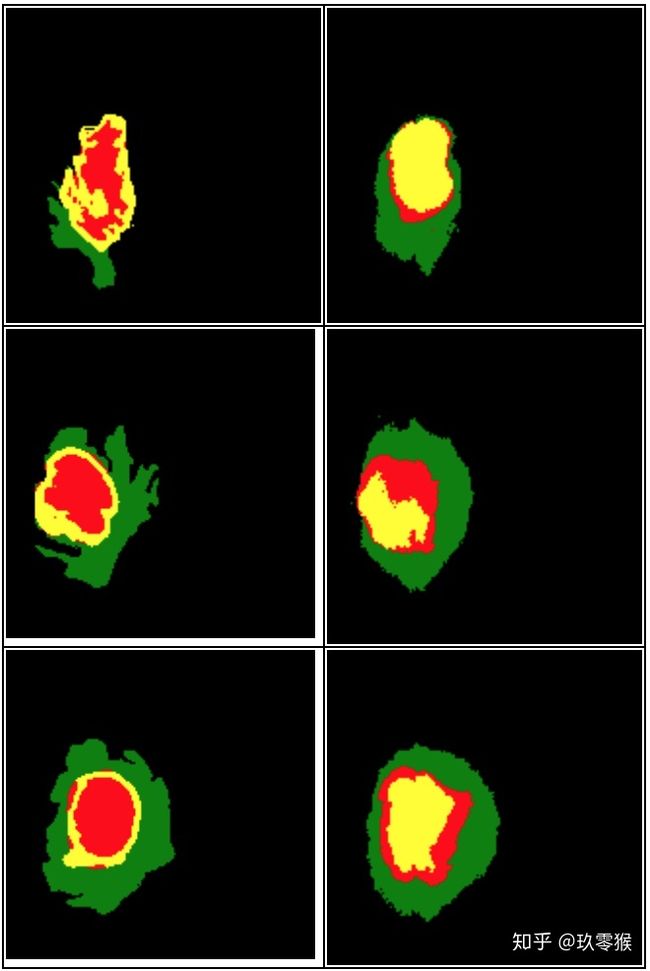
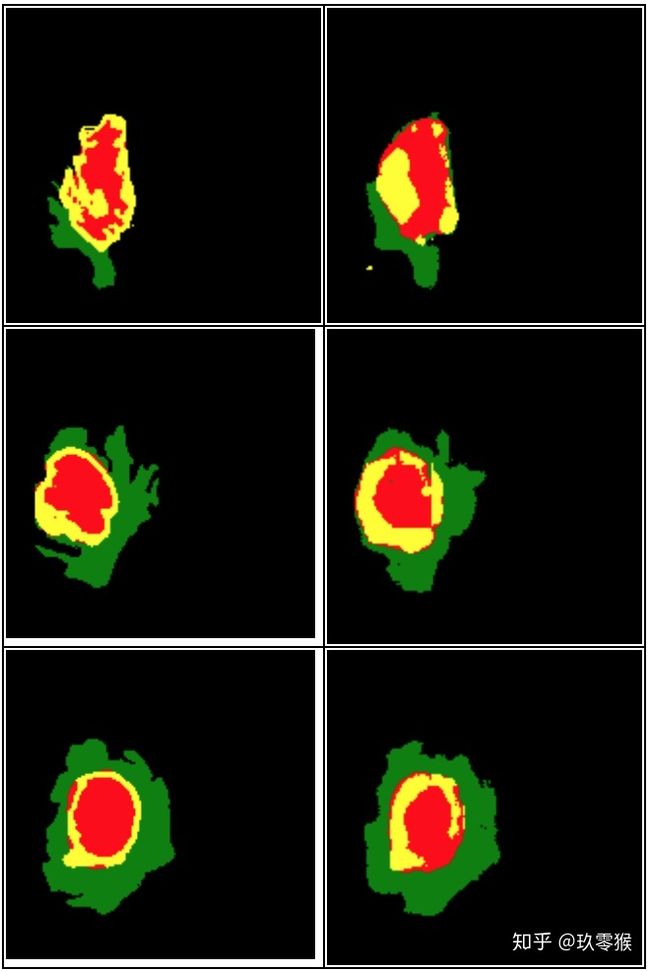
运行的结果: