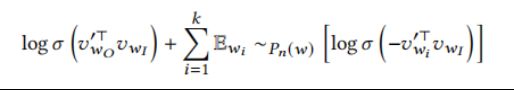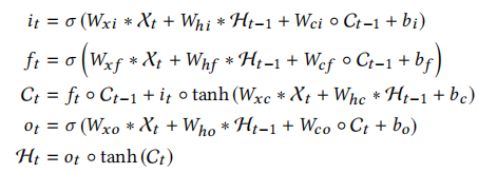基于KG嵌入和卷积-LSTM网络的药物-药物相互作用预测——方法细述
一定要看本系列第三篇:https://blog.csdn.net/zhuge2017302307/article/details/117673769
非常重要!!!!!讲重现的!!!
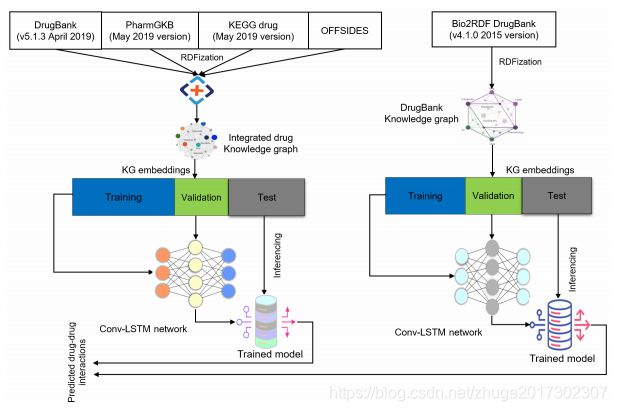
本章详细讨论所用方法,包括问题制定,数据收集和集成,KG嵌入,Conv-LSTM网络构建以及具有超参数优化的网络训练,最后一步推断DDI预测。下图是拟定方法的工作流程。
1. 问题表述
由于DDIs形成了一个复杂的网络,其中节点指药物,链路指潜在的相互作用,因此我们将DDIs预测作为一个链路预测问题处理.
给定一个有向DDIKG作为G=(V,E),其中每个边e=(u,v)表示药物u和v之间的相互作用,定义矩阵意义为:
当![]() =1时说明药物u和v之间存在相互作用,0时意味着不存在相互作用或者还未发现.接下来进行DDIs提取和KG建设.
=1时说明药物u和v之间存在相互作用,0时意味着不存在相互作用或者还未发现.接下来进行DDIs提取和KG建设.
2. DDIS开采和KG建设
根据来自DrugBank、KEGG药物和PharmGKB的药物和药物靶点相关数据构建集成知识图,同时,OFFSIDES,TWOSIDES和MEDICINE的科学文献被用来寻找具有足够证据的DDI.
2.1 数据收集
DrugBank数据库是一种生物信息学和化学信息学资源,他将详细的药物相关信息,包括化学,药理学和药物数据与全面的药物目标信息结合起来.
PharmGKB数据库包含12664个药物条目,该数据库是基因组,分子和细胞表型数据的存储库,其包含基因,疾病,药物和途径相关数据以及关于470种影响药物代谢的基因变异的详细信息.
KEGG药物数据库有10979种药物相关信息和50169种DDI关系.
OFFSIDES数据库包含从基于PharmGKB药物种的438802种药物副作用.
从这些来源,我们创建了两个数据集.1)DDI数据集,包含药物-药物相互作用对;2)知识图.
2.2 DDI提取
采用半监督方法从上述来源中提取DDIs.从DrugBank提供的XML文件中解析了DDI信息,并编译了药物标识符组合的边缘列表,给出了2641889个成对的DDI和2630796个唯一的DDI,涵盖12112种药物.KEGG药物数据库映射完毕后得到58205种相互作用.
根据使用临床副作用的标签传播预测,报道了145068个DDIs,将这些添加到数据集中.此外,还纳入了来自DDI语料库中227分MEDLINE摘要的相互作用,这提供了327个DDI,其基础是1826种药理物质.整个DDI数据集由这些来源的信息组合而造成.
2.3 KG构建和整合
为了构建知识图,使用来自DrugBank、KEGG和PharmGKB的数据。虽然PharmGKB不包含DDI信息,但它发布已知药物名称和同义词的词汇以及基因和疾病术语和遗传途径数据.
以前,Bio2RDF创建了一个大型RDF图,将包含药物,蛋白质,途径和疾病等生物试题的主要数据库中的数据相互关联。我们通过搜集各个药物数据库的原始数据,使用Bio2RDF脚本的修改版本将他们转换为RDF。然后每个RDF知识图被上传到三元组存储的图中,然后执行三元组提取。
对于我们的数据集,包含五种类型的药物相关实体,即药物,基因,蛋白质,途径和酶,表型(即疾病、副作用)也包含在内。此外,还考虑了九种生物关系:(药物,目标,蛋白质),(药物,目标,基因),(药物,酶,蛋白质),(药物,酶,基因),(药物,载体,蛋白质),(药物,载体,基因),(药物,存在,路径),(基因,存在,路径)和(路径,关联,表型)。
在这之前,基于owl:sameAs和owl:eauivalentProperty公理创造映射,其中各自的药物标识符被映射到DrugBankID。虽然基因包含了蛋白质功能分子的信息,但我们认为基因和蛋白质之间的关系是不同的,因此PharmGKB包含了关于基因和蛋白质的信息,提取信息得到三元组。
我们药物KG中的形式(主语,谓语,宾语),表明主语与对象之间有指定的关系。由于这个综合知识图不应包含任何关于药物-药物相互作用的明确信息,因此没有像DrugBank和KEGG知识图里的里的DDI-interactor-in和Interaction(相互关系)之类的词汇。下表给出了单个KG和集成KG的三元组,实体和关系类型的数量,
| 三元组 | 实体个数 | 关系类型 | |
| DrugBank | 7740864 | 2116569 | 72 |
| KEGG | 308690 | 107916 | 41 |
| PharmGKB | 2793078 | 1583910 | 135 |
接下来说明我们准备如何将这些数据作为分类器的输入。
3. 知识图嵌入
我们希望知识图中的信息来预测每对药物之间的相互作用,然而,ML分类器通常期望他们的输入作为固定长度的向量,因此,执行KG嵌入过程,将图中的信息编码成密集向量。其中KG嵌入包括三个步骤:表示实体和关系,定义评分函数,学习实体和关系表示。我们使用RDF2Vec、SimpleIE、TransE、KGloVe、CrossE和PBG进行KG嵌入。这些表示形式表示了节点的邻域和相邻节点的关系类型。由于这些方法不将文字信息合并到嵌入中,所以文字从KG中删除。
RDF2Vec通过从图中的每个实体开始执行均匀的随即游走来生成文本语料库。然后,利用技巧图SGword2vec模型使用边缘标记随机步数的语料库C作为学习每个节点的嵌入的输入。从一个给定的药物事实序列(w1,w2,…,wn)![]() ,SG模型的目的是根据固定窗口内的上下文,最大化平均对数概率Lp(见下式),其中c表示上下文:
,SG模型的目的是根据固定窗口内的上下文,最大化平均对数概率Lp(见下式),其中c表示上下文:
为了定义![]() ,我们使用负采样,将
,我们使用负采样,将![]() 替换为一个函数来区分目标词
替换为一个函数来区分目标词![]() 和噪声分布
和噪声分布![]() ,从
,从![]() 中提取k个单词:
中提取k个单词:
在语料库中出现的概念s的嵌入是上式的向量![]() 通过最大化方程得出。
通过最大化方程得出。
除了RDF2Vec之外,我们还训练了TransE嵌入作为基于翻译的KG嵌入方法的代表。在这里,每个实体和关系都被嵌入为一个低维向量,其中关系表示为从头部实体到尾部实体的平移。
还采用了CrossE嵌入方法,它显式的模拟了交叉相互作用。虽然每个实体和关系的一般嵌入和多个三元组的特定嵌入都可以使用CrossE生成,但我们只使用一般嵌入。
还采用了SimplE嵌入方法,该方法允许独立学习每个实体的两个嵌入。通过SimplE学习到的i嵌入是可解释的,这有利于将药物相关的背景知识纳入嵌入中。
学习KG中的关系表示依赖于将正实例与负实例进行对比,然后,KG通常只包含正关系实例。解决这一问题的办法是使用隐含的否定证据,在这些证据中,在KG中没有被观察到的情况被认为是否定的。查询一些资料后,我们通过破坏正样本生成负实例。
KGloVe使用了一种不同的技术来识别创建向量空间嵌入的全局模式。首先,通过个性化计算PageBank为每个节点创建共现矩阵,然后对于边都相反的图,重复这个过程。这两个矩阵被求和在一起并归一化。最后,该矩阵作为FloVe词嵌入算法的输入。此处我们只用了无偏的步行。
作为最后一个模型,我们使用PBG实现训练ComplEx,因为我们的集成KG包含许多三元组,从技术上讲,ComplEx只使用Hermitian点积创建嵌入。所有的使用ComplEx的嵌入方法可以说更简单,但由于复杂嵌入的组成可以处理大量的二进制关系(其中包括对称关系和反对称关系),所以复杂嵌入优于其他的几种模型。
我们训练了PBG的ComplEx嵌入模型来 i)创建嵌入更快,可伸缩的大图,并进行并行化训练;ii)观察该模型生成的嵌入是否对预测DDI有用。有了这些密集的表示,我们现在将图中的信息输入到我们的机器学习模型中。为了表示一个药物对的特征向量,我们将这对中的每个药物的嵌入向量连接到一对中,通过对每个现有边缘的新源或目的地进行采样来破坏正边缘,从而产生负样本。
4. 网络建设
训练各种基线ML模型,稍后将他们作为基线。在这里描述更复杂的神经网络结构。我们通过结合CNN和LSTM层来构造一个所谓的Conv-LSTM网络。如下图所示,虽然CNN使用卷积滤波器来捕获药物特征中的局部关系值,但LSTM网络可以从CNN提取的特征中携带整体关系。Conv-LSTM在不同的预测任务上表现出良好的性能。
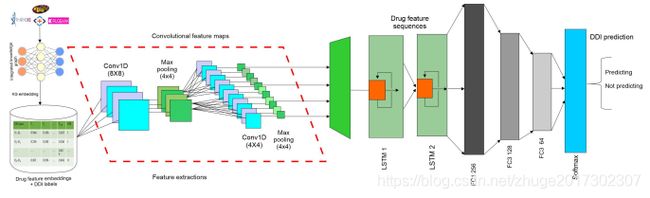 Conv-LSTM网络的示意图表示,它从将输入到n维嵌入空间并传递到CNN和LSTM层,然后得到最重要特征的向量表示,以通过dense、dropout、高斯噪声和Softmax层来预测可能的药物-药物相互作用。
Conv-LSTM网络的示意图表示,它从将输入到n维嵌入空间并传递到CNN和LSTM层,然后得到最重要特征的向量表示,以通过dense、dropout、高斯噪声和Softmax层来预测可能的药物-药物相互作用。
5. 网络培训
使用PBG,CrossE,TransE和SimpleIE的开源实现KG嵌入,并提供默认参数。另一方面,使用KGloVe的修改版本,在第400次迭代时收敛。而RDF2Vec是通过设置窗口大小为5训练的,图的的深度为5,每个实体的步长为500。每种嵌入方法都是通过改变负样本将特征向量的维数设置为300实现的。通过过滤没有计算特征向量的药物,我们能够从12664中药物中提取出12439中药物特征。然后利用RDF2Vec、TransE、PBG、KGloVe、CrossE和SimpleIE生成的嵌入来训练Conv-LSTM网络。我们的目的是估计顶点v1和v2之间存在与标记为![]() 的关系或链接的概率,给出他们的向量表示,
的关系或链接的概率,给出他们的向量表示,![]() 和
和![]() :
:![]() .
.
采用基于一阶梯度的优化技术Adam、AdaGrad、RMSprop和AdaMax,他们具有不同的学习速率和不同的批处理大小,用于学习模型参数,这些参数常熟使用优化二元交叉熵损失方程:
超参数优化是基于随即搜索和交叉验证进行的。其中模型在128个批次大小上进行训练,其中每5个都将数据的70%用于训练,30%用于评估网络,10%来自训练集的数据被随即用于验证。
6. 试验
评估代码用python编写。我们还将LR、KNN、NB、SVM、RF和GBT训练为ML基线模型。类似于Conv-LSTM网络,通过随机搜索和5倍交叉验证测试对这些分类器进行超参数优化。在实验中,80%的数据用于使用5倍交叉验证的训练,并对20%的数据进行优化模型评估,其中通过随即搜索产生最佳超参数。我们使用京都召回曲线(AUPR)下的面积和Matthias相关系数(MCC)以及AUC和F1分数来测量分类器的性能。最后,使用最好的三个模型的平均集成(MAE)来报告最终预测。