论文翻译——Skin Lesion Synthesis with Generative Adversarial Networks
Abstract
皮肤癌是目前最常见的癌症类型。早期发现是显著增加成功治疗机会的关键。目前,深度神经网络是皮肤癌自动分类的最新成果。为了进一步推动结果,我们需要解决缺乏注释数据的问题,这是昂贵的,需要专家付出很多努力。为了绕过这个问题,我们提出使用生成对抗网络生成真实的合成皮肤损伤图像。据我们所知,我们的结果是第一个展示了视觉上吸引人的合成图像,其中包含有临床意义的信息。
1 Introduction
黑素瘤是最危险的皮肤癌。它导致的死亡人数最多,约占美国所有皮肤癌的1%。治疗黑色素瘤的关键是及早发现。确诊患者的5年生存率从晚期的15%上升到早期的97%以上1。
由于皮肤病变外观的细微变化,使用图像对皮肤病变进行自动分类是一项具有挑战性的任务。自采用深度神经网络(DNNs)以来,皮肤癌分类的技术水平迅速提高2345。为了进一步推进,我们需要解决缺乏注释数据的问题,这是昂贵的,需要专家付出很多努力。为了绕过这个问题,我们提出使用生成对抗网络(GANs)6来生成真实的合成皮肤损伤图像。
GAN的目的是通过强迫合成样本与真实图像不可区分来模拟真实图像的分布。在这些生成模型的基础上,提出了许多基于GAN生成合成图像的方法789。GAN算法的一个缺点是合成图像的分辨率10。绝大多数作品是在低分辨率的数据集上进行评估的,如CIFAR (32×32)和MNIST (28×28)。然而,对于皮肤癌的分类,图像必须具有更高的细节水平(高分辨率),以便能够显示区分良性和恶性皮肤病变的恶性标记。
很少有作品显示出高分辨率图像生成的良好结果。例如,Karras等人的11渐进式训练程序生成名人面孔,最高可达1024×1024像素。他们首先向网络输入低分辨率的样本。逐渐地,网络接收到越来越高的分辨率的训练样本,同时放大各层对输出的影响。同样,Wang等人10从语义和实例图生成高分辨率图像。他们建议在不同分辨率下使用操作多个鉴别器和生成器来评估合成样本的细粒度细节和全局一致性。我们用这两个网络都进行了皮肤病变合成,并比较了所取得的结果。
在这项工作中,我们提出了一种基于GAN的方法来生成高清、有视觉吸引力和临床意义的合成皮肤病变图像。据我们所知,这项工作是第一次成功地生成了真实的皮肤病变图像(为说明,见图1)。为了评估合成图像的相关性,我们用合成图像和真实图像训练了一个皮肤癌分类网络,达到了1个百分点的改进。完整的实现可在github上获得https://github.com/alceubissoto/gan-skin-lesion.

图1:我们的方法成功地生成了高清、视觉上吸引人、具有临床意义的人造皮肤病变图像。所有的样品都是合成的。详情见第二章。
2 Proposed Approach
我们的目标是生成具有细粒度细节的皮肤病变的高分辨率合成图像。为了明确地告诉网络恶性标记,同时结合病变边界的特异性,我们将这些信息直接输入到网络。我们不是从噪声(通常的GANs过程)生成图像,而是从语义标签映射(该图的每个像素值代表对象类)和实例映射(像素结合其对象类和实例信息的图像)合成图像。因此,我们的图像合成问题指定为图像到图像的翻译问题。
2.1 GAN Architecture:The pix2pixHD Baseline
我们使用Wang等人的10pix2pixHD GAN,该GAN通过使用粗到细的生成器、多尺度判别器体系结构和鲁棒的对抗学习目标函数来改进pix2pix网络7 (一种条件图像到图像翻译GAN)。改进后的网络可以处理高分辨率的样本。
为了生成1024×512分辨率的图像,我们只利用了pix2pixHD的Global生成器。生成器输出的分辨率与我们数据集数据中最小的公共数据相适应。它由一组卷积层、一组残差层12和一组反卷积层组成。
为了处理全局和更精细的细节,我们使用Wang等人10的三个判别器。这三个判别器都以不同的分辨率接收相同的输入。这样,对于第二和第三个判别器,合成图像和真实图像分别下采样2次和4次。图2总结了GAN网络的体系结构。

图2:GAN体系结构总结。在左下角,我们展示了流程。我们详细介绍了鉴别器和生成器,以及组成它们的块。我们给出了每个卷积层的参数:k取值为kernel size;n为channel数量;s围为步长stride。下采样和上采样块后面的数字是通道的数量。
损失函数中加入了特征匹配损失9来稳定训练。它比较了所有判别器不同层的真实图像和合成图像的特征。生成器学习创建与真实图像在多个尺度上的统计数据相匹配的样本。这样,损失函数就是条件GAN损失和特征匹配损失的组合。
2.2 Modeling Skin Lesion Knowledge
建立有意义的皮肤病变知识模型是合成高质量、高分辨率皮肤病变图像的关键条件。接下来,我们将展示如何将皮肤病变场景建模为图像到图像转换的语义和实例映射。
语义映射13是一个图像,其中每个像素都有其对象类的值,通常被视为像素分割任务的结果。
为了构建我们的语义图,我们建议使用显示五种恶性肿瘤标记和相同病变的分割mask。没有病变的皮肤,没有标记的病变,每种恶性标记物都有不同的标记。为了保持病变的宽高比,同时保持输入的大小不变,与Wang等人10的原始实现相同,我们给不构成皮肤图像的边界分配另一个标签。
实例映射13是一个图像,其中像素组合了来自它的对象类和它的实例的信息。同一类的每个实例都接收到不同的像素值。当处理汽车、人和树木时,这个信息是直接的,但对于皮肤损伤内的结构,它是主观的。
为了构造实例映射,我们利用了超像素14。Superpixels 将相似像素分为一组,创建有视觉意义的实例。它们用于恶性肿瘤标记掩膜的注释过程。首先,将SLIC算法14应用于病变图像,生成超像素。然后,专家在每个超像素上标注是否存在五种恶性肿瘤标记物。因此,超像素是区分每个类中的个体的完美候选,因为它们已经作为类的最小单元在注释过程中了。在图3中,我们展示了一个损伤的语义图,它的超像素代表了它的实例图。
接下来,我们进行实验来分析我们的合成图像,并比较不同的方法来生成它们。

图3:病变的语义图,它的超像素代表它的实例图。注意超像素是如何在毛发旁边改变形状,并捕捉病变边界和内部的信息的。
3 Experiments
在本节中,我们评估基于GAN的合成皮肤病变图像生成方法:1)DCGAN 8, 2) PGAN11的条件版本,3)仅使用语义映射的pix2pixHD10版本,4)使用语义映射和实例映射。由于DCGAN的传统结构,我们选择DCGAN来代表低分辨率GAN。其他低分辨率GAN的结果没有显示出很大的改善。
3.1 Datasets
为了训练和测试pix2pixHD,我们需要特定的mask来显示是否存在有临床意义的皮肤损伤模式(包括色素网络、阴性网络、条纹、粟粒样囊肿和小球)。这些mask可从2018 ISIC挑战赛任务2(2594张图像)的训练数据集获得。2018年ISIC挑战赛任务1中获得了用于构成语义图和实例图的相同病变分割mask。我们将数据分为train(2346张图像)和test(248张图像)。测试集用于使用掩码生成网络从未见过的图像。
为了训练DCGAN和我们的PGAN版本,我们使用以下数据集:ISIC 2017挑战赛(2000张皮肤镜图像15)、ISIC存档(13000张皮肤镜图像)、Dermofit图像库16 (1300张图像)和PH2数据集17 (200张皮肤镜图像)。
为了训练分类网络,我们只使用“训练”集(2346张图片)。为了进行测试,我们使用了包含900张皮肤镜图像(270张黑素瘤)的交互式皮肤镜图集18。
3.2 Experimental Setup
对于pix2pixHD, DCGAN(官方PyTorch实现)和PGAN(除了下面列出的修改),我们保留每个实现的默认参数。
除了最后一层,我们在判别器和生成器上连接每一层的标签(良性或黑色素瘤)来修改PGAN,除了鉴别器和生成器上的最后一层。对于训练,我们从4×4分辨率开始,总是在60个epoch后淡入到下一个分辨率,其中30个epoch用于稳定。为了生成分辨率为256×256的图像,我们训练了330个epoch。我们用的都是Theano的原始版本。
对于皮肤病变分类,我们使用了在ISIC 2017挑战赛中排名第一的黑色素瘤分类19网络(inception-v420)。正如Menegola等人19所做的,我们应用随机的垂直和水平翻转、随机旋转和颜色变化作为数据增强。我们还使用50个副本进行测试增强,但跳过元学习支持向量机。
3.3 Quanlitative Evaluation
在图4中,我们直观地比较了基于GAN的方法生成的样本。
DCGAN(图4a)是最常用的GAN体系结构之一。我们表明,由DCGAN生成的样本与在我们的模型中观察到的质量相差甚远。它缺乏细粒度的细节,不适合生成高分辨率的样本。
尽管PGAN的视觉结果(图4b)比我们所知的任何其他工作都要好,但它缺乏内聚性,没有恰当的标准定位恶性肿瘤标志物。我们不能将PGAN结果与真实图像进行像素级比较。该合成图像是由噪声产生的,除了是GAN的训练集的一部分之外,与采样的真实图像没有联系。但是,我们可以比较其清晰度,恶性肿瘤标记物的存在和它们的细微细节。
当我们向网络输入语义标签映射(图4c),告知如何排列恶性肿瘤标记时,结果显著改善。当结合语义图和实例图(图4d)时,我们简化了学习过程,获得了整体最佳的视觉效果。该网络了解皮肤和病变本身的模式。

图4:基于GAN的不同方法的结果:(a) DCGAN 8, (b)我们的PGAN版本,©只使用语义映射的pix2pixHD版本,(d)使用语义映射和实例映射的pix2pixHD版本,(e)真实图像。在第一行,我们呈现完整的图像,而在第二行,我们放大以聚焦于细节。
3.4 Quantitative Evaluation
为了评价完整的合成图像集,我们使用真实训练集和合成训练集训练皮肤分类网络,并比较仅使用真实图像测试时的ROC曲线的面积(AUC)。我们使用三种不同的合成图像进行比较:Instance为使用我们的pix2pixHD10版本同时使用语义和实例映射生成的样本;Semantic是仅使用语义标签映射生成的样本;PGAN是使用我们的条件版本的PGAN11生成的样本。为了统计显著性,我们对每个实验进行了10次。
对于每个单独的集合,我们使用2346张图像,这是我们针对pix2pixHD的训练集(包含语义和实例映射)的大小。对于PGAN,我们能够生成的样本数量没有限制,但我们保持了相同的数量,保持了良性和恶性病变之间的比例。我们的结果见表1。为了验证统计显著性(将“Real + Instance + PGAN”与其他结果进行比较),我们使用配对样本的p-value进行t-test。置信为95%时,所有差异均显著(p-value<0.05)。
使用实例映射生成的合成样本是合成样本中最好的。AUC紧随感知到的视觉质量。
合成图像的结果证实它们包含了表征病变是恶性或良性的特征。结果还表明,合成图像中包含了超出真实图像边界的特征,使得分类网络平均提高了1.3个百分点,且保持了网络的稳定性。
为了研究实例图像对“Real + instance + PGAN”实现的AUC的影响,我们用新的PGAN替换实例图像样本(("Real + 2×PGAN”)。尽管两个训练集具有相同的大小,结果并没有显示出比其更小的版本“Real + PGAN”的改进。因此,相对于AUC实现的改进表明,它与“实例”图像携带的变化有关,而(不仅仅)与训练数据集的大小有关。
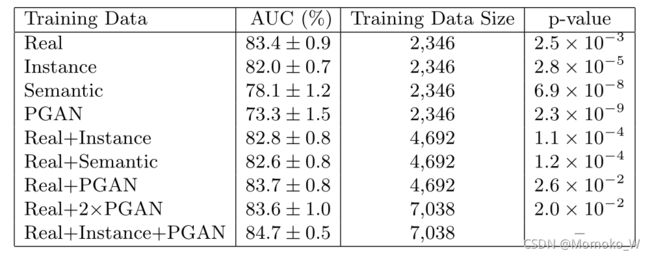
表1:皮肤癌分类网络的真实训练集与合成训练集性能比较。我们用每组训练网络10次。在合成图像中呈现的特征不仅具有视觉吸引力,而且包含了有意义的信息,有助于正确地分类皮肤病变。
4 Conclusion
在这项工作中,我们提出了基于GAN的方法来生成真实的合成皮肤病变图像。我们直观地比较结果,显示包含细粒度细节的高分辨率样本(直到1024×512)。恶性肿瘤标记物的位置连贯,清晰度高,图像美观。我们使用一个分类网络来评估一个恶性或良性病变的特征。结果表明,合成的图像携带这种信息,适合于分类目的。
然而,我们基于pix2pixHD的解决方案需要带注释的数据来生成图像。为了克服这一限制,我们正在研究使用pix2pixHD的不同方法来生成不同的图像,而不需要额外的数据:结合不同损伤的语义和实例mask,扭曲现有的真实mask以创建新的mask,甚至使用GAN来更容易地生成mask。尽管使用了这种方法,利用合成图像进行分类还是很有前景的。
Reference
American Cancer Society: Survival rates for melanoma skin cancer, by stage (2016), www.cancer.org/cancer/melanoma-skin-cancer/ detection-diagnosis-staging ↩︎
Esteva, A., Kuprel, B., Novoa, R., Ko, J., Swetter, S., Blau, H., Thrun, S.: Dermatologist-level classification of skin cancer with deep neural networks. Nature 542(7639), 115{118 (2017) ↩︎
Fornaciali, M., Carvalho, M., Bittencourt, F.V., Avila, S., Valle, E.: Towards automated melanoma screening: Proper computer vision & reliable results. arXiv:1604.04024 (2016) ↩︎
Perez, F., Vasconcelos, C., Avila, S., Valle, E.: Data augmentation for skin lesion analysis. In: ISIC Skin Image Analysis Workshop (2018) ↩︎
Valle, E., Fornaciali, M., Menegola, A., Tavares, J., Bittencourt, F.V., Li, L.T., Avila, S.: Data, depth, and design: Learning reliable models for melanoma screening. arXiv:1711.00441 (2018) ↩︎
Goodfellow, I., Pouget-Abadie, J., Mirza, M., Xu, B., Warde-Farley, D., Ozair, S., Courville, A., Bengio, Y.: Generative adversarial nets. In: NIPS (2014) ↩︎
Isola, P., Zhu, J.Y., Zhou, T., Efros, A.A.: Image-to-image translation with conditional adversarial networks. In: IEEE CVPR (2017) ↩︎ ↩︎
Radford, A., Metz, L., Chintala, S.: Unsupervised representation learning with deep convolutional generative adversarial networks. In: ICLR (2016) ↩︎ ↩︎ ↩︎
Salimans, T., Goodfellow, I., Zaremba, W., Cheung, V., Radford, A., Chen, X.: Improved techniques for training GANs. In: NIPS (2016) ↩︎ ↩︎
Wang, T.C., Liu, M.Y., Zhu, J.Y., Tao, A., Kautz, J., Catanzaro, B.: Highresolution image synthesis and semantic manipulation with conditional GANs. In: IEEE CVPR (2018) ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎ ↩︎
Karras, T., Aila, T., Laine, S., Lehtinen, J.: Progressive growing of GANs for improved quality, stability, and variation. In: ICLR (2018) ↩︎ ↩︎ ↩︎
He, K., Zhang, X., Ren, S., Sun, J.: Deep residual learning for image recognition. In: IEEE CVPR (2016) ↩︎
Lin, T.Y., Maire, M., Belongie, S., Hays, J., Perona, P., Ramanan, D., Doll´ar, P., Zitnick, C.L.: Microsoft COCO: Common objects in context. In: ECCV (2014) ↩︎ ↩︎
Achanta, R., Shaji, A., Smith, K., Lucchi, A., Fua, P., S¨usstrunk, S.: SLIC superpixels. Tech. Rep. No. EPFL-REPORT-149300 (2010) ↩︎ ↩︎
Codella, N.C., Gutman, D., Celebi, M.E., Helba, B., Marchetti, M.A., et al.: Skin Lesion Analysis Toward Melanoma Detection: A Challenge at the 2017 International Symposium on Biomedical Imaging (ISBI), Hosted by the International Skin Imaging Collaboration (ISIC). arXiv:1710.05006 (2017) ↩︎
Ballerini, L., Fisher, R., Aldridge, B., Rees, J.: A color and texture based hierarchical K-NN approach to the classification of non-melanoma skin lesions. In: Color Medical Image Analysis (2013) ↩︎
Mendon¸ca, T., Ferreira, P., Marques, J., Marcal, A., Rozeira, J.: PH2: A dermoscopic image database for research and benchmarking. In: IEEE EMBS (2013) ↩︎
Argenziano, G., Soyer, H.P., De Giorgi, V., Piccolo, D., Carli, P., Delfino, M., et al.: Dermoscopy: a tutorial. EDRA, Medical Publishing & New Media p. 16 (2002) ↩︎
Menegola, A., Tavares, J., Fornaciali, M., Li, L.T., Avila, S., Valle, E.: RECOD Titans at ISIC Challenge 2017. arXiv:1703.04819 (2017) ↩︎ ↩︎
Szegedy, C., Ioffe, S., Vanhoucke, V., Alemi, A.A.: Inception-v4, inception-resnet and the impact of residual connections on learning. In: AAAI (2017) ↩︎