K-means聚类算法
K-means聚类算法
聚类
在无监督学习中, 训练样本的标记信息是未知的, 目标是通过对无标记训练样本的学习来揭示数据的内在性质及规律, 为进一步的数据分析提供基础,此类学习任务中研究最多、应用最广的是“聚类”(clustering)
聚类试图将数据集中的样本划分为若干个通常是不相交的子集, 每个子集称为一个“簇”(cluster)
通过这样的划分, 每个簇可能对应于一些潜在的概念(类别), 需说明的是, 这些概念对聚类算法而言事先是未知的, 聚类过程仅能自动形成簇结构, 簇所对应的概念语义需由使用者来把握
关于簇的完整定义尚未达成共识,传统的定义如下
-
同一簇中的实例必须尽可能相似
-
不同簇中的实例必须尽可能不同
-
相似度和相异度的度量必须清楚并具有实际意义
性能度量
聚类性能度量大致有两类
-
将聚类结果与某个参考模型进行比较, 称为“外部指标” (external index)
-
直接考察聚类结果而不利用任何参考模型, 称为“内部指标” (internalindex)
外部指标
对数据集 D = { x 1 , x 2 , … , x m } , D=\left\{\boldsymbol{x}_{1}, \boldsymbol{x}_{2}, \ldots, \boldsymbol{x}_{m}\right\}, D={x1,x2,…,xm}, 假定通过聚类给出的族划分为 C = { C 1 , C 2 , … , C k } , \mathcal{C}=\left\{C_{1},C_{2}, \ldots, C_{k}\right\}, C={C1,C2,…,Ck}, 参考模型给出的族划分为 C ∗ = { C 1 ∗ , C 2 ∗ , … , C s ∗ } . \mathcal{C}^{*}=\left\{C_{1}^{*}, C_{2}^{*}, \ldots, C_{s}^{*}\right\} . C∗={C1∗,C2∗,…,Cs∗}. 相应地, 令 λ \boldsymbol{\lambda} λ 与 λ ∗ \lambda^{*} λ∗ 分别表示与 C \mathcal{C} C 和 C ∗ \mathcal{C}^{*} C∗ 对应的族标记向量. 我们将样本两两配对考虑, 定义
a = ∣ S S ∣ , S S = { ( x i , x j ) ∣ λ i = λ j , λ i ∗ = λ j ∗ , i < j ) } b = ∣ S D ∣ , S D = { ( x i , x j ) ∣ λ i = λ j , λ i ∗ ≠ λ j ∗ , i < j ) } c = ∣ D S ∣ , D S = { ( x i , x j ) ∣ λ i ≠ λ j , λ i ∗ = λ j ∗ , i < j ) } d = ∣ D D ∣ , D D = { ( x i , x j ) ∣ λ i ≠ λ j , λ i ∗ ≠ λ j ∗ , i < j ) } \begin{aligned} a &\left.=|S S|, \quad S S=\left\{\left(\boldsymbol{x}_{i}, \boldsymbol{x}_{j}\right) \mid \lambda_{i}=\lambda_{j}, \lambda_{i}^{*}=\lambda_{j}^{*}, i
其中集合 S S S S SS 包含了在 C \mathcal{C} C 中隶属于相同族且在 C ∗ \mathcal{C}^{*} C∗ 中也隶属于相同族的样本
集合 S D S D SD 包含了在 C \mathcal{C} C 中隶属于相同族但在 C ∗ \mathcal{C}^{*} C∗ 中隶属于不同族的样本
集合 D S D S DS 包含了在 C \mathcal{C} C 中隶属于 不同族但在 C ∗ \mathcal{C}^{*} C∗ 中隶属于相同族的样本
集合 D D DD DD 包含了在 C \mathcal{C} C 中隶属于不同族在 C ∗ \mathcal{C}^{*} C∗ 中也隶属于不同族的样本
可计算出下面这些聚类性能度量外部指标
- Jaccard系数
J C = a a + b + c \mathrm{JC}=\frac{a}{a+b+c} JC=a+b+ca
-
FM指数(Fowlkes and Mallows Index,FMI)
F M I = a a + b ⋅ a a + c \mathrm{FMI}=\sqrt{\frac{a}{a+b} \cdot \frac{a}{a+c}} FMI=a+ba⋅a+ca -
Rand指数(Rand Index,RI)
R I = 2 ( a + d ) m ( m − 1 ) \mathrm{RI}=\frac{2(a+d)}{m(m-1)} RI=m(m−1)2(a+d)
显然上述性能度量的结果值均在[0,1]区间,值越大越好
内部指标
考虑聚类结果的族划分 C = { C 1 , C 2 , … , C k } , \mathcal{C}=\left\{C_{1}, C_{2}, \ldots, C_{k}\right\}, C={C1,C2,…,Ck}, 定义
avg ( C ) = 2 ∣ C ∣ ( ∣ C ∣ − 1 ) ∑ 1 ⩽ i < j ⩽ ∣ C ∣ dist ( x i , x j ) diam ( C ) = max 1 ⩽ i < j ⩽ ∣ C ∣ dist ( x i , x j ) d min ( C i , C j ) = min x i ∈ C i , x j ∈ C j dist ( x i , x j ) d cen ( C i , C j ) = dist ( μ i , μ j ) \begin{aligned} \operatorname{avg}(C) &=\frac{2}{|C|(|C|-1)} \sum_{1 \leqslant i
其中,dist ( ⋅ , ⋅ ) (\cdot, \cdot) (⋅,⋅) 用于计算两个样本之间的距离
μ \boldsymbol{\mu} μ 代表族 C C C 的中心点 μ = \boldsymbol{\mu}= μ= 1 ∣ C ∣ ∑ 1 ⩽ i ⩽ ∣ C ∣ x i \frac{1}{|C|} \sum_{1 \leqslant i \leqslant|C|} \boldsymbol{x}_{i} ∣C∣1∑1⩽i⩽∣C∣xi
显然, avg ( C ) \operatorname{avg}(C) avg(C) 对应于族 C C C 内样本间的平均距离 , diam ( C ) , \operatorname{diam}(C) ,diam(C) 对应于族 C C C 内样本间的最远距离 , d min ( C i , C j ) , d_{\min }\left(C_{i}, C_{j}\right) ,dmin(Ci,Cj) 对应于族 C i C_{i} Ci 与族 C j C_{j} Cj 最近样本间的距离 , d c e n ( C i , C j ) , d_{\mathrm{cen}}\left(C_{i}, C_{j}\right) ,dcen(Ci,Cj) 对应于族 C i C_{i} Ci 与族 C j C_{j} Cj 中心点间的距离.
可计算出下面这些聚类性能度量内部指标
- DB 指数(Davies-Bouldin Index, DBI)
D B I = 1 k ∑ i = 1 k max j ≠ i ( avg ( C i ) + avg ( C j ) d c e n ( μ i , μ j ) ) \mathrm{DBI}=\frac{1}{k} \sum_{i=1}^{k} \max _{j \neq i}\left(\frac{\operatorname{avg}\left(C_{i}\right)+\operatorname{avg}\left(C_{j}\right)}{d_{\mathrm{cen}}\left(\mu_{i}, \mu_{j}\right)}\right) DBI=k1i=1∑kj=imax(dcen(μi,μj)avg(Ci)+avg(Cj)) - Dunn 指数(Dunn Index, DI)
D I = min 1 ⩽ i ⩽ k { min j ≠ i ( d min ( C i , C j ) max 1 ⩽ l ⩽ k diam ( C l ) ) } \mathrm{DI}=\min _{1 \leqslant i \leqslant k}\left\{\min _{j \neq i}\left(\frac{d_{\min }\left(C_{i}, C_{j}\right)}{\max _{1 \leqslant l \leqslant k} \operatorname{diam}\left(C_{l}\right)}\right)\right\} DI=1⩽i⩽kmin{j=imin(max1⩽l⩽kdiam(Cl)dmin(Ci,Cj))}
显然, DBI 的值越小越好, 而 DI 则相反, 值越大越好
距离度量
可参考k-近邻算法(KNN)中距离度量部分
常见的聚类算法
原型聚类
原型聚类亦称“基于原型的聚类” (prototype-based clustering)
假设聚类结构能通过一组原型(指样本空间中具有代表性的点)刻画, 通常情形下,算法先对原型进行初始化, 然后对原型进行迭代更新求解. 采用不同的原型表示、不同的求解方式将产生不同的算法,下面是几种著名的原型聚类算法
- k-means
- 学习向量量化(Learning Vector Quantization,LVQ)
- 高斯混合聚类(Mixture-of-Guassian)
层次聚类
层次聚类(hierarchical clustering)试图在不同层次对数据集进行划分,从而形成树形的聚类结构
数据集的划分可采用“自底向上”的聚合策略,它将最相似的两个点合并,直到所有点都合并到一个群集中为止
也可采用“自顶向下"的分拆策略,它以所有点作为一个簇开始,并在每一步拆分相似度最低的簇,直到仅剩下单个数据点
在分层聚类中,聚类数(k)通常由用户预先确定,通过在指定深度切割树状图来分配聚类,从而得到k组较小的树状图
与许多分区聚类技术不同,分层聚类是确定性过程,这意味着当您对相同的输入数据运行两次算法时,聚类分配不会改变
层次聚类方法的优点包括
- 它们通常会揭示有关数据对象之间的更详细的信息。
- 它们提供了可解释的树状图。
层次聚类方法的缺点包括
- 算法复杂度高
- 对噪音和异常值很敏感
基于密度的聚类
基于密度的聚类根据区域中数据点的密度确定聚类分配,在高密度的数据点被低密度区域分隔的地方分配簇
与其他类别的聚类不同,该方法不需要用户指定群集数量,而是有一个基于距离的参数充当可调阈值来确定是否将接近的点视为簇成员
DBSCAN是一种著名的密度聚类算法,它基于一组领域参数来刻画样本分布的紧密程度
基于密度的聚类方法的优点包括
- 他们擅长识别非类圆型簇。
- 它们可以抵抗异常值。
基于密度的聚类方法的缺点包括
- 它们不太适合在高维空间中聚类
- 很难确定密度不同的簇。
K-means
k均值聚类算法(k-means clustering algorithm)是一种迭代求解的聚类分析算法
它试图将数据集划分为K个不同的非重叠子组(簇),其中每个数据点只属于一个组
同时使得簇内数据点尽可能相似,还要尽可能保持簇之间的差异
聚类分配的质量是通过计算质心收敛后的平方误差和(sum of the squared error,SSE)来确定的,或者与先前的迭代分配相符
SSE定义为每个点与其最接近的质心的欧几里德距离平方的总和,k-means的目的是尝试最小化该值
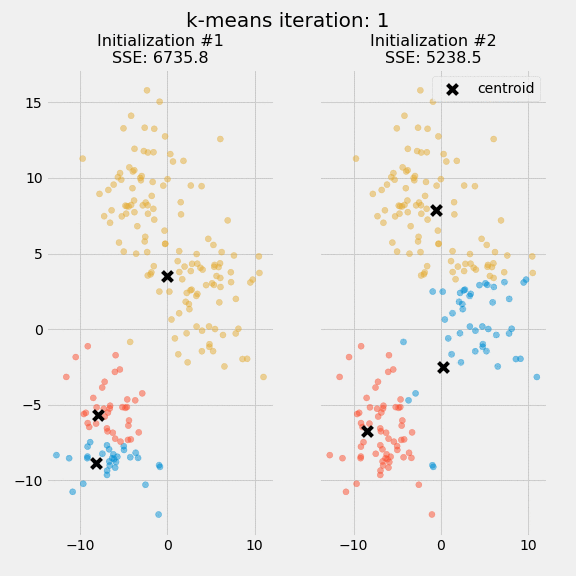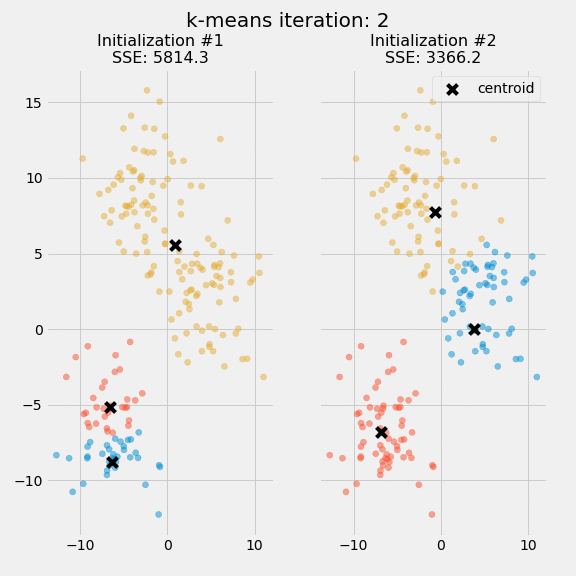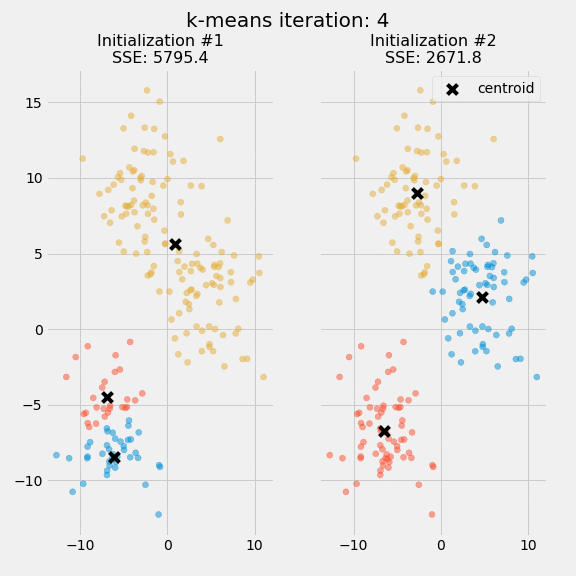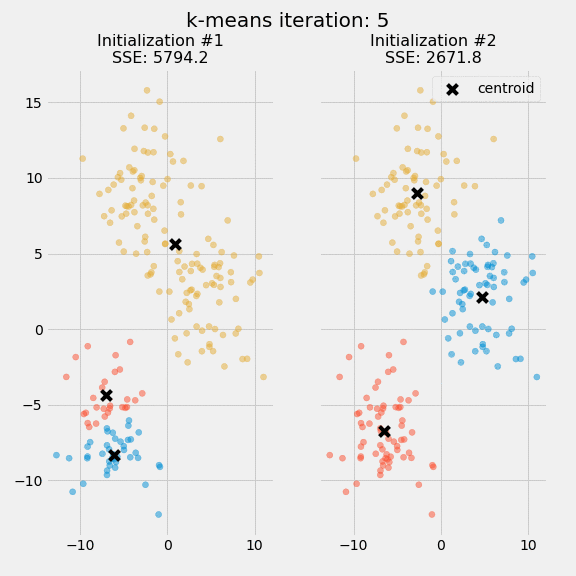
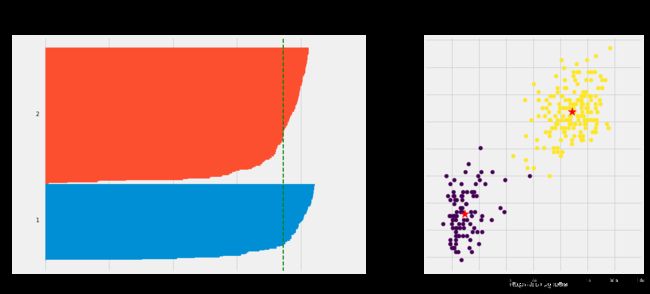
下图显示了在同一数据集上两次不同的k -means算法运行的前五个迭代中的质心和SSE更新:
此图的目的是表明质心的初始化是重要的一步,随机初始化步骤导致k -means算法不确定,这意味着如果您在同一数据集上运行两次相同的算法,则簇分配将有所不同
研究人员通常会对整个k均值算法进行几次初始化,并从具有最低SSE的初始化
工作方式
- 指定簇数K
- 首先对数据集shuffle,然后为质心随机选择K个数据点
- 不断迭代,直到质心没有变化,即数据点分配给群集的操作没有改变
- 计算数据点和所有质心之间的平方距离之和
- 将每个数据点分配给最近的群集(质心)
- 通过获取属于每个群集的所有数据点的平均值,计算群集的质心
k-means解决问题的方法称为期望最大化(Expectation Maximization,EM)
目标函数是
E = ∑ i = 1 k ∑ x ∈ C i ∥ x − μ i ∥ 2 2 E=\sum_{i=1}^{k} \sum_{\boldsymbol{x} \in C_{i}}\left\|\boldsymbol{x}-\boldsymbol{\mu}_{i}\right\|_{2}^{2} E=i=1∑kx∈Ci∑∥x−μi∥22
其中 μ i = 1 ∣ C i ∣ ∑ x ∈ C i x \mu_{i}=\frac{1}{|C_{i}|} \sum_{x \in C_{i}} x μi=∣Ci∣1∑x∈Cix 是族 C i C_{i} Ci 的均值向量
最小化目标函数并不容易, 找到它的最优解需考察样本集 D D D 所有可能的族划分, 这是一个 N P \mathrm{NP} NP 难问题
因此k-means算法采用了贪心策略, 通过迭代优化来近似求解
Tips
-
类别数 k k k 值需要预先指定,而在实际应用中最优的 k k k 值是不知道的,解决这个问题的一个方法是尝试用不同的 k k k 值聚类, 检验各自得到聚类结果的质量, 推测最优的 k k k 值
-
由于包括kmeans在内的聚类算法使用基于距离的度量来确定数据点之间的相似性,因此建议对数据进行标准化
-
由于kmeans算法可能停留在局部最优而不收敛于全局最优,因此不同的初始化可能导致不同的聚类,建议使用不同的质心初始化来运行算法,并选择产生较低SSE的运行结果
找到合适的K
下面将介绍两个指标,这些指标可能使我们更直接的观察k的取值
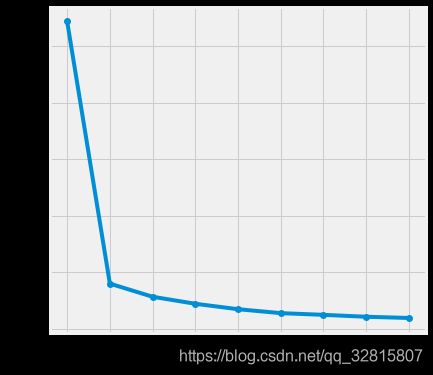
肘部法则(Elbow Method)
k-means是以最小化样本与质点平方误差作为目标函数,将每个簇的质点与簇内样本点的平方距离误差和称为畸变程度(distortions),那么,对于一个簇,它的畸变程度越低,代表簇内成员越紧密,畸变程度越高,代表簇内结构越松散
畸变程度会随着类别的增加而降低,但对于有一定区分度的数据,在达到某个临界点时畸变程度会得到极大改善,之后缓慢下降,这个临界点就可以考虑为聚类性能较好的点
存在当曲线是单调递减的时仍然很难找出合适的簇数的可能
轮廓系数(Silhouette Coefficient)
轮廓系数是聚类结合和分离程序的评价指标,它基于两个因素来量化数据点适合其分配的簇的程度
-
数据点与簇中其他点的距离有多近
-
数据点与其他簇中的点有多远
计算距同一簇中所有数据点的平均距离 a i a_i ai
计算到最近的簇中所有数据点的平均距离 b i b_i bi
b i − a i max ( a i , b i ) \frac{b^{i}-a^{i}}{\max \left(a^{i}, b^{i}\right)} max(ai,bi)bi−ai
轮廓系数值介于-1和1之间,越接近1表示样本所在簇合理
若近似为0,则说明样本在两个簇的边界上
缺点
如果簇是类圆形的,那么Kmeans算法是可以胜任的,它总是尝试在质心周围构造一个不错的球形
但这这意味着,当群集具有复杂的几何形状时,Kmeans的表现不好,如下
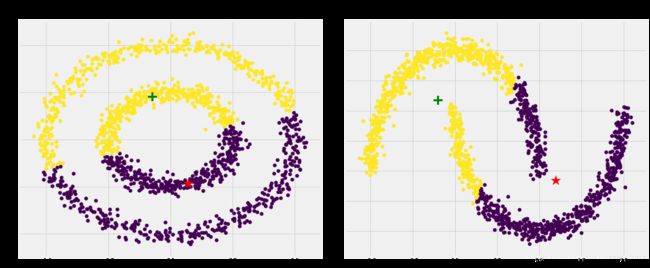
不出所料,kmeans无法找出两个数据集的正确聚类
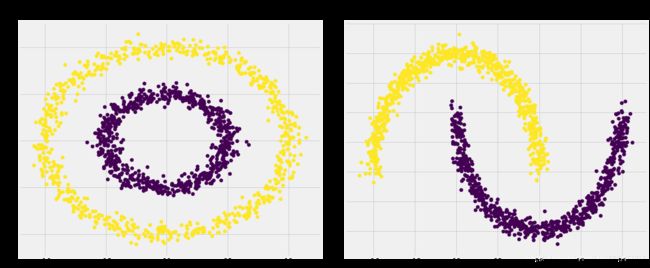
但是,如果我们使用核方法,将其转换为高维表示从而使数据线性可分离,则可以帮助kmeans完美地聚类此类数据集
实现代码
# coding = utf-8
import numpy as np
class KMeans:
def __init__(self,n_clusters, max_iters=100, random_state=666):
"""初始化Kmeans模型"""
self.n_clusters = n_clusters
self.max_iters = max_iters
self.random_state = random_state
def initializ_centroids(self, X):
np.random.RandomState(self.random_state)
random_idx = np.random.permutation(X.shape[0])
centroids = X[random_idx[:self.n_clusters]]
return centroids
def compute_centroids(self, X, labels):
centroids = np.zeros((self.n_clusters, X.shape[1]))
for k in range(self.n_clusters):
centroids[k, :] = np.mean(X[labels == k, :], axis=0)
return centroids
def compute_distance(self, X, centroids):
distance = np.zeros((X.shape[0], self.n_clusters))
for k in range(self.n_clusters):
row_norm = np.linalg.norm(X - centroids[k, :], axis=1)
distance[:, k] = np.square(row_norm)
return distance
def find_closest_cluster(self, distance):
return np.argmin(distance, axis=1)
def compute_sse(self, X, labels, centroids):
distance = np.zeros(X.shape[0])
for k in range(self.n_clusters):
distance[labels == k] = np.linalg.norm(X[labels == k] - centroids[k], axis=1)
return np.sum(np.square(distance))
def fit(self, X):
self.centroids = self.initializ_centroids(X)
for i in range(self.max_iters):
old_centroids = self.centroids
distance = self.compute_distance(X, old_centroids)
self.labels = self.find_closest_cluster(distance)
self.centroids = self.compute_centroids(X, self.labels)
if np.all(old_centroids == self.centroids):
break
self.error = self.compute_sse(X, self.labels, self.centroids)
return self
def predict(self, X_predict):
distance = self.compute_distance(X, old_centroids)
return self.find_closest_cluster(distance)
if __name__ == '__main__':
from sklearn.datasets import make_blobs
from sklearn.preprocessing import StandardScaler
features, true_labels = make_blobs(
n_samples=200,
n_features=2,
centers=3,
cluster_std=2.75,
random_state=42
)
scaler = StandardScaler()
scaled_features = scaler.fit_transform(features)
kmeans = KMeans(
n_clusters=3,
max_iters=300,
random_state=42
)
kmeans.fit(scaled_features)
参考
机器学习-周志华
K-Means Clustering in Python: A Practical Guide
K-means Clustering: Algorithm, Applications, Evaluation Methods, and Drawbacks