生信学习——生信人的20个R语言习题(下)(附详细答案解读)
题目目录
- 12. 理解统计学指标mean,median,max,min,sd,var,mad并计算出每个基因在所有样本的这些统计学指标,最后按照mad值排序,取top 50 mad值的基因,得到列表。
- 13. 根据第12步骤得到top 50 mad值的基因列表来取表达矩阵的子集,并且热图可视化子表达矩阵。试试看其它5种热图的包的不同效果。
- 14. 取不同统计学指标mean,median,max,mean,sd,var,mad的各top50基因列表,使用UpSetR包来看他们之间的overlap情况。
- 15. 在第二步的基础上面提取CLL包里面的data(sCLLex)数据对象的样本的表型数据。
- 16. 添加样本的临床表型数据信息(更改样本名),对所有样本的表达矩阵进行聚类并且绘图。
- 17. 对所有样本的表达矩阵进行PCA分析并且绘图,同样要添加表型信息。
- 18. 根据表达矩阵及样本分组信息进行批量T检验,得到检验结果表格。
- 19. 使用limma包对表达矩阵及样本分组信息进行差异分析,得到差异分析表格,重点看logFC和P值,画个火山图(就是logFC和-log10(P值)的散点图)。
- 20. 对T检验结果的P值和limma包差异分析的P值画散点图,看看哪些基因相差很大。
写在前面——17-20题设计的知识点比较多,本人还没理解透彻,所以没有做完。挖坑挖坑。
(这么多坑,啥时候能填完,/(ㄒoㄒ)/~~)
2021.12.11第一次编辑—— 红色字体
一些新的理解
1. 12-17题中取各种指标进行操作,绘图以及聚类和PCA分析都是为了判断该数据的可用性。聚类的效果越好或PCA分析中不同组分的越开,数据可用性越高。
2. 18,19题都是对数据进行差异分析。提到表达量数据分析,不管是通过芯片技术还是高通量测序技术得到的表达量矩阵,我们都需要根据样本的分组信息来对所检测到的所有基因或者蛋白分子来做差异分析,想找到显著性变化的生物大分子。它们的本质就是对表达量矩阵做一个归一化,让不同组样本的表达量具有可比性,然后利用理想的统计分布检验函数来计算差异的显著性。
3. 更新了19题的代码及注释
题目原文:http://www.bio-info-trainee.com/3409.html
参考答案:https://www.jianshu.com/p/dd4e285665e1 https://www.jianshu.com/p/788010093c90
参考答案:https://www.jianshu.com/p/c62cbb9e1a2e
上篇指路:https://blog.csdn.net/narutodzx/article/details/119775154
12. 理解统计学指标mean,median,max,min,sd,var,mad并计算出每个基因在所有样本的这些统计学指标,最后按照mad值排序,取top 50 mad值的基因,得到列表。
# 对exprSet的每行求平均值,默认为升序排列,top50即为后50位
g_mean <- tail(sort(apply(exprSet,1,mean)),50)
# 中位数
g_median <- tail(sort(apply(exprSet,1,median)),50)
# 最大值
g_max <- tail(sort(apply(exprSet,1,max)),50)
# 最小值
g_min <- tail(sort(apply(exprSet,1,min)),50)
# 标准差
g_sd <- tail(sort(apply(exprSet,1,sd)),50)
# 方差
g_var <- tail(sort(apply(exprSet,1,var)),50)
# 绝对中位差
g_mad <- tail(sort(apply(exprSet,1,mad)),50)
g_mad
str(g_mad)
names(g_mad)
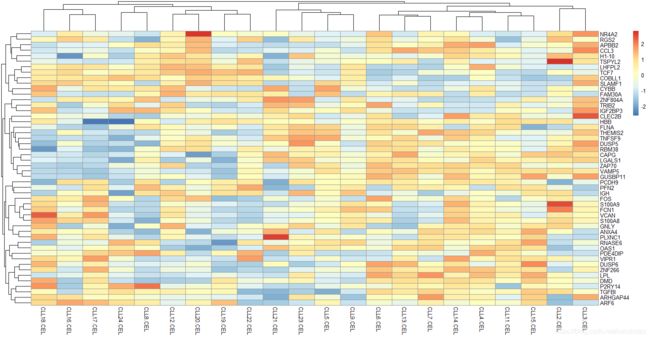
13. 根据第12步骤得到top 50 mad值的基因列表来取表达矩阵的子集,并且热图可视化子表达矩阵。试试看其它5种热图的包的不同效果。
# 紧接上题
library(pheatmap)
# 将top50mad值的基因列表取出
top50mad_exprSet <- exprSet[names(g_mad),]
# scale对数据的“列”进行归一化
# 需要将矩阵转置,scale之后,再转置回来
top50mad_exprSet <- t(scale(t(top50mad_exprSet)))
pheatmap(top50mad_exprSet)
14. 取不同统计学指标mean,median,max,mean,sd,var,mad的各top50基因列表,使用UpSetR包来看他们之间的overlap情况。
# 集合不超过5个时,一般使用韦恩图
# 五个以上可以用UpSetR包进行集合可视化
# install.packages("UpSetR")
library(UpSetR)
# 取这7个统计量的所有基因名,去除重复的
g_all <- unique(c(names(g_mean), names(g_median), names(g_max),
names(g_min), names(g_sd), names(g_var), names(g_mad)))
# 第一列为基因名,第二列是将g_mean在g_all中存在的位置映射为1,反之为0,后面类推
# 最后将所有列合并成数据框
dat <- data.frame(g_all=g_all,
g_mean=ifelse(g_all %in% names(g_mean), 1, 0),
g_median=ifelse(g_all %in% names(g_median), 1, 0),
g_max=ifelse(g_all %in% names(g_max), 1, 0),
g_min=ifelse(g_all %in% names(g_min), 1, 0),
g_sd=ifelse(g_all %in% names(g_sd), 1, 0),
g_var=ifelse(g_all %in% names(g_var), 1, 0),
g_mad=ifelse(g_all %in% names(g_mad), 1, 0)
)
upset(dat, nsets = 7)


图中左下方:展示了七个集合的名称以及每个集合含多少数据。
图中右侧的直方图和点阵:点的左侧对应集合名称,点阵上方的直方图表示交集数据。
例如:
g_mad这一个点,表示在g_mad中值为1的数量有25个,且这25个基因是g_mad独有的。
g_var和g_sd这两个点连线,表示g_var和g_sd的交集有25个,且这25个基因是g_var和g_sd共同独有的。
把这个想象成韦恩图,会好理解一些…
15. 在第二步的基础上面提取CLL包里面的data(sCLLex)数据对象的样本的表型数据。
pdata <- pData(sCLLex)
group_list <- as.character(pdata[,2])
group_list
table(group_list)
16. 添加样本的临床表型数据信息(更改样本名),对所有样本的表达矩阵进行聚类并且绘图。
# 修改表达矩阵列名,修改为group_list加数字的形式
colnames(exprSet) <- paste(group_list,1:22,sep='')
# dist()计算矩阵所有“行”之间的距离(欧几里得距离)
# 因为要对样本名聚类,所以需要先把exprSet转置
# hclust()来实现层次聚类
hc <- hclust(dist(t(exprSet)))
# 设置图片的各种样式,可以改改数字跑一下,就知道修改的是什么东西了
nodePar <- list(lab.cex = 0.6, pch = c(NA, 19), cex = 0.7, col = "blue")
# 设置plot的四个边指定的边距行数(下,左,上,右)
par(mar=c(5,5,5,10))
# 设置图片为水平,绘图
plot(as.dendrogram(hc), nodePar = nodePar, horiz = TRUE)


可以看到大部分的progres.或stable都聚集在一起,说明这个数据集是有意义的,可以进行下一阶段的分析。
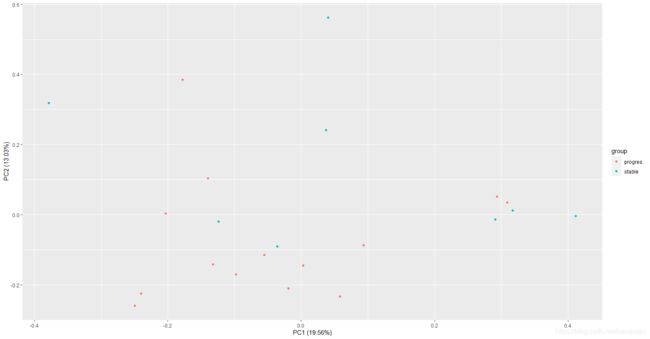
17. 对所有样本的表达矩阵进行PCA分析并且绘图,同样要添加表型信息。
本题不懂,挖个坑,学透PCA来填。
不懂为啥要重新获得表达矩阵,里面不是有好多与基因不对应的探针嘛,不对应也能当影响指标吗?
转置是因为prcomp只对列操作吗?
绘制的图片代表什么意思?图片中不同颜色的点分的越开越好,说明我们找的数据可以用
# 主成分分析,也称主分量分析,旨在利用降维的思想,把多指标转化为少数几个综合指标
# install.packages("ggfortify")
library(ggfortify)
exprSet2 <- exprs(sCLLex)
df <- as.data.frame(t(exprSet2))
df$group <- group_list
autoplot(prcomp(df[,1:(ncol(df)-1)]), data=df, colour = 'group')
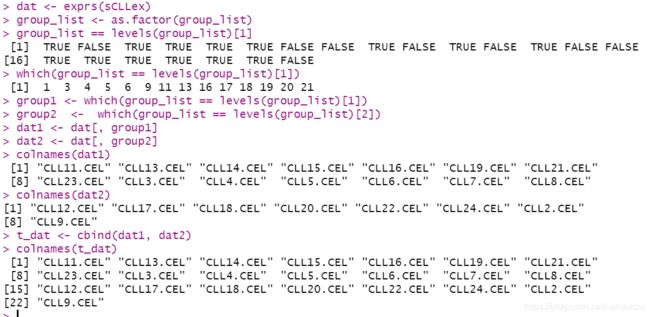
18. 根据表达矩阵及样本分组信息进行批量T检验,得到检验结果表格。
# 挖坑待埋...
# 注意这时候使用的dat等同于最初的exprSet
dat <- exprs(sCLLex)
# 表型数据因子化
group_list <- as.factor(group_list)
# 整理矩阵
group1 <- which(group_list == levels(group_list)[1])
group2 <- which(group_list == levels(group_list)[2])
dat1 <- dat[, group1]
dat2 <- dat[, group2]
t_dat <- cbind(dat1, dat2)
# T检验,取p.value
pvals <- apply(dat, 1, function(x){
t.test(as.numeric(x) ~ group_list)$p.value
})
# 调整多个比较的p值
p.adj <- p.adjust(pvals, method = "BH")
# log2FC = log2 (mean(处理组/对照组))
# dat是取log2之后的结果
# log2FC = log2(mean(处理组))-log2(mean(对照组)) = avg_2-avg_1
avg_1 <- rowMeans(dat1)
avg_2 <- rowMeans(dat2)
log2FC <- avg_2-avg_1
DEG_t.test <- cbind(avg_1, avg_2, log2FC, pvals, p.adj)
DEG_t.test<-DEG_t.test[order(DEG_t.test[,4]),]
DEG_t.test<-as.data.frame(DEG_t.test)
head(DEG_t.test)
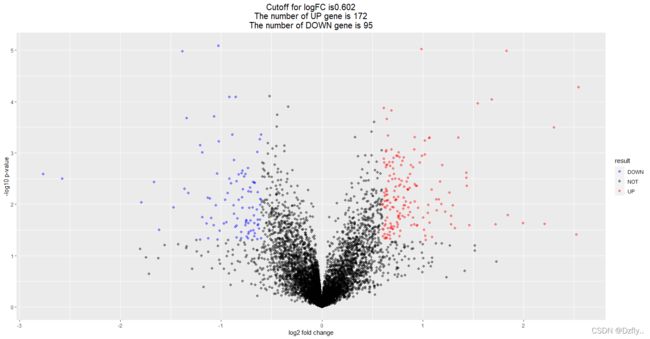
19. 使用limma包对表达矩阵及样本分组信息进行差异分析,得到差异分析表格,重点看logFC和P值,画个火山图(就是logFC和-log10(P值)的散点图)。
library(limma)
# 得到按组分离的矩阵
design <- model.matrix(~0 + factor(group_list))
colnames(design) <- levels(factor(group_list))
rownames(design) <- colnames(exprSet)
design
# 差异比较矩阵
contrast.matrix<-makeContrasts(paste0(unique(group_list),collapse = "-"),levels = design)
contrast.matrix
##step1
# 在给定一系列序列的情况下,对每个基因拟合线性模型
# exprSet要求行对应于基因,列对应于样本
# design要求行对应样本,列对应系数
fit <- lmFit(exprSet,design)
##step2
# 根据lmFit的拟合结果进行统计推断,计算给定一组对比的估计系数和标准误差
# fit由lmFit得到的
# contrasts要求:行对应拟合系数,列包含对比度
fit2 <- contrasts.fit(fit, contrast.matrix)
# Methods of assessing differential expression
fit2 <- eBayes(fit2)
##step3
# 从线性模型拟合中提取出排名靠前的基因表
# For topTable, fit should be an object of class MArrayLM as produced by lmFit and eBayes.
# topTable 默认显示前10个基因的统计数据;使用选项n可以设置,n=Inf就是不设上限,全部输出
# 只有progres.-stable一组的差异基因,就用coef = 1
tempOutput <- topTable(fit2, coef=1, n=Inf)
# 去除缺失值
nrDEG <- na.omit(tempOutput)
head(nrDEG)
## volcano plot
DEG <- nrDEG
# 设定阈值,选出UP、DOWN、NOT表达基因
# mean+2SD可以反映95%以上的观测值,设为mean+3SD,就可以反映97%以上的观测
logFC_cutoff <- with(DEG, mean(abs(logFC)) + 2*sd(abs(logFC)))
# 首先判断p值和logFC的绝对值是不是达到了设定的阈值,如果是则进行下一步判断,如果不是则返回NOT
# 然后判断logFC与阈值的大小关系,返回UP或DOWN
DEG$result <- as.factor(ifelse(DEG$P.Value < 0.05 & abs(DEG$logFC) > logFC_cutoff,
ifelse(DEG$logFC >logFC_cutoff, 'UP', 'DOWN'), 'NOT')
)
# 设置火山图标题
this_tile <- paste0('Cutoff for logFC is', round(logFC_cutoff, 3),
'\nThe number of UP gene is ', nrow(DEG[DEG$result == 'UP', ]),
'\nThe number of DOWN gene is ', nrow(DEG[DEG$result == 'DOWN', ]))
this_tile
head(DEG)
library(ggplot2)
# 对p值进行对数转换绘制的图就像火山喷发一样更美观
# 设置一系列的美化条件
ggplot(data=DEG, aes(x=logFC, y=-log10(P.Value), color=result)) +
geom_point(alpha=0.4, size=1.75) +
theme_set(theme_set(theme_bw(base_size=20)))+
xlab("log2 fold change") + ylab("-log10 p-value") +
ggtitle( this_tile ) + theme(plot.title = element_text(size=15,hjust = 0.5))+
scale_colour_manual(values = c('blue','black','red'))
# blue对应DOWN,black对应NOT,red对应UP