Pheatmap做热图数据处理过程
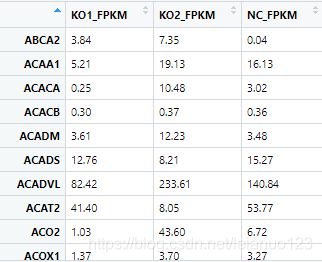
1.原始的FPKM数据a5
View(a5)
a4=a5[,c(2,3)]
View(a4)
a6=a4
a7=log2(a6+0.0005)
col = colorRampPalette(colors = c("blue","white","red"))(80)
##col = colorRampPalette(c("lightblue","lightyellow","orange","#FF6A6A"),bias=1)(300)
##300表示色阶,从高到低300个色阶,如果设置5个色阶,即可切成5块
View(a7)
colnames(a7)=c("KO","NC")
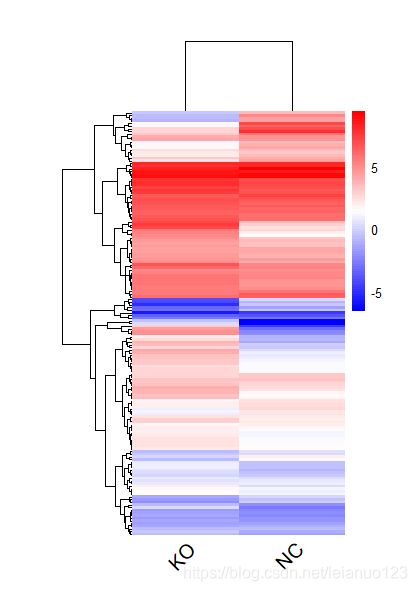
pheatmap(a7,angle_col = 45,cellheight = 2,cellwidth = 80,color=col, show_rownames = F,fontsize_col=15)
最终得到的结果图
在这里值得注意的是数据的处理过程。
实际应用中,异常值的出现会毁掉一张热图。这通常不是我们想要的。为了更好的可视化效果,需要对数据做些预处理,主要有对数转换,Z-score转换,抹去异常值,非线性颜色等方式。
Pheatmap的数据需要进行转换,常见的转换有
1.对数转换
a7=log2(a6+0.0005)
- +1是为了防止对0取对数;是加1还是加个更小的值取决于数据的分布。
加的值一般认为是检测的低阈值,低于这个值的数字之间的差异可以忽略。(主要看数据的分布范围,如果较大的数据较多,较少的数据较小,可以加的小点,拉开差距。
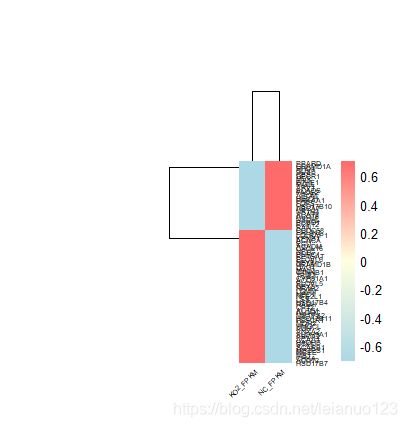
2.抹去异常值,比如下面这张图,红框里面的数据整体偏大,会影响数据的整体归一化过程,可以选择将大于30的值(看数据分布情况)赋值30
a3[a3>30] <-30 #将a3中的大于30的值赋值到30
data <-data[apply(data,1,var)!=0,] ##去掉方差为0的行,也就是值全都一致的行
#标准化数据,获得Z-score,并转换为data.frame
data_scale <-as.data.frame(t(apply(data,1,scale)))
colnames(data_scale)<- colnames(data)
4.非线性转换
正常来讲,颜色的赋予在最小值到最大值之间是均匀分布的。非线性颜色则是对数据比较小但密集的地方赋予更多颜色,数据大但分布散的地方赋予更少颜色,这样既能加大区分度,又最小的影响原始数值。通常可以根据数据模式,手动设置颜色区间。为了方便自动化处理,我一般选择用四分位数的方式设置颜色区间。
data_ori <- "Grp_1;Grp_2;Grp_3;Grp_4;Grp_5
a;6.6;20.9;100.1;600.0;5.2
b;20.8;99.8;700.0;3.7;19.2
c;100.0;800.0;6.2;21.4;98.6
d;900;3.3;20.3;101.1;10000"
data <- read.table(text=data_ori,
header=T, row.names=1, sep=";", quote="")
data$ID = rownames(data)
data_m = melt(data, id.vars=c("ID"))
# 获取数据的最大、最小、第一四分位数、中位数、第三四分位数
summary_v <- summary(data_m$value)
summary_v
Min. 1st
Qu. Median Mean 3rd
Qu. Max.
3.30
16.05 60.00 681.40 225.80 10000.00
# 在最小值和第一四分位数之间划出6个区间,第一四分位数和中位数之间划出6个区间,中位数和第三四分位数之间划出5个区间,最后的数划出5个区间
break_v <-unique(c(seq(summary_v[1]*0.95,summary_v[2],length=6),seq(summary_v[2],summary_v[3],length=6),seq(summary_v[3],summary_v[5],length=5),seq(summary_v[5],summary_v[6]*1.05,length=5)))
break_v
[1]
3.135 5.718
8.301 10.884 13.467
16.050 24.840
[8]
33.630 42.420 51.210
60.000 101.450 142.900 184.350
[15] 225.800
2794.350 5362.900 7931.450 10500.000
# 安照设定的区间分割数据
# 原始数据替换为了其所在的区间的数值
data_m$value <-cut(data_m$value,breaks=break_v,labels=break_v[2:length(break_v)])
break_v=unique(data_m$value)
data_m
# 虽然看上去还是数值,但已经不是数字类型了
# 而是不同的因子了,这样就可以对不同的因子赋予不同的颜色了
is.numeric(data_m$value)
[1] FALSE
is.factor(data_m$value)
[1] TRUE
break_v
#[1] 8.301 24.84
101.45 2794.35 5.718 10500
#18 Levels: 5.718 8.301 10.884 13.467 16.05
24.84 33.63 42.42 51.21 … 10500
#产生对应数目的颜色
gradientC=c('green','yellow','red')
col <-colorRampPalette(gradientC)(length(break_v))
试剂上数据转换的格式主要取决与你的数据分布,值得夺取探索,毕竟是数学,总有他背后的规律和名堂。