Cell Genomics封面|北大吴华君课题组利用空间多组学技术解析肿瘤内空间异质性...
Cell Genomics封面|吴华君课题组利用空间多组学技术解析肿瘤内空间异质性
肿瘤内异质性(intra-tumor heterogeneity,ITH)是癌症复发转移的重要驱动因素之一。随着单细胞多组学技术的广泛应用,我们对肿瘤内异质性如何影响癌症治疗的理解逐步加深。但是,肿瘤亚克隆在肿瘤内部的空间分布模式,及其如何影响免疫微环境和癌症预后仍是领域内的空白。
2022年8月10日,北京大学基础医学院精准医疗多组学研究中心/北京大学肿瘤医院吴华君课题组、美国哈佛大学Franziska Michor课题组和美国斯隆凯特琳癌症中心Robert J. Downey课题组合作在Cell新子刊Cell Genomics发表封面文章Spatial intra-tumor heterogeneity is associated with survival of lung adenocarcinoma patients,利用空间多组学技术和开发的计算方法在蛋白质组和基因组层面上揭示了肿瘤的空间异质性特征,发现了两种不同的空间异质性模式与肺腺癌病人的复发和死亡风险相关,探讨了这两种空间异质性肿瘤的肿瘤细胞DNA拷贝数变化、转录组特性和免疫微环境特征,为深入研究肿瘤空间异质性提供了可行的技术路线和计算方法。
肺癌是最常见的癌症类型,也是全世界癌症死亡的主要原因。肺腺癌是非小细胞肺癌最常见的亚型,其特点是肿瘤之间的异质性和单个肿瘤中不同区域之间的异质性较高。这种区域间的异质性,即肿瘤内异质性,已被证明与先存耐药亚克隆(pre-existing resistant subclones)的扩增和治疗失败相关。例如,在用EGFR酪氨酸激酶抑制剂(TKIs)治疗非小细胞肺癌的过程中观察到EGFR T790M阳性细胞。此外,一些研究表明,在包括非小细胞肺癌在内的多种癌症类型中,某些肿瘤内异质性模式,主要是以亚克隆变异负担(subclonal alteration burden)来衡量,与不良的临床预后相关。目前的研究试图用多区域分析法来解析这种异质性的空间模式。然而,每个肿瘤所分析的区域数量很少,限制了这些研究的结论,也导致我们对肿瘤内空间异质性及其对癌症进展和免疫微环境贡献的理解存在较大空白。
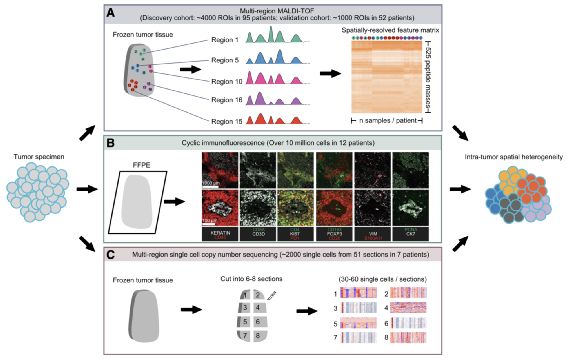
本文利用多区域质谱分析(>5000个区域),多区域单细胞拷贝数测序(~2000个单细胞),以及循环免疫荧光(>1000万个细胞)三种空间组学技术,对147名肺腺癌患者的肿瘤进行了空间多组学分析(图 1)。在肿瘤中发现了两种不同的空间异质性模式,定义为聚集模式(Clustered)和随机模式(Random)空间多样性(geographic diversification, GD)。并利用蛋白质组和基因组数据在相同的样本中观察到了一致的空间模式。随机模式的肿瘤,其特点是细胞粘附力不足和肿瘤互作内皮细胞水平较低,在两个独立的患者队列中与复发或死亡风险的增加显著相关。
图 1. 空间多组学技术解析肿瘤内空间异质性。
本文首先利用多区域质谱分析数据,提出了肿瘤内空间异质性的计算方法和分类标准。借助地理学方法定义了肿瘤内细胞的两种空间异质性模式:聚集模式(Clustered GD)和随机模式(Random GD),并发现随机模式的癌症患者具有更高的复发几率和死亡风险(图 2)。
图 2. 癌症内空间异质性的计算和与病人预后的关系。
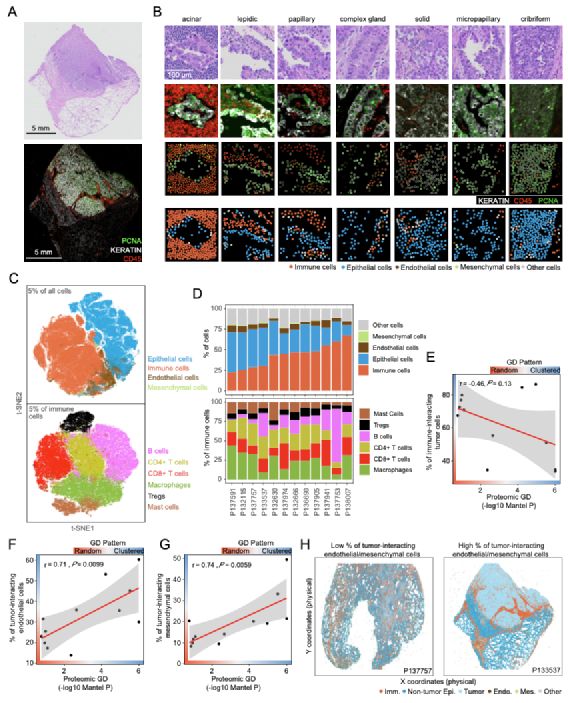
接下来作者利用转录组数据发现随机模式的肿瘤具有细胞粘附力不足和免疫激活的特征。并利用循环免疫荧光技术分析了12个肺腺癌肿瘤的>1000万个单细胞空间信息,作者设计了一种快速的一致性聚类算法对海量单细胞进行了聚类和注释,从而发现随机模式的肿瘤具有肿瘤互作内皮细胞水平较低和浸润免疫细胞较高的特征(图 3)。
图 3. 循环免疫荧光技术研究不同空间模式的肿瘤微环境。
之后作者利用多区域单细胞拷贝数测序发现肿瘤细胞在DNA拷贝数水平上也具有相同的两种空间异质性模式,并证明在相同病人中,蛋白层面和DNA拷贝数层面上具有一致的空间异质性模式。进一步的分析表明特定基因的拷贝数扩增在相同肿瘤的不同区域具有不同的克隆型,揭示了更加复杂的DNA拷贝数扩增的空间异质性模式(图 4)。
图 4. 多区域DNA拷贝数测序研究肿瘤空间异质性。
本研究利用多种空间组学技术,结合多组学计算方法,构建肺腺癌中肿瘤内异质性的空间图谱,对肿瘤内空间异质性的机制和与临床预后的关系提出了新的见解,为空间多组学技术在癌症中的研究提供了新的思路。
吴华君课题组主要利用生物统计和机器学习等技术进行多组学数据整合和相关算法的开发。目前课题组正在招聘有多组学计算背景的博士后和科研助理若干。欢迎有志于进行多组学计算方法开发和数据挖掘的优秀人才加入团队,课题组将提供一流的科研环境和良好的个人发展空间。有意者请将个人简历(包括学习工作经历、科研业绩、发表论文、联系方式等)发送至[email protected](邮件标题请注明:应聘-申请的岗位-姓名)。
招聘详情见:https://multiomics-bjmu.github.io/careers
原文链接: https://www.cell.com/cell-genomics/fulltext/S2666-979X(22)00107-0
1. 北京大学吴华君课题组高新诚聘 医学生信 助理研究员和博士后
2. Nat. Commun.|北京大学吴华君,基因组3D结构调控胚胎干细胞分化
往期精品(点击图片直达文字对应教程)
机器学习
后台回复“生信宝典福利第一波”或点击阅读原文获取教程合集