手写算法-python代码实现DBSCAN
手写算法-python代码实现DBSCAN
- 原理解析
- 代码实现
- 实例演示与sklearn对比
- 总结
原理解析
上篇文章我们优化了Kmeans聚类算法,最后留下一个问题:
Kmeans只适合处理凸样本集,不适合处理非凸样本集,这个问题,怎么解决?
链接: 手写算法-python代码实现Kmeans++以及优化
生成演示数据集:
import numpy as np
import matplotlib.pyplot as plt
from sklearn.datasets import make_circles,make_blobs,make_moons
#演示数据集
x2,y2 = make_moons(n_samples=500,noise = 0.1,random_state=2020)
plt.scatter(x2[:,0],x2[:,1],marker='o')
plt.show()
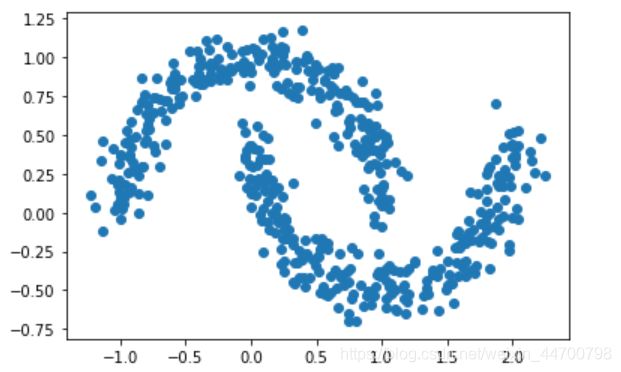
从图像上看,这个数据集,我们适合聚类成2个类别,下面我们用sklearn里面的Kmeans算法来演示一下:
from sklearn.cluster import KMeans
Kmeans = KMeans(n_clusters=2)
y_preds = Kmeans.fit_predict(x2)
plt.scatter(x2[:,0],x2[:,1],c=y_preds,marker='o')
plt.show()
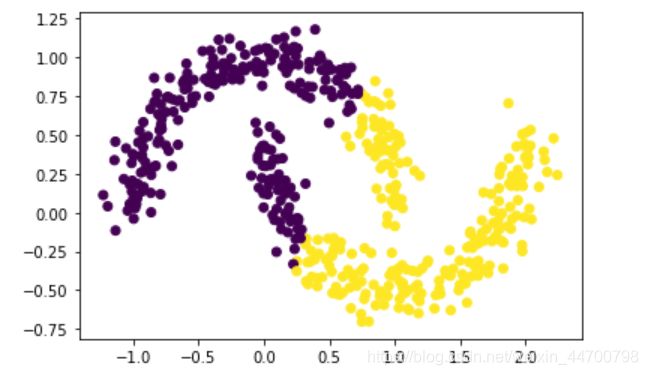
然后聚类结果是这样,对于这种非凸样本集,该怎么处理呢?
我们一般用DBSCAN聚类算法。
DBSCAN(Density-Based Spatial Clustering of Applications with Noise),
直译就是:具有噪声的基于密度的聚类方法,2个特点:
1、可以找出噪声数据;
2、基于密度的聚类算法(Kmeans是基于划分);
原理解析:
下面引用刘建平博士的一段话:
DBSCAN是基于一组邻域来描述样本集的紧密程度的,参数(ϵ, MinPts)用来描述邻域的样本分布紧密程度。其中,ϵ描述了某一样本的邻域距离阈值,MinPts描述了某一样本的距离为ϵ的邻域中样本个数的阈值。
假设我的样本集是D=(x1,x2,…,xm),则DBSCAN具体的密度描述定义如下:
1) ϵ-邻域:对于xj∈D,其ϵ-邻域包含样本集D中与xj的距离不大于ϵ的子样本集,即Nϵ(xj)={xi∈D|distance(xi,xj)≤ϵ},
这个子样本集的个数记为|Nϵ(xj)|2) 核心对象:对于任一样本xj∈D,如果其ϵ-邻域对应的Nϵ(xj)至少包含MinPts个样本,即如果|Nϵ(xj)|≥MinPts,则xj是核心对象。
3)密度直达:如果xi位于xj的ϵ-邻域中,且xj是核心对象,则称xi由xj密度直达。注意反之不一定成立,即此时不能说xj由xi密度直达,
除非且xi也是核心对象。4)密度可达:对于xi和xj,如果存在样本样本序列p1,p2,…,pT,满足p1=xi,pT=xj, 且pt+1由pt密度直达,则称xj由xi密度可达。也就是说,密度可达满足传递性。此时序列中的传递样本p1,p2,…,pT−1均为核心对象,因为只有核心对象才能使其他样本密度直达。注意密度可达也不满足对称性,这个可以由密度直达的不对称性得出。
5)密度相连:对于xi和xj,如果存在核心对象样本xk,使xi和xj均由xk密度可达,则称xi和xj密度相连。注意密度相连关系是满足对称性的。
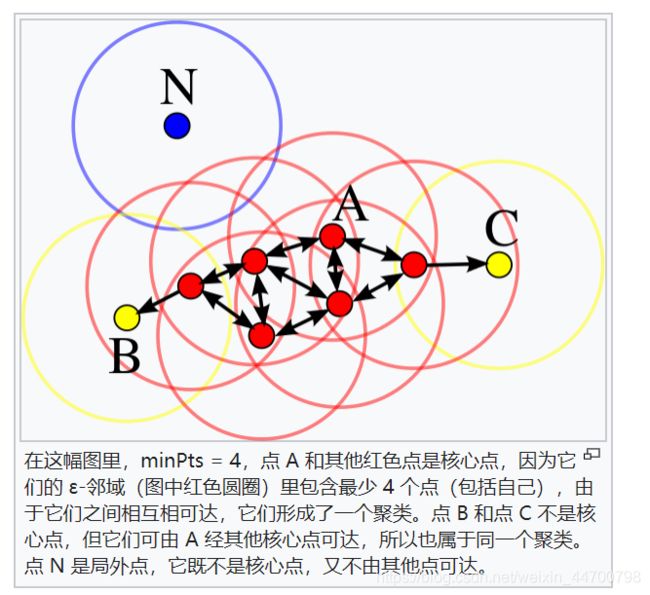
算法过程:
1、DBSCAN 需要两个参数:ε (eps) 和形成高密度区域所需要的最少点数 (minPts),它由一个任意未被访问的点开始,然后探索这个点的 ε-邻域,如果 ε-邻域里有足够的点,则建立一个新的聚类,否则这个点被标签为噪声。注意这个点之后可能被发现在其它点的 ε-邻域里,而该 ε-邻域可能有足够的点,届时这个点会被加入该聚类中。
2、如果一个点位于一个聚类的密集区域里,它的 ε-邻域里的点也属于该聚类,当这些新的点被加进聚类后,如果它(们)也在密集区域里,它(们)的 ε-邻域里的点也会被加进聚类里。这个过程将一直重复,直至不能再加进更多的点为止,这样,一个密度连结的聚类被完整地找出来。然后,一个未曾被访问的点将被探索,从而发现一个新的聚类或噪声。
代码实现
根据上面所诉,我们来编写python代码实现DBSCAN:
class DBscan:
def __init__(self,eps,MinPts):
self.eps = eps
self.MinPts = MinPts
def regionQuery(self,i,x):
diff = i - x
distances = np.sqrt(np.square(diff).sum(axis=1))
#返回邻域点的索引
return list(np.where(distances <= self.eps)[0])
def fit(self,x):
#初始标签为-1
label = -1
m =len(x)
#初始化所有样本点所属的类别,定为-1
cluster = [-1 for i in range(m)]
#已访问样本点
visited = []
for p in range(m):
if p not in visited:
NeighborPts = self.regionQuery(x[p],x)
if len(NeighborPts) < self.MinPts:
#默认标记为-1,后面可能会被加入到其他的邻域内,成为一个密度可达点
continue
else:
label += 1
cluster[p] = label
#列表NeighborPts是动态变化的,添加没有访问过的全部密度可达点,划分到一个簇
for p_1 in NeighborPts:
if p_1 not in visited:
visited.append(p_1)
Ner_NeighborPts = self.regionQuery(x[p_1],x)
if len(Ner_NeighborPts) >= self.MinPts:
for a in Ner_NeighborPts:
if a not in NeighborPts:
NeighborPts.append(a)
#同时把p_1的类别改为label
cluster[p_1] = label
return cluster
实例演示与sklearn对比
下面,我们生成数据集,来查看我们写的DBscan聚类效果
1、小数据集验证
import numpy as np
import matplotlib.pyplot as plt
from sklearn.cluster import DBSCAN
from sklearn.datasets import make_circles,make_blobs,make_moons
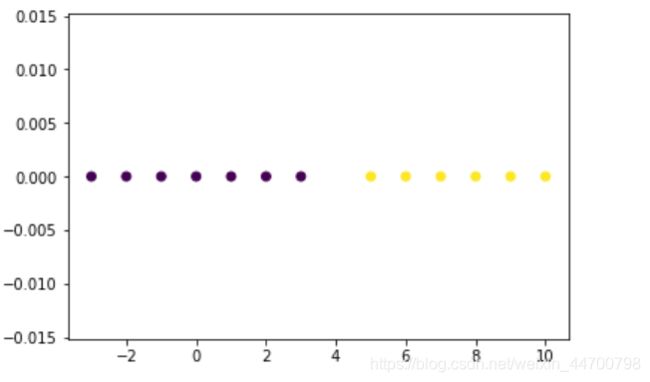
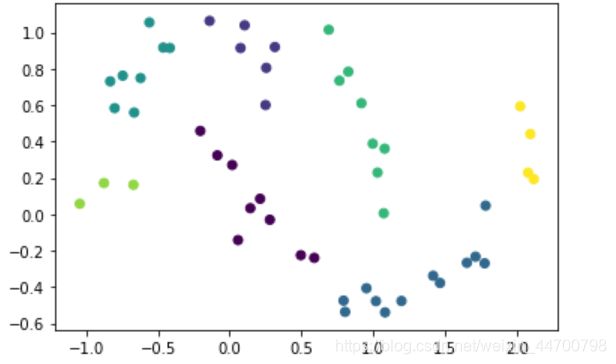
x= np.array([[-3,0],[-2,0],[-1,0],[0,0],[1,0],[2,0],[3,0],[5,0],[6,0],[7,0],[8,0],[9,0],[10,0]])
Dbscan =DBscan(eps=2,MinPts=5)
y_pred = Dbscan.fit(x)
plt.scatter(x[:,0],x[:,1],c=y_pred,marker='o')
plt.show()
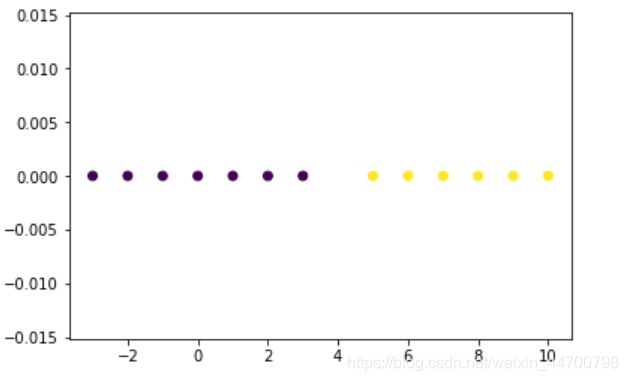
model = DBSCAN(eps=2,min_samples=5)
y_preds = model.fit_predict(x)
plt.scatter(x[:,0],x[:,1],c=y_preds,marker='o')
plt.show()
2、大数据集验证
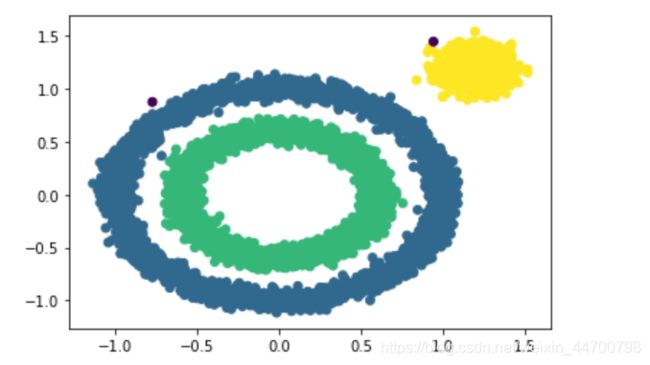
x1,y1 = make_circles(n_samples=5000,factor=0.6,noise=0.05,random_state=2020)
x2,y2 = make_blobs(n_samples=1000,n_features=2,centers=[[1.2,1.2]],cluster_std=0.1,random_state=9)
x1 = np.concatenate((x1,x2))
Dbscan =DBscan(eps=0.1,MinPts=10)
y_pred = Dbscan.fit(x1)
plt.scatter(x1[:,0],x1[:,1],c=y_pred,marker='o')
plt.show()
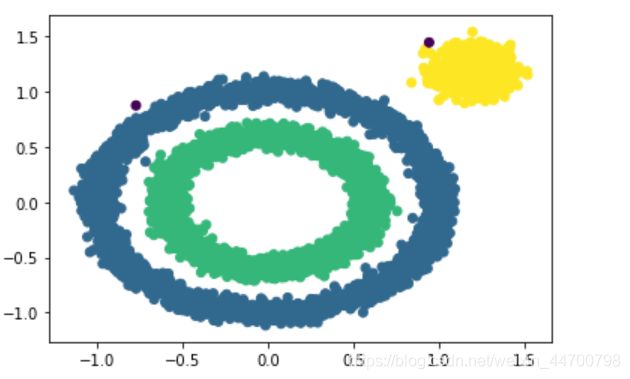
以下是sklearn对比:
model = DBSCAN(eps=0.1,min_samples=10)
y_preds = model.fit_predict(x1)
plt.scatter(x1[:,0],x1[:,1],c=y_preds,marker='o')
plt.show()
3、中等数据集
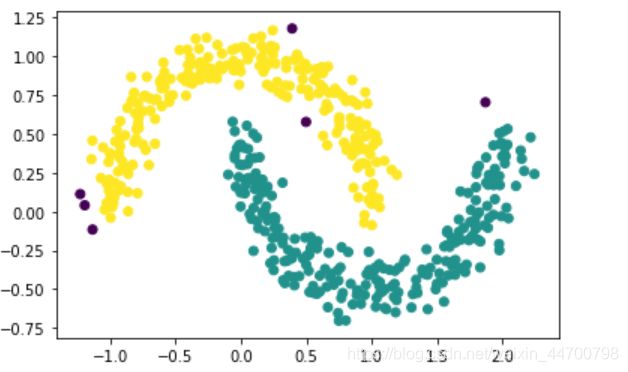
x2,y2 = make_moons(n_samples=500,noise = 0.1,random_state=2020)
Dbscan =DBscan(eps=0.14,MinPts=3)
y_pred = Dbscan.fit(x2)
plt.scatter(x2[:,0],x2[:,1],c=y_pred,marker='o')
plt.show()
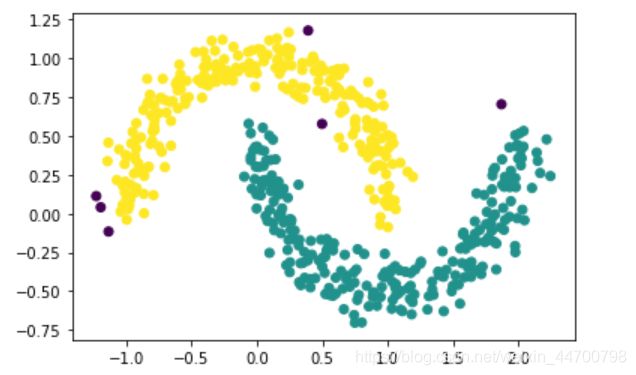
model = DBSCAN(eps=0.14,min_samples=3)
y_preds = model.fit_predict(x2)
plt.scatter(x2[:,0],x2[:,1],c=y_preds,marker='o')
plt.show()
x3,y3 = make_moons(n_samples=50,noise = 0.1,random_state=2020)
Dbscan =DBscan(eps=0.3,MinPts=3)
y_pred = Dbscan.fit(x3)
plt.scatter(x3[:,0],x3[:,1],c=y_pred,marker='o')
plt.show()
model = DBSCAN(eps=0.3,min_samples=3)
y_preds = model.fit_predict(x3)
plt.scatter(x3[:,0],x3[:,1],c=y_preds,marker='o')
plt.show()
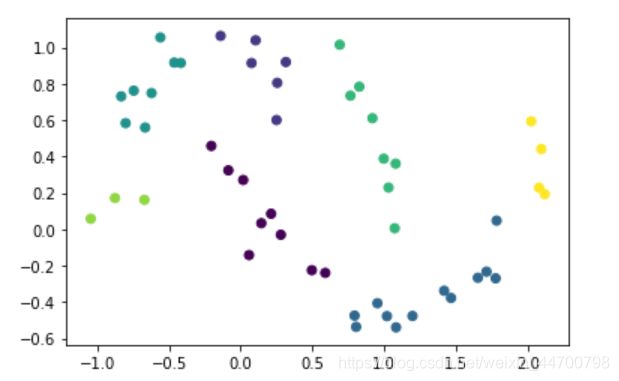
OK,结果都一样,证明我们写的DBSCAN是对的。
(Ps:为什么验证这么多次,因为写DBSCAN代码时,验证的时候老有bug,终于修改好了。。。)
总结
刚才验证大数据集时,为什么sklearn速度比较快,是因为:

参数‘“algorithm”,算法一共有三种,第一种是蛮力实现,第二种是KD树实现,第三种是球树实现。K近邻法(KNN)中也是这样,一共有4种可选输入,‘brute’对应蛮力实现,也就是我们这种,‘kd_tree’对应第二种KD树实现,‘ball_tree’对应第三种的球树实现, ‘auto’则会在上面三种算法中做权衡,选择一个拟合最好的最优算法。需要注意的是,如果输入样本特征是稀疏的时候,无论我们选择哪种算法,最后scikit-learn都会去用蛮力实现‘brute’。
默认的距离也是欧氏距离。
和传统的K-Means算法相比,DBSCAN最大的不同就是不需要输入类别数k,当然它最大的优势是可以发现任意形状的聚类簇,而不是像K-Means,一般仅仅使用于凸的样本集聚类。同时它在聚类的同时还可以找出异常点,这点和BIRCH算法类似。
那么我们什么时候需要用DBSCAN来聚类呢?一般来说,如果数据集是稠密的,并且数据集不是凸的,那么用DBSCAN会比K-Means聚类效果好很多。如果数据集不是稠密的,则不推荐用DBSCAN来聚类。
下面对DBSCAN算法的优缺点做一个总结。
DBSCAN的主要优点有:
1) 可以对任意形状的稠密数据集进行聚类,相对的,K-Means之类的聚类算法一般只适用于凸数据集。
2) 可以在聚类的同时发现异常点,对数据集中的异常点不敏感。
3) 聚类结果没有偏倚,相对的,K-Means之类的聚类算法初始值对聚类结果有很大影响。
DBSCAN的主要缺点有:
1)如果样本集的密度不均匀、聚类间距差相差很大时,聚类质量较差,这时用DBSCAN聚类一般不适合。
2) 如果样本集较大时,聚类收敛时间较长,此时可以对搜索最近邻时建立的KD树或者球树进行规模限制来改进。
3) 调参相对于传统的K-Means之类的聚类算法稍复杂,主要需要对距离阈值ϵ,邻域样本数阈值MinPts联合调参,不同的参数组合对最后的聚类效果有较大影响。
DBSCAN就写到这里。