数据挖掘与机器学习作业_03 特征筛选
特征筛选
import pandas as pd
import matplotlib
import matplotlib.pyplot as plt
import matplotlib.font_manager as fm
import numpy as np
import scipy
import sklearn
import re
import sys
import random
# 方差
from sklearn.feature_selection import VarianceThreshold
# 相关性过滤
from sklearn.feature_selection import SelectKBest
from sklearn.feature_selection import chi2
# 皮尔逊相关系数
from scipy.stats import pearsonr
from collections import OrderedDict
# 使用决策树筛选特征
from sklearn import tree
# 互信息法
from sklearn.feature_selection import mutual_info_classif as MIC
from sklearn.feature_selection import SelectKBest,chi2
from my_tools import *
import warnings
warnings.filterwarnings("ignore")
读取数据
jibing = pd.read_excel("./jibing_yuchuli_final.xlsx")
jibing.shape
(1598, 63)
消去方差为 0 的属性
selector = VarianceThreshold() #实例化,不填参数默认方差为0
jibing_var0 = selector.fit_transform(jibing) #获取删除不合格特征之后的新特征矩阵
jibing_var0.shape
(1598, 63)
jibing.head()
| 左右 | 是否外伤 | 症状持续时间 | 明显夜间痛 | 性别 | 年龄 | 高血压 | 高血脂 | 2型糖尿病 | 吸烟与否 | ... | 果糖胺 | 肌酸激酶 | α-L-盐藻糖苷酶 | 乳酸 | 淀粉酶 | 同型半胱氨酸 | 铁 | 总铁结合力 | 血型 | 结果 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 3 | 0 | 0 | 65 | 1 | 0 | 0 | 0 | ... | 1.32 | 48.0 | 12.0 | 1.9 | 49.0 | 9.9 | 12.3 | 43.5 | 3 | 0 |
| 1 | 1 | 1 | 2 | 0 | 0 | 62 | 1 | 0 | 0 | 0 | ... | 1.67 | 77.0 | 16.0 | 1.4 | 81.0 | 9.2 | 16.9 | 55.5 | 0 | 1 |
| 2 | 1 | 0 | 4 | 1 | 0 | 55 | 0 | 0 | 0 | 0 | ... | 1.86 | 78.0 | 22.0 | 1.9 | 89.0 | 9.9 | 7.0 | 51.4 | 0 | 1 |
| 3 | 1 | 0 | 3 | 0 | 0 | 60 | 0 | 0 | 0 | 0 | ... | 1.68 | 92.0 | 12.0 | 1.4 | 69.0 | 9.3 | 15.8 | 53.0 | 0 | 0 |
| 4 | 0 | 1 | 3 | 0 | 0 | 61 | 0 | 0 | 0 | 0 | ... | 1.60 | 58.0 | 14.0 | 1.7 | 153.0 | 8.1 | 13.2 | 45.9 | 0 | 1 |
5 rows × 63 columns
相关性分析
皮尔逊相关系数
col = jibing.columns.tolist()
col.remove("结果")
dict_ = dict()
feature_ls = []
for col_ in col:
corr, p = pearsonr(jibing["结果"], jibing[col_])
"""
p > 0.05 才会有统计学意义
所以这里将 p > 0.05 的特征放到一个字典中
查看他们与结果的相关性
"""
if p > 0.05:
feature_ls.append(col_)
dict_[col_] = abs(corr)
len(dict_)
59
feature_ls
['左右',
'症状持续时间',
'明显夜间痛',
'性别',
'年龄',
'高血压',
'高血脂',
'2型糖尿病',
'吸烟与否',
'饮酒与否',
'红细胞计数*10^12/L',
'血红蛋白',
'红细胞压积',
'血小板计数',
'血小板压积',
'总蛋白g/L',
'白蛋白g/L',
'球蛋白g/L',
'白球比',
'ALT丙氨酸氨基转移酶',
'AST天门冬氨酸氨基转移酶',
'碱性磷酸酶',
'谷氨酸转肽酶',
'AST:ALT',
'总胆红素',
'直接胆红素',
'间接胆红素',
'钾',
'钠',
'氯',
'钙',
'磷',
'镁',
'葡萄糖',
'尿素',
'尿酸',
'甘油三酯',
'总胆固醇',
'H高密度胆固醇',
'L低密度胆固醇',
'载脂蛋白A1',
'载脂蛋白B',
'载脂蛋白E mg/l',
'aPoB/aPoA1',
'脂蛋白小a',
'乳酸脱氢酶LDH',
'β-2微球蛋白',
'胆碱酯酶',
'前白蛋白mg/l',
'总胆汁酸',
'腺苷脱氨酶ADA',
'果糖胺',
'肌酸激酶',
'α-L-盐藻糖苷酶',
'淀粉酶',
'同型半胱氨酸',
'铁',
'总铁结合力',
'血型']
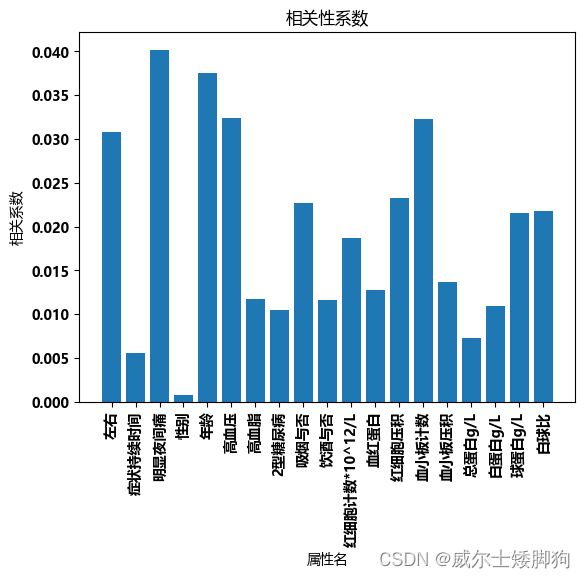
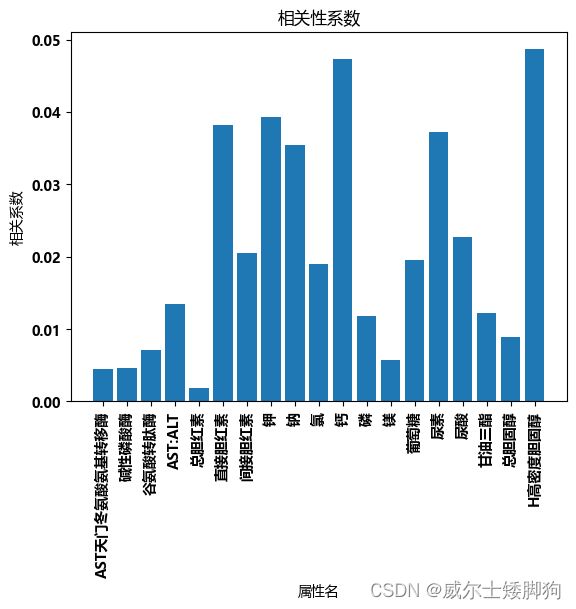
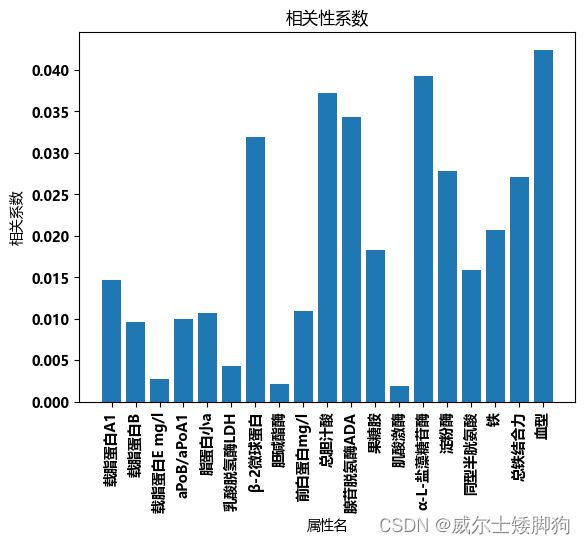
# 设置字体
set_font()
start = 0
end = 19
for i in range(3):
# 使用自定义字体
show_x = list(dict_.keys())[start:end]
show_y = list(dict_.values())[start:end]
plt.xlabel('属性名')
plt.ylabel('相关系数')
# 绘制柱状图
plt.bar(show_x, show_y)
# 显示图形
start = start + 20
end = end + 20
plt.xticks(rotation=90)
plt.title("相关性系数")
plt.show()
使用决策树筛选特征
clf = tree.DecisionTreeClassifier(random_state=30)
clf = clf.fit(jibing.iloc[:,:-1],jibing.iloc[:,-1])
获取特征的重要性
根据决策树的某些系数划分重要程度
tree_importance = clf.feature_importances_
tree_importance
array([0.00642058, 0.00428039, 0.00499379, 0.02252402, 0. ,
0.01654977, 0.01911454, 0. , 0. , 0. ,
0. , 0.03454957, 0.00667023, 0.0113456 , 0.02181291,
0.008653 , 0.0320874 , 0.06006682, 0.02659235, 0.02146013,
0.00285359, 0. , 0.01941168, 0.01444173, 0.02098024,
0.00456575, 0.02118258, 0.00256741, 0.01527166, 0.01516202,
0.02008489, 0.00494003, 0. , 0.01235312, 0.03628761,
0.02015343, 0.02685477, 0.03634301, 0.01141437, 0.03151978,
0.01307189, 0.00508406, 0.01698537, 0.00808379, 0.01866136,
0.02006391, 0.0165685 , 0.03874059, 0.01265598, 0.02484254,
0.01521916, 0.0041057 , 0.01748961, 0.0339876 , 0.00445256,
0.01425327, 0.02496806, 0.01092751, 0.01736746, 0.05684635,
0.01211596, 0. ])
由于特征是比较少的,所以将皮尔逊系数和决策树特征重要性放到一起考虑
# 确定一会儿要删除的特征
# 把它们放到drop_list中
drop_list = []
# 获取重要性为0的特征
indexes = [i for i, x in enumerate(tree_importance) if x == 0]
indexes
[4, 7, 8, 9, 10, 21, 32, 61]
for index in indexes:
if dict_[col[index]] < 0.01:
drop_list.append(col[index])
drop_list
['性别', 'AST天门冬氨酸氨基转移酶']
将无关属性去掉
jibing.shape
(1598, 63)
type(jibing)
pandas.core.frame.DataFrame
for name in drop_list:
jibing.drop(name, axis=1,inplace = True)
jibing.shape
(1598, 61)
特征和结果分开
jibing_res = jibing.iloc[:,-1]
jibing_res.head()
0 0
1 1
2 1
3 0
4 1
Name: 结果, dtype: int64
jibing = jibing.iloc[:,:-1]
jibing.head()
| 左右 | 是否外伤 | 症状持续时间 | 明显夜间痛 | 年龄 | 高血压 | 高血脂 | 2型糖尿病 | 吸烟与否 | 饮酒与否 | ... | 腺苷脱氨酶ADA | 果糖胺 | 肌酸激酶 | α-L-盐藻糖苷酶 | 乳酸 | 淀粉酶 | 同型半胱氨酸 | 铁 | 总铁结合力 | 血型 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 3 | 0 | 65 | 1 | 0 | 0 | 0 | 0 | ... | 10.0 | 1.32 | 48.0 | 12.0 | 1.9 | 49.0 | 9.9 | 12.3 | 43.5 | 3 |
| 1 | 1 | 1 | 2 | 0 | 62 | 1 | 0 | 0 | 0 | 0 | ... | 10.0 | 1.67 | 77.0 | 16.0 | 1.4 | 81.0 | 9.2 | 16.9 | 55.5 | 0 |
| 2 | 1 | 0 | 4 | 1 | 55 | 0 | 0 | 0 | 0 | 0 | ... | 15.0 | 1.86 | 78.0 | 22.0 | 1.9 | 89.0 | 9.9 | 7.0 | 51.4 | 0 |
| 3 | 1 | 0 | 3 | 0 | 60 | 0 | 0 | 0 | 0 | 0 | ... | 16.0 | 1.68 | 92.0 | 12.0 | 1.4 | 69.0 | 9.3 | 15.8 | 53.0 | 0 |
| 4 | 0 | 1 | 3 | 0 | 61 | 0 | 0 | 0 | 0 | 0 | ... | 13.0 | 1.60 | 58.0 | 14.0 | 1.7 | 153.0 | 8.1 | 13.2 | 45.9 | 0 |
5 rows × 60 columns
jibing
| 左右 | 是否外伤 | 症状持续时间 | 明显夜间痛 | 年龄 | 高血压 | 高血脂 | 2型糖尿病 | 吸烟与否 | 饮酒与否 | ... | 腺苷脱氨酶ADA | 果糖胺 | 肌酸激酶 | α-L-盐藻糖苷酶 | 乳酸 | 淀粉酶 | 同型半胱氨酸 | 铁 | 总铁结合力 | 血型 | |
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 0 | 0 | 0 | 3 | 0 | 65 | 1 | 0 | 0 | 0 | 0 | ... | 10.0 | 1.32 | 48.0 | 12.0 | 1.9 | 49.0 | 9.9 | 12.3 | 43.5 | 3 |
| 1 | 1 | 1 | 2 | 0 | 62 | 1 | 0 | 0 | 0 | 0 | ... | 10.0 | 1.67 | 77.0 | 16.0 | 1.4 | 81.0 | 9.2 | 16.9 | 55.5 | 0 |
| 2 | 1 | 0 | 4 | 1 | 55 | 0 | 0 | 0 | 0 | 0 | ... | 15.0 | 1.86 | 78.0 | 22.0 | 1.9 | 89.0 | 9.9 | 7.0 | 51.4 | 0 |
| 3 | 1 | 0 | 3 | 0 | 60 | 0 | 0 | 0 | 0 | 0 | ... | 16.0 | 1.68 | 92.0 | 12.0 | 1.4 | 69.0 | 9.3 | 15.8 | 53.0 | 0 |
| 4 | 0 | 1 | 3 | 0 | 61 | 0 | 0 | 0 | 0 | 0 | ... | 13.0 | 1.60 | 58.0 | 14.0 | 1.7 | 153.0 | 8.1 | 13.2 | 45.9 | 0 |
| ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... | ... |
| 1593 | 0 | 0 | 4 | 1 | 76 | 1 | 0 | 0 | 0 | 0 | ... | 12.0 | 1.61 | 60.0 | 5.0 | 1.5 | 60.0 | 7.9 | 21.5 | 50.1 | 3 |
| 1594 | 1 | 1 | 3 | 1 | 58 | 1 | 0 | 0 | 0 | 0 | ... | 16.0 | 1.92 | 96.0 | 22.0 | 1.1 | 89.0 | 10.3 | 16.3 | 55.2 | 3 |
| 1595 | 0 | 1 | 3 | 1 | 68 | 0 | 0 | 0 | 0 | 0 | ... | 11.0 | 1.84 | 84.0 | 21.0 | 2.3 | 43.0 | 13.1 | 17.3 | 46.7 | 0 |
| 1596 | 0 | 0 | 3 | 1 | 62 | 0 | 0 | 0 | 0 | 0 | ... | 19.0 | 2.30 | 76.0 | 36.0 | 1.1 | 125.0 | 18.8 | 24.0 | 51.5 | 0 |
| 1597 | 0 | 0 | 4 | 0 | 53 | 0 | 0 | 0 | 0 | 0 | ... | 14.0 | 1.54 | 77.0 | 11.0 | 1.3 | 79.0 | 11.0 | 11.5 | 54.0 | 0 |
1598 rows × 60 columns
保存
jibing.to_excel("./jibing_feature_final.xlsx", index=False)
jibing_res.to_excel("./jibing_feature_res_final.xlsx", index=False)