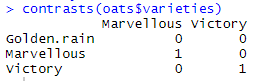r语言结构方程模型可视化_混合线性模型——R语言
线性混合模型简介
混合线性模型(mixed linear model)是一种方差分量模型。在方差分量模型中,把既含有固定效应,又含有随机效应的模型,称为混合线性模型 【信息来源:百度】 一般线性模型中仅包含固定效应和噪声两项影响因素,也就是不会考虑随机因素对模型的影响。 数据集:R的MASS包中oast数据集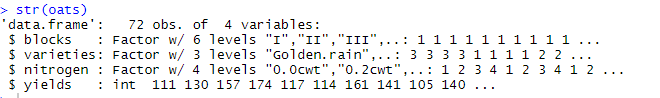 数据解释 : 通过三个品种四个水平的施肥处理,对燕麦进行了小区试验。实验分为6个区块,每个区块分为4个子区块。主小区采用品种,次小区采用施肥处理。 线性模型是需要求出关于在因变量上的权重(斜率),那么针对分类型的因变量要怎么计算呢?——创建虚拟变量代替我们的分类水平进行运算。
数据解释 : 通过三个品种四个水平的施肥处理,对燕麦进行了小区试验。实验分为6个区块,每个区块分为4个子区块。主小区采用品种,次小区采用施肥处理。 线性模型是需要求出关于在因变量上的权重(斜率),那么针对分类型的因变量要怎么计算呢?——创建虚拟变量代替我们的分类水平进行运算。
虚拟变量
因变量属于分类变量,所以在建立模型之前需要创建虚拟变量:虚拟变量的作用就是对固定因子的因子进行量化。例如:在varieties中有三种不同的品种分别为:Golden.rain、Marvellous、Victory,可以通过R的contrasts()函数查看或设置不同品种的虚拟变量。
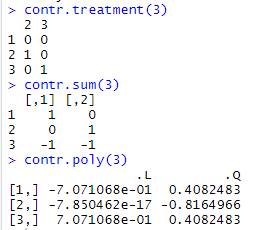
默认采用treatment编码方式,每一列的求和等于1,至于为什么Golden.rain该行全都是0,因为默认情况下,第一行是其他组的参照组。注:若是采用sum编码方式:每一列的求和等于0。
展示一下treatment编码方式和sum编码方式。注:contr.sum是定义无序因子的,若为有序可以采用contr.poly来定义。
例如:在3水平的因子变量上展示
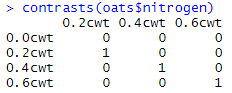
同理nitrogen变量的虚拟变量编码方式如下:
观察两张图片可以得到关于虚拟变量创建的规律:1、对于某一个因子型变量包含K个水平的因子,那么会产生K-1个对比向量;
2、对比向量以一个K行*(K-1)列的矩阵组成。
3、默认的情况下以第一行作为其他因子的参照。(比如:Golden.rain,0.0cwt)
那么有了虚拟变量后要如何计算呢?
1、单因素多水平(treatment编码方式):
运算公式:y=b+wx+beta ;其中,b是截距,w是权重(斜率),beta(随机误差)
Y = b+w1*x1+w2*x1+beta
A水平:YA = b+w1*0+w2+0+beta = b+beta
B水平:YB = b+w1*1+w2*0+beta = b+w1+beta
C水平:YC = b+w1*0+w2*1+beta = b+w2+beta
那么模型的回归系数就等于:
W1=YB — YA
W2=YC — YA
结论:回归系数等于真实效应差异。
2、两因素两水平有交互作用的情况(sum编码方式):
Y=b+w1*Xa+w2*Xb+w3*Xa*Xb+beta
主效应的回归系数:
Ya1=b-w1-w2+beta
Ya2=b+w1-w2+beta
W1=(Ya2-Ya1)/2
同理:
W2=(Yb2-Yb1)/2
结论:主效应的回归系数等于真实效应的一半。
交互效应的回归系数:
Ya1b1=b-w1-w2+w3+beta
Ya1b2=b-w1+w2-w3+beta
Ya2b1=b+w1-w2-w3+beta
Ya2b2=b+w1+w2+w3+beta
W3=((Ya2b2-Ya2b1)-(Ya1b2-Ya1b1))/4
结论:交互效应等于真实交互效应的四分之一
你们也可以自己去尝试使用treatment编码方式,也就是将上面的Xa、Xb中的-1改为0。若不考虑交互作用的话,将公式中的w3*Xa*Xb剔除,再运算即可。
treatment编码方式做两因素两水平无交互作用时,其主效应的回归系数是等于真实效应的。若有交互作用时,其主效应的回归系数是不等于真实效应的,但交互作用的回归系数等于真实效应。
那么对于本案例中属于双因素多水平的怎么计算回归系数呢?
3、K因素N水平的情况下,如何保证回归系数等于真实的效应值?
R里面是没有这个函数的,可以自己设置编码。即每个因素产生N*(N-1)的矩阵,其中每一列代表一个比较,每一行代表一个水平。第一个水平一般是作为为参照组,其编码为-(1/N),每一列对应基线之外的某个水平与基线的对比,所在的行编码为1-(1/N),其他行编码为-(1/N)。你们可以带入公式去验证一下。
DV m for(i in 1:n){ for(j in 1:n-1){ if(i-1 == j) m[i,j] else m[i,j] } } return(m)} DV(3) [,1] [,2][1,] -0.3333333 -0.3333333[2,] 0.6666667 -0.3333333[3,] -0.3333333 0.6666667DV(4) [,1] [,2] [,3][1,] -0.25 -0.25 -0.25[2,] 0.75 -0.25 -0.25[3,] -0.25 0.75 -0.25[4,] -0.25 -0.25 0.75#将虚拟变量赋值给案例中固定因子contrasts(oats$varieties) contrasts(oats$nitrogen) contrasts(oats$varieties) [,1] [,2]Golden.rain -0.3333333 -0.3333333Marvellous 0.6666667 -0.3333333Victory -0.3333333 0.6666667contrasts(oats$nitrogen) [,1] [,2] [,3]0.0cwt -0.25 -0.25 -0.250.2cwt 0.75 -0.25 -0.250.4cwt -0.25 0.75 -0.250.6cwt -0.25 -0.25 0.75其中我们将blocks变量作为被试,那么我们还需要增加一个项目变量,因为不同的项目之间的差异也是需要考虑的。本例考虑每一个被试在每个条件下有12次观测。
#添加项目变量item for(i in 1:6){ item }oats 构建模型
建立混合线性模型:使用lme4包中的lmer函数lmer(data = , formula = y ~ Fixed_Factor + (Random_intercept + Random_Slope | Random_Factor))这里出现报错了,也就是随机效应的个数(144)大于样本数(72)。所以在建立模型过程,将过多的随机效应增加到模型中,当随机效应的个数大于样本数时会报错,这时需适当的调整、取消一些随机效应或者增加样本数。
题外话
* 表示即考虑固定因子的效应,也考虑其交互效应
+ 表示只考虑固定因子的效应,
:表示只考虑其交互效应
如果说对formula这个参数的设置不清楚,可以去看线性回归部分。点击转换
为了能继续下去,就需要对随机效应进行虚拟变量编码,有选择性的对随机效应选取。【注:模型会随着加入的随机效应的不同而不同。这里仅为了展示所以选择随机效应时比较随意,真实中需重复试验考察。】
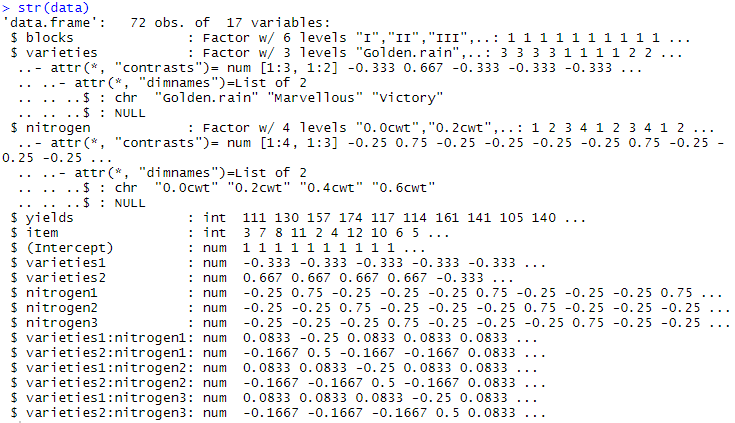
展示一下编码后的数据(由于数据太多了,仅展示数据结构)
由原本的4列变成了17列,增加13列。包括item(项目变量)以及从(intercept)开始后面的12项随机效应的。
构建虚拟变量具体代码如下:
mm head(mm)data View(data)同时为了方便,就建立一个零模型(不考虑协方差的作用),将被试和项目前面的竖线改为两条即可。以下为更改后的模型。
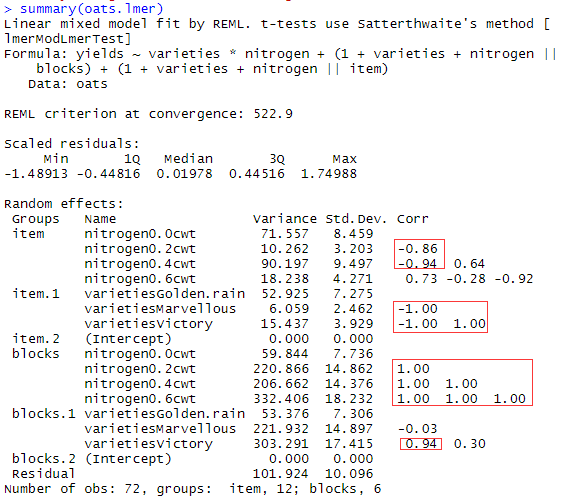
oats.lmer = lmer(yields ~ varieties*nitrogen + (1+varieties1+varieties2+nitrogen1+nitrogen2+nitrogen3+varieties1:nitrogen1+varieties2:nitrogen1+varieties1:nitrogen2+varieties2:nitrogen2+varieties1:nitrogen3+varieties2:nitrogen3 || blocks)+ (1+varieties1+varieties2+nitrogen1+nitrogen2+nitrogen3+varieties1:nitrogen1+varieties2:nitrogen1+varieties1:nitrogen2+varieties2:nitrogen2+varieties1:nitrogen3+varieties2:nitrogen3 || item), data = data)更改后的模型,尽管模型没有报错,但是还是出现警告了。这个警告的意思就是模型中出现了畸形协方差矩阵。一般情况下零是不会出现畸形协方差矩阵的,因为零模型不考虑协方差的作用。尽管有警告也没影响。
畸形协方差是什么意思呢?我列举一个简单的例子(如下所示的模型):
该模型也出现了畸形协方差矩阵。通过去观察随机效应可以得到为什么会存在警告。
在summary详细信息里面的randomeffects(随机效应),最右边的corr就表示协方差,可以看到有一些相关性是非常强的,有的还等于1了,这就是协方差矩阵了。
如何判断模 型是否存在畸形协方差?通过函数isSingular()检查,若返回为TRUE,则说明模型存在畸形协方差。
isSingular(oats.lmer)[1] TRUE注:模型有时候可能还会存在一种警告:模型会出现无法收敛的情况,也是通过调整或者取消一部分的随机效应。
回归正题
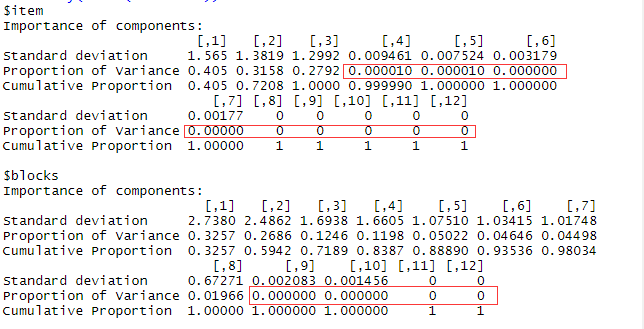
那么回到我们的零模型当中, 当出 现 这些问题的时候我们 要 如何去解决或者优化模型呢?可通过主成分分析,确认有效的随机效应的成分数量,也就是剔除哪些对模型没有影响的随机效应成分。
在item随机因子上的主成分效应,第二行表示解释一个随机截距和十一个随机斜率的效应的方差,从第4个主成分开始能解释的比例为0了,因此在item随机因子上有9个随机效应时多余的。虽然后面的4和5仍有贡献,但是很小就不考虑进来了,选择剔除了,但是具体哪些是多余的,目前还不清楚。同理在blocks随机因子上,第9个主成分开始能够解释的比例为0了,所以在blocks随机因子上有4个随机效应是多余的。在本次建模剔除item随机因子上的9个随机效应和blocks随机因子上的4个随机效应。
怎么确定需要剔除的随机效应呢?
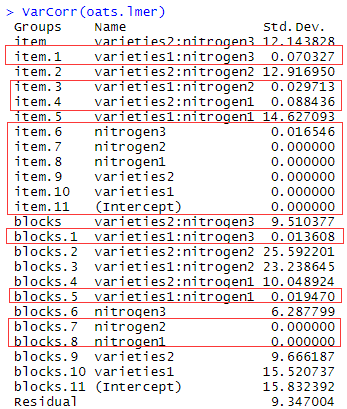
通过观察随机效应的标准差有两种方式:
一:通过查看原始模型中的Random effects中的Std.Dev
二:通过函数VarCorr()
两者的结果是一样的。
通过前面的主成分分析在item随机因子上剔除9个随机效应,也就是剔除标准差比较小的随机效应。同理在bolcks随机因子上剔除4个随机效应。重新建模以后发现仍存在畸形协方差,再次采用主成分优化,经过N次以后得到最终的模型即:仅考虑blocks随机因子下的随机截距,item随机因子下的品种2和施肥2的交互与品种2和施肥3的交互随机效应。具体模型如下:
oats.lmer1 = lmer(yields ~ varieties*nitrogen + (1| blocks)+ (-1+varieties2:nitrogen2+varieties2:nitrogen3 || item), data = data)尽管模型的没有报错和警告,但是模型的效果并不是很好吧!(可以尝试将其他随机效应放入模型中)
模型解读
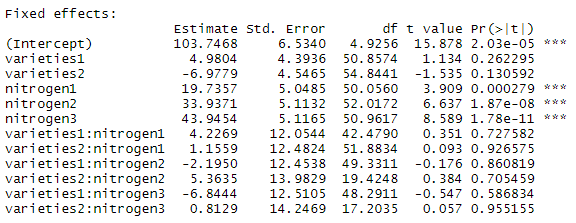
固定效应是通过summary(model)查看,其有四个部分:
一:Scaled residuals:包含标准差的最小值、中位数、四分位数。
二:Random effects:随机效应部分
三:Fixed effects:固定效应部分
四:Correlation of Fixed Effects:固定效应的协方差。
固定效应的结果如上图所示,这里是将varieties=Golden.rain和nitrogen=0.0cwt作为参照。可以看出nitrogen1、nitrogen2、nitrogen3(0.2cwt/0.4cwt/0.6cwt)其在参数估计(Estimate)要显著高于0.0cwt)。在品种varieties上和两者的交互作用上却没有显著差异。其余部分可自行去跑一下,这里由于篇幅问题就不展示了。
主效应和交互作用是通过anova(model)查看:
> anova(oats.lmer1)Type III Analysis of Variance Table with Satterthwaite's method Sum Sq Mean Sq NumDF DenDF F value Pr(>F) varieties 1530.6 765.3 2 51.918 3.5552 0.03572 * nitrogen 18146.0 6048.7 3 50.194 28.0982 8.313e-11 ***varieties:nitrogen 277.5 46.2 6 38.619 0.2148 0.96984 ---Signif. codes: 0 ‘***’ 0.001 ‘**’ 0.01 ‘*’ 0.05 ‘.’ 0.1 ‘ ’ 1结果如上,可看出燕麦的品种和施肥处理上的主效应显著,但其交互作用都不显著。
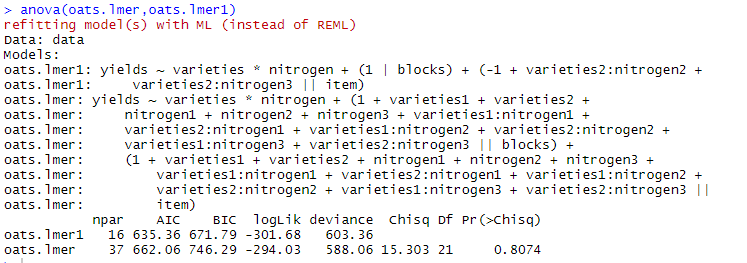
最后我们通过方差分析判断两个模型是否存在差异
方差分析的P值等于0.8,也就是模型间不存在显著性差异。即两个模型是一样的。
事后检验
模型的主效应显著后需要进行事后比较
由于我们模型的交互作用并不显著仅是主效应显著,那么对主效应进行多重比较。首先对品种(varieties)做多重比较。结果如下:
> emmeans(oats.lmer1,pairwise~varieties,adjust="none")$emmeans varieties emmean SE df lower.CL upper.CL Golden.rain 104.4 7.03 6.53 87.5 121 Marvellous 109.4 7.00 6.47 92.6 126 Victory 97.4 7.22 6.93 80.3 115Results are averaged over the levels of: nitrogen Degrees-of-freedom method: kenward-roger Confidence level used: 0.95 $contrasts contrast estimate SE df t.ratio p.value Golden.rain - Marvellous -4.98 4.50 49.6 -1.106 0.2742 Golden.rain - Victory 6.98 4.77 54.9 1.462 0.1495 Marvellous - Victory 11.96 4.67 50.2 2.562 0.0134 Results are averaged over the levels of: nitrogen Degrees-of-freedom method: kenward-rogercontrasts返回的就是多重比较的结果。可以看到只有品种(marvellous)与品种(victory)是存在显著差异的。也可以通过emmeans返回的结果中可以看到品种(marvellous)燕麦的产量均值是109.4而品种(victory)的产量均值为97.4。
我们再来看看施肥处理(nitrogen)的多重比较。结果如下:
> emmeans(oats.lmer1,pairwise~nitrogen,adjust="none")$emmeans nitrogen emmean SE df lower.CL upper.CL 0.0cwt 79.3 7.26 7.37 62.4 96.3 0.2cwt 99.1 7.23 7.31 82.1 116.0 0.4cwt 113.3 7.35 7.61 96.2 130.4 0.6cwt 123.3 7.45 7.81 106.0 140.5Results are averaged over the levels of: varieties Degrees-of-freedom method: kenward-roger Confidence level used: 0.95 $contrasts contrast estimate SE df t.ratio p.value 0.0cwt - 0.2cwt -19.7 5.17 49.1 -3.819 0.0004 0.0cwt - 0.4cwt -33.9 5.26 50.6 -6.452 <.0001> 0.0cwt - 0.6cwt -43.9 5.29 50.4 -8.314 <.0001> 0.2cwt - 0.4cwt -14.2 5.30 47.3 -2.680 0.0101 0.2cwt - 0.6cwt -24.2 5.43 45.7 -4.461 0.0001 0.4cwt - 0.6cwt -10.0 5.54 54.3 -1.807 0.0763 Results are averaged over the levels of: varieties Degrees-of-freedom method: kenward-roger同理contrasts返回的是施肥处理的多重比较结果。从上面可以看到除了0.4cwt与0.6cwt不存在显著差异外,其余的均存在显著性差异。
总结
一般来说做事后检验时,首先看交互作用,交互作用显著的话,就需要进行简单分析,简单分析中主效应显著了,再做多重比较。若交互作用不显著,那就直接对主效应做多重比较。也就是说,当你交互作用显著时,那么单纯做主效应分析的意义不大了,总之,交互作用的显著性优先考虑。(虽然该模型的交互作用不显著,但是还是走一遍流程吧!)
例子:
> #简单效应分析:比较不同品种上施肥处理的主效应> joint_tests(oats.lmer1,by="varieties")varieties = Golden.rain: model term df1 df2 F.ratio p.value nitrogen 3 40.31 9.431 0.0001 varieties = Marvellous: model term df1 df2 F.ratio p.value nitrogen 3 42.20 7.063 0.0006 varieties = Victory: model term df1 df2 F.ratio p.value nitrogen 3 14.94 8.103 0.0019结果:varieties = Golden.rain时,施肥处理的主效应是显著的;同时其他两种的品种在施肥处理上的主效应是显著的。
那么我的主效应显著以后就需要做多重比较了。
> emmeans(oats.lmer1,pairwise~nitrogen|varieties,adjust="none")$emmeansvarieties = Golden.rain: nitrogen emmean SE df lower.CL upper.CL 0.0cwt 80.2 8.80 14.9 61.5 99.0 0.2cwt 98.2 8.76 14.8 79.5 116.8 0.4cwt 113.1 9.27 16.6 93.5 132.7 0.6cwt 126.2 9.19 16.4 106.7 145.6varieties = Marvellous: nitrogen emmean SE df lower.CL upper.CL 0.0cwt 86.4 8.79 15.0 67.7 105.1 0.2cwt 108.6 8.76 14.8 89.9 127.3 0.4cwt 117.1 9.02 16.1 98.0 136.2 0.6cwt 125.5 9.16 16.4 106.1 144.9varieties = Victory: nitrogen emmean SE df lower.CL upper.CL 0.0cwt 71.4 9.12 16.6 52.1 90.7 0.2cwt 90.5 9.26 17.0 71.0 110.0 0.4cwt 109.7 10.03 14.0 88.1 131.2 0.6cwt 118.2 10.55 13.0 95.4 141.0Degrees-of-freedom method: kenward-roger Confidence level used: 0.95 $contrastsvarieties = Golden.rain: contrast estimate SE df t.ratio p.value 0.0cwt - 0.2cwt -17.94 8.57 40.3 -2.093 0.0427 0.0cwt - 0.4cwt -32.88 9.21 43.9 -3.571 0.0009 0.0cwt - 0.6cwt -45.96 9.18 43.4 -5.007 <.0001> 0.2cwt - 0.4cwt -14.94 9.18 44.0 -1.628 0.1107 0.2cwt - 0.6cwt -28.01 9.17 42.4 -3.055 0.0039 0.4cwt - 0.6cwt -13.07 9.82 45.7 -1.331 0.1898 varieties = Marvellous: contrast estimate SE df t.ratio p.value 0.0cwt - 0.2cwt -22.17 8.65 42.2 -2.563 0.0140 0.0cwt - 0.4cwt -30.69 8.97 46.8 -3.420 0.0013 0.0cwt - 0.6cwt -39.11 9.14 44.8 -4.279 0.0001 0.2cwt - 0.4cwt -8.52 8.98 46.2 -0.949 0.3476 0.2cwt - 0.6cwt -16.94 9.09 45.8 -1.864 0.0687 0.4cwt - 0.6cwt -8.43 9.55 51.1 -0.882 0.3817 varieties = Victory: contrast estimate SE df t.ratio p.value 0.0cwt - 0.2cwt -19.10 9.49 54.5 -2.012 0.0492 0.0cwt - 0.4cwt -38.24 10.35 16.4 -3.693 0.0019 0.0cwt - 0.6cwt -46.77 10.74 14.9 -4.354 0.0006 0.2cwt - 0.4cwt -19.15 10.75 12.2 -1.780 0.0998 0.2cwt - 0.6cwt -27.67 10.98 13.1 -2.521 0.0255 0.4cwt - 0.6cwt -8.52 11.88 12.7 -0.717 0.4861 Degrees-of-freedom method: kenward-roger式子:pairwise~nitrogen|varieties,其中:竖线前是预测变量,竖线后调节变量,如果实在不理解的话,你就理解是一个分式,竖线前是分子,竖线后是分母。当你要比较分子间的差异时,是不是要保证分母是相同的,也就是说,我要比较不同的施肥处理在燕麦产量上的差异时,我要保证的是施肥处理应作用在同一个品种上才可以进行比较?(在相同的品种上,不同施肥处理的方式间的差异是否显著)
结果还是看contrasts部分。例如varieties(品种) = Golden.rain上,施肥处理为0.2cwt - 0.4cwt和0.4cwt - 0.6cwt之间是不存在显著性(p值小于0.05)差异的,其他同理。
反之,如果你的简单主效应分析的是:不同施肥处理上品种的主效应,若显著后,需要做多重比较(在相同的施肥处理方式上,不同品种间的差异是否显著)
【注:多重比较有很多比较的方法】如下:
> p.adjust.methods[1] "holm" "hochberg" "hommel" "bonferroni" "BH" "BY" "fdr" [8] "none"拓展
一:混合线性模型不仅可以使用lme4包中lmer()函数,也可以使用nlme包中lme()函数,这两个包语法都差不多,相对来说lme4这个包的运算速度会快一点。还有一个ASReml-R包中asreml()函数。
二:如果因变量是分类变量,则需要用广义线性模型。
glmer(data = , formula = , family = ,...)其中,data就是数据集,formula和上面是一样的,family和逻辑回归中是一样的。
代码
#释放内存rm(list=ls())gc()#加载数据:采用R的MASS包中的oats数据集library(MASS)data(oats)names(oats) = c('blocks', 'varieties', 'nitrogen', 'yields')str(oats)#虚拟变量编码方式library(lme4)contrasts(oats$varieties)contr.treatment(3)contr.sum(3)contr.poly(3)contrasts(oats$nitrogen)table(oats$varieties)table(oats$nitrogen)DV function(n){ m 1) for(i in 1:n){ for(j in 1:n-1){ if(i-1 == j) m[i,j] 1-( else m[i,j] 1/n) } } return(m)} DV(2)#重置品种和施肥处理的虚拟变量编码contrasts(oats$varieties) 3)contrasts(oats$nitrogen) 4)#添加项目变量item for(i in 1:6){ item 1:}oats #创建虚拟变量mm View(mm)data View(data)str(oats)str(data)#建模oats.lmer_full = lmer(yields ~ varieties*nitrogen + (1+varieties*nitrogen | blocks)+ (1+varieties*nitrogen | item), data = oats)#模型优化oats.lmer = lmer(yields ~ varieties*nitrogen + (1+varieties1+varieties2+nitrogen1+nitrogen2+nitrogen3+varieties1:nitrogen1+varieties2:nitrogen1+varieties1:nitrogen2+varieties2:nitrogen2+varieties1:nitrogen3+varieties2:nitrogen3 || blocks)+ (1+varieties1+varieties2+nitrogen1+nitrogen2+nitrogen3+varieties1:nitrogen1+varieties2:nitrogen1+varieties1:nitrogen2+varieties2:nitrogen2+varieties1:nitrogen3+varieties2:nitrogen3 || item), data = data)summary(oats.lmer)isSingular(oats.lmer)summary(rePCA(oats.lmer))VarCorr(oats.lmer)#最终模型oats.lmer1 = lmer(yields ~ varieties*nitrogen + (1| blocks)+ (-1+varieties2:nitrogen2+varieties2:nitrogen3 || item), data = data)summary(oats.lmer1)anova(oats.lmer1)anova(oats.lmer,oats.lmer1)#主效应事后比较library(emmeans)p.adjust.methodsemmeans(oats.lmer1,pairwise~varieties,adjust="none")emmeans(oats.lmer1,pairwise~nitrogen,adjust="none")#简单效应分析:比较不同品种上施肥处理的主效应joint_tests(oats.lmer1,by="varieties")emmeans(oats.lmer1,pairwise~nitrogen|varieties,adjust="none")感谢您的支持