(CNS复现)CLAM——Chapter_01
(CNS复现)CLAM——Chapter_01
CLAM: A Deep-Learning-based Pipeline for Data Efficient and Weakly Supervised Whole-Slide-level Analysis
文章目录
- (CNS复现)CLAM——Chapter_01
-
-
- 前言
- Step-01: imports
- Step-02:初始化配置信息
- Step-03:调试代码
-
- Step-03.01 初始化配置文件
- Step-03.02 初始化WSI
- Step-03.03 分割细节解读
-
- 获取描边和空洞的可视化
-
前言
(CNS复现)CLAM——Chapter_00
(CNS复现)CLAM——Chapter_01
(CNS复现)CLAM——Chapter_02
(CNS复现)CLAM——Chapter_03
这一部分主要是针对数据预处理的了解 create_patches_fp.py 中获取 mask 的方法和具体流程
Step-01: imports
示例:pandas 是基于NumPy 的一种工具,该工具是为了解决数据分析任务而创建的。
# imports
# 这一部分都是基础包,不需要讲解
import os
import numpy as np
import time
import pdb
import cv2
import matplotlib.pyplot as plt
plt.rcParams['axes.unicode_minus'] = False
import pandas as pd
import warnings
# my imports
from wsi_core.batch_process_utils import initialize_df # 这个包的作用是将输入的参数转换为EXCEL,方便记录
from wsi_core.WholeSlideImage import WholeSlideImage # WSI对象,里面的功能到时候一一讲解
from wsi_core.wsi_utils import StitchCoords # 这个是一个可视化工具
# other options
warnings.filterwarnings("ignore")
Step-02:初始化配置信息
关于设置基本参数这一部分的内容暂时不解释,因为涉及到的参数太多,后面会陆续解释
首先我们先看一下官方的参数配置信息:
seg参数列表如下:
seg_level:
用于细分WSI的下采样级别(默认值:-1,使用WSI中最接近64倍下采样的下采样)
sthresh:细分阈值(正整数,默认值:8,使用更高的阈值会导致前景减少和背景检测更多)
mthresh:中值过滤器大小(正数,奇数整数,默认值:7)
use_otsu:使用otsu的方法而不是简单的二进制阈值(默认值:False)
close:在初始阈值之后应用其他形态学闭合(正整数或-1,默认值:4)
轮廓过滤参数列表如下:
a_t:组织的区域过滤器阈值(正整数,相对于0级参考面块大小512 x 512而言要考虑的检测到的前景轮廓的最小大小,例如,值10表示仅检测到的前景轮廓的大小大于10 512 x 0级将处理512个大小的补丁,默认值:100)
这个意思是这样的,用一个大小为512*512的检测框检测,若前景轮廓面积(组织面积)大小大于给定与值,则保留该位置,否则改为值被删除
a_h:孔的面积过滤器阈值(正整数,要避免的前景轮廓中检测到的孔/腔的最小尺寸,相对于级别0的512 x 512尺寸色块,默认值:16)
这个参数的意思是,前景轮廓中混存某些天然组织空洞和制作标本的时候留存的人工空洞,当空洞小于某个阈值的时候认为是天然空洞
max_n_holes:检测轮廓中最大空洞数量(正整数,默认值:10,最大值越大,修补越准确,但会增加计算成本)
细分可视化参数列表如下:
假设一个前景轮廓中有15个天然空洞,每个面积大小不一,如果设置的范围太小的话,就会将几个邻近的空洞合并变成一个大空洞,这样就会导致误差,将天然空洞当作人工空洞,不过阈值越大,算力越大
vis_level:下采样级别以可视化细分结果(默认值:-1,使用WSI中最接近64倍下采样的下采样)
line_thickness:绘制的线条粗细以可视化分割结果(正整数,以级别0处绘制的线条所占据的像素数为单位,默认值:250)
修补参数列表如下:
use_padding:是否填充幻灯片的边框(默认值:True)
contour_fn:轮廓检查功能,决定是否应将补丁视为前景还是背景(“ four_pt”之间的选择-检查围绕补丁中心的小网格中的所有四个点是否都在轮廓内,“ center”-检查补丁的中心在轮廓内,“基本”-检查补丁的左上角是否在轮廓内,默认值:“ four_pt”)
four_pt 有两个,一个是hard 一个是easy
hard 的话要是要四个点全部在轮廓内,
easy的话只需要四个有一个在里面就可以
# 初始化配置信息
# 元路径
save_dir = '/media/yuansh/14THHD/CLAM/Result'
source = '/media/yuansh/14THHD/CLAM/DataSet/LUAD'
# 输出文件路径
patch_save_dir = os.path.join(save_dir, 'patches') # patch文件,以.H5形式保存,其中记录了各个patch 的四个坐标的位置
mask_save_dir = os.path.join(save_dir, 'masks') # mask文件,即去除背景色后的jpg文件,可以显著减少image大小
stitch_save_dir = os.path.join(save_dir, 'stitches') # 可视化文件
directories = {'source': source,
'save_dir': save_dir,
'patch_save_dir': patch_save_dir,
'mask_save_dir': mask_save_dir,
'stitch_save_dir': stitch_save_dir}
# 判断目标文件夹是否存在,如果不存在则创建文件夹
for key, val in directories.items():
print("{} : {}".format(key, val))
if key not in ['source']:
os.makedirs(val, exist_ok=True)
# 设置基本参数
process_list = None # 一个文本文件,里面存放各个数据的处理参数
patch_size = 256 # 每个patch 的大小
step_size = 256 # 每个patch搜索框的步长,当与patch size相同的时候每个patch没有交集
patch_level = 0 # 下采样水平,水平越高,下采样程度越大,数据大小越小
use_default_params = False # 是否使用初始化参数
seg = True # 是否切割图片
save_mask = True # 保存切割后的数据
stitch = True # 是否对输出的patch数据可视化
patch = False # 是否输出patch文件
auto_skip = False # 是否跳过已经处理的数据
# 操作参数
seg_params = {'seg_level': -1, 'sthresh': 8, 'mthresh': 7, 'close': 4, 'use_otsu': False,
'keep_ids': 'none', 'exclude_ids': 'none'}
filter_params = {'a_t': 100, 'a_h': 16, 'max_n_holes': 8}
vis_params = {'vis_level': -1, 'line_thickness': 500}
patch_params = {'use_padding': True, 'contour_fn': 'four_pt'}
parameters = {'seg_params': seg_params,
'filter_params': filter_params,
'patch_params': patch_params,
'vis_params': vis_params}
# 如果有参数表,则输入参数表
if process_list:
process_list = os.path.join(save_dir, process_list)
else:
process_list = None
Step-03:调试代码
这里我们需要参考 WholeSlideImage 这个文件
在主文件create_patches_fp.py中,只有一句主函数 seg_and_patch
因此,我们仅需对这一部分的内容进行调试即可
要获取的 mask 主要涉及如下函数
def segment(WSI_object, seg_params, filter_params):
# Start Seg Timer
start_time = time.time()
# Segment
WSI_object.segmentTissue(**seg_params, filter_params=filter_params)
# Stop Seg Timers
seg_time_elapsed = time.time() - start_time
return WSI_object, seg_time_elapsed
Step-03.01 初始化配置文件
# 加载数据目录,并获取数据处理的参数
# 参数主要分为四个部分:
# 1. 语义分割部分
# 2. 可视化部分
# 3. 过滤的部分
# 4. patch 的部分
# 初始化配置参数完全相同
# 读取数据
# 这一部分的内容比较简单,就是将输入的参数保存并出书成一个excel文件
# initialize_df 这个函数不用管他
# 因为他做的事情,就是记录所有样本的输入的参数,并将其保存为csv文件
slides = sorted(os.listdir(source))
slides = [slide for slide in slides if os.path.isfile(
os.path.join(source, slide))]
if process_list is None:
df = initialize_df(slides, seg_params, filter_params,
vis_params, patch_params)
else:
df = pd.read_csv(process_list)
df = initialize_df(df, seg_params, filter_params,
vis_params, patch_params)
mask = df['process'] == 1
process_stack = df[mask]
total = len(process_stack)
legacy_support = 'a' in df.keys()
if legacy_support:
print('detected legacy segmentation csv file, legacy support enabled')
df = df.assign(**{'a_t': np.full((len(df)), int(filter_params['a_t']), dtype=np.uint32),
'a_h': np.full((len(df)), int(filter_params['a_h']), dtype=np.uint32),
'max_n_holes': np.full((len(df)), int(filter_params['max_n_holes']), dtype=np.uint32),
'line_thickness': np.full((len(df)), int(vis_params['line_thickness']), dtype=np.uint32),
'contour_fn': np.full((len(df)), patch_params['contour_fn'])})
# 记录时间
seg_times = 0.
patch_times = 0.
stitch_times = 0.
df.to_csv(os.path.join(save_dir, 'process_list_autogen.csv'), index=False) # 保存处理参数
# 其中 process=1 代表这个数据需要处理
Step-03.02 初始化WSI
# 初始化WSI对象
i = 50 # 输入索引
idx = process_stack.index[i] # 获取索引
slide = process_stack.loc[idx, 'slide_id'] # 获取slide id
print("\n\nprogress: {:.2f}, {}/{}".format(i/total, i, total))
print('processing {}'.format(slide))
df.loc[idx, 'process'] = 0 # 正在处理第 i 个文件
slide_id, _ = os.path.splitext(slide) # 获取id前缀
# 跳过已经处理过的数据,我这里点False
if auto_skip and os.path.isfile(os.path.join(patch_save_dir, slide_id + '.h5')):
print('{} already exist in destination location, skipped'.format(slide_id))
df.loc[idx, 'status'] = 'already_exist'
# continue
# 初始化WSI对象
# 基本属性包括:
# 1. 样本名
# 2. wsi对象
# 3. level_dim 下采样对应维度
# 4. 可下采样水平
full_path = os.path.join(source, slide)
WSI_object = WholeSlideImage(full_path)
if use_default_params:
current_vis_params = vis_params.copy()
current_filter_params = filter_params.copy()
current_seg_params = seg_params.copy()
current_patch_params = patch_params.copy()
else:
current_vis_params = {}
current_filter_params = {}
current_seg_params = {}
current_patch_params = {}
for key in vis_params.keys():
if legacy_support and key == 'vis_level':
df.loc[idx, key] = -1
current_vis_params.update({key: df.loc[idx, key]})
for key in filter_params.keys():
if legacy_support and key == 'a_t':
old_area = df.loc[idx, 'a']
seg_level = df.loc[idx, 'seg_level']
scale = WSI_object.level_downsamples[seg_level]
adjusted_area = int(
old_area * (scale[0] * scale[1]) / (512 * 512))
current_filter_params.update({key: adjusted_area})
df.loc[idx, key] = adjusted_area
current_filter_params.update({key: df.loc[idx, key]})
for key in seg_params.keys():
if legacy_support and key == 'seg_level':
df.loc[idx, key] = -1
current_seg_params.update({key: df.loc[idx, key]})
for key in patch_params.keys():
current_patch_params.update({key: df.loc[idx, key]})
if current_vis_params['vis_level'] < 0:
if len(WSI_object.level_dim) == 1:
current_vis_params['vis_level'] = 0
else:
wsi = WSI_object.getOpenSlide()
best_level = wsi.get_best_level_for_downsample(64)
current_vis_params['vis_level'] = best_level
if current_seg_params['seg_level'] < 0:
if len(WSI_object.level_dim) == 1:
current_seg_params['seg_level'] = 0
else:
wsi = WSI_object.getOpenSlide()
best_level = wsi.get_best_level_for_downsample(64)
current_seg_params['seg_level'] = best_level
keep_ids = str(current_seg_params['keep_ids'])
if keep_ids != 'none' and len(keep_ids) > 0:
str_ids = current_seg_params['keep_ids']
current_seg_params['keep_ids'] = np.array(
str_ids.split(',')).astype(int)
else:
current_seg_params['keep_ids'] = []
exclude_ids = str(current_seg_params['exclude_ids'])
if exclude_ids != 'none' and len(exclude_ids) > 0:
str_ids = current_seg_params['exclude_ids']
current_seg_params['exclude_ids'] = np.array(
str_ids.split(',')).astype(int)
else:
current_seg_params['exclude_ids'] = []
w, h = WSI_object.level_dim[current_seg_params['seg_level']]
if w * h > 1e8:
print(
'level_dim {} x {} is likely too large for successful segmentation, aborting'.format(w, h))
df.loc[idx, 'status'] = 'failed_seg'
# continue
df.loc[idx, 'vis_level'] = current_vis_params['vis_level']
df.loc[idx, 'seg_level'] = current_seg_params['seg_level']
查看输入的参数:
# IN
WSI_object.level_dim
WSI_object.level_downsamples
use_default_params
legacy_support
current_seg_params
current_filter_params
((101591, 46431), (25397, 11607), (6349, 2901), (3174, 1450))
[(1.0, 1.0),
(4.000118124187896, 4.000258464719566),
(16.001102535832416, 16.00517063081696),
(32.007246376811594, 32.02137931034483)]
False
False
{'seg_level': 3,
'sthresh': 8,
'mthresh': 7,
'close': 4,
'use_otsu': False,
'keep_ids': [],
'exclude_ids': []}
{'a_t': 100.0, 'a_h': 16.0, 'max_n_holes': 8}
先运行具体函数,并查看输出结果
WSI_object, seg_time_elapsed = segment(WSI_object, current_seg_params, current_filter_params)
mask = WSI_object.visWSI(**current_vis_params)
Step-03.03 分割细节解读
这里主要涉及到WSI类方法segmentTissue
# 现在进行类方法调试
# 初始化输入数据
# 若输入参数在字典中,则使用字典中的参数,否则使用默认参数
seg_level=2
sthresh=8
sthresh_up = 255
mthresh=7
close = 4
use_otsu=False
filter_params={'a_t': 100.0, 'a_h': 16.0, 'max_n_holes': 8}
ref_patch_size=512
exclude_ids=[]
keep_ids=[]
### 请务必注意初始化的参数必须与 current_vis_params 一致
请务必注意初始化的参数必须与 current_vis_params
current_seg_params
current_filter_params
一致
{'vis_level': 3, 'line_thickness': 500}
{'seg_level': 3,
'sthresh': 8,
'mthresh': 7,
'close': 4,
'use_otsu': False,
'keep_ids': [],
'exclude_ids': []}
{'a_t': 100.0, 'a_h': 16.0, 'max_n_holes': 8}
注意到,类方法中有 _filter_contours 方法,现在暂时不管
已经将类方法中的self改为WSI_object
# 读取数据
img = np.array(WSI_object.wsi.read_region((0,0), seg_level, WSI_object.level_dim[seg_level]))
# 将数据转化为HSV
img_hsv = cv2.cvtColor(img, cv2.COLOR_RGB2HSV) # Convert to HSV space
# 中值波滤
img_med = cv2.medianBlur(img_hsv[:,:,1], mthresh) # Apply median blurring
# 二值化
if use_otsu:
_, img_otsu1 = cv2.threshold(img_med, 0, sthresh_up, cv2.THRESH_OTSU+cv2.THRESH_BINARY)
else:
_, img_otsu1 = cv2.threshold(img_med, sthresh, sthresh_up, cv2.THRESH_BINARY)
# 这又是一个波滤操作,默认的话是一个核大小为4的波滤器
if close > 0:
kernel = np.ones((close, close), np.uint8)
img_otsu2 = cv2.morphologyEx(img_otsu1, cv2.MORPH_CLOSE, kernel)
# 对原始数据进行下采样尺度转换
img_otsu = img_otsu2
scale = WSI_object.level_downsamples[seg_level]
scaled_ref_patch_area = int(ref_patch_size**2 / (scale[0] * scale[1]))
filter_params = filter_params.copy()
filter_params['a_t'] = filter_params['a_t'] * scaled_ref_patch_area
filter_params['a_h'] = filter_params['a_h'] * scaled_ref_patch_area
这里做一个总结,
首先为每一个样本初始化一个数据处理参数
然后读取数据,如果给定的WSI有多个level,那么提取最大的level,level越大对应的数值矩阵越小
# 可视化一下结果看一下他都做了什么操作
fig = plt.figure(figsize=(24,24))
from mpl_toolkits.axes_grid import ImageGrid
grid = ImageGrid(fig, 111, # 与 subplot(111) 的作用一致
nrows_ncols=(5, 1), # image 排版
axes_pad=0.5, # 子图间距
)
grid[0].imshow(img)
grid[0].set_title("img:Raw image", fontsize=15)
grid[0].axis("off")
grid[1].imshow(img_hsv)
grid[1].set_title("img_hsv:HSV", fontsize=15)
grid[1].axis("off")
grid[2].imshow(img_med)
grid[2].set_title("img_med:mid filter", fontsize=15)
grid[2].axis("off")
grid[3].imshow(img_otsu1)
grid[3].set_title("img_otsu1:Thresholding binary", fontsize=15)
grid[3].axis("off");
grid[4].imshow(img_otsu1)
grid[4].set_title("img_otsu2:Thresholding binary", fontsize=15)
grid[4].axis("off");
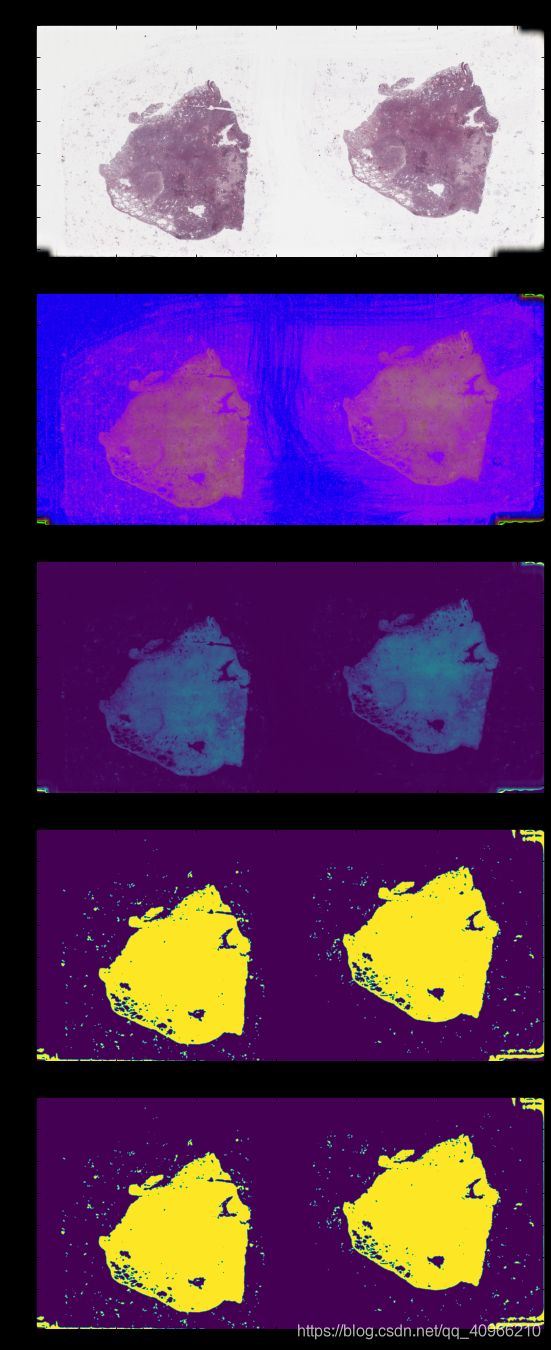
接下来进行轮廓过滤,需要使用 _filter_contours
先对函数进行调试
# cv2.findContours()函数接受的参数为二值图,即黑白的(不是灰度图),所以读取的图像要先转成灰度的,再转成二值图
# opencv2返回两个值:contours:hierarchy。注:opencv3会返回三个值,分别是img, countours, hierarchy
# 参数:
# 第一个参数是寻找轮廓的图像;
# 第二个参数表示轮廓的检索模式,有四种(本文介绍的都是新的cv2接口):
# cv2.RETR_EXTERNAL 表示只检测外轮廓
# cv2.RETR_LIST 检测的轮廓不建立等级关系
# cv2.RETR_CCOMP 建立两个等级的轮廓,上面的一层为外边界,里面的一层为内孔的边界信息。如果内孔内还有一个连通物体,这个物体的边界也在顶层。
# cv2.RETR_TREE 建立一个等级树结构的轮廓。
#
# 第三个参数method为轮廓的近似办法
# cv2.CHAIN_APPROX_NONE 存储所有的轮廓点,相邻的两个点的像素位置差不超过1,即max(abs(x1-x2),abs(y2-y1))==1
# cv2.CHAIN_APPROX_SIMPLE 压缩水平方向,垂直方向,对角线方向的元素,只保留该方向的终点坐标,例如一个矩形轮廓只需4个点来保存轮廓信息
# cv2.CHAIN_APPROX_TC89_L1,CV_CHAIN_APPROX_TC89_KCOS 使用teh-Chinl chain 近似算法
# cv2.findContours()函数返回两个值,一个是轮廓本身,还有一个是每条轮廓对应的属性。
# hierarchy(轮廓属性)分别表示后一个轮廓、前一个轮廓、父轮廓、内嵌轮廓的索引编号,如果没有对应项,则该值为负数。
contours, hierarchy = cv2.findContours(img_otsu, cv2.RETR_CCOMP, cv2.CHAIN_APPROX_NONE) # Find contours
hierarchy = np.squeeze(hierarchy, axis=(0,))[:, 2:] #只需要提取父轮廓和子轮廓即可
# 轮廓的可视化
# cv2.drawContours()函数
# cv2.drawContours(image, contours, contourIdx, color[, thickness[, lineType[, hierarchy[, maxLevel[, offset ]]]]])
#
#
# 第一个参数是指明在哪幅图像上绘制轮廓;
# 第二个参数是轮廓本身,在Python中是一个list。
# 第三个参数指定绘制轮廓list中的哪条轮廓,如果是-1,则绘制其中的所有轮廓。后面的参数很简单。其中thickness表明轮廓线的宽度,如果是-1(cv2.FILLED),则为填充模式。绘制参数将在以后独立详细介绍。
# (0,255,0)轮廓颜色
plt.figure(figsize= (12,12))
plt.imshow(cv2.drawContours(img,contours,-1,(0,255,0),10,lineType=cv2.LINE_8));

大体上的轮廓已经描边出来了,现在进行组织区域和空洞过滤, 这里和上面输出的mask还是有区别的
# 过滤轮廓
# 组织内部轮廓必有父轮廓,因此没有副父轮廓的一律全部过滤
# 计算轮廓面积,轮廓面积减掉子轮廓的面积大于给定面积(a_t),则去掉该轮廓
def _filter_contours(contours, hierarchy, filter_params):
filtered = []
# 提取候选前景轮廓的索引ID,前景轮廓就是没有父轮廓的轮廓 (parent == -1)
hierarchy_1 = np.flatnonzero(hierarchy[:,1] == -1)
all_holes = []
# 根据上面介绍的方法,一次循环每一个候选前景轮廓,最后筛选出最终轮廓
# loop through foreground contour indices
for cont_idx in hierarchy_1:
# 实际的image轮廓必须是候选前景轮廓(这个应该很好理解吧)
cont = contours[cont_idx]
# 把候选前景轮廓中的洞提取出来 (children of parent contour)
holes = np.flatnonzero(hierarchy[:, 1] == cont_idx)
# 计算候选前景轮廓的面积,包括洞 (includes holes)
a = cv2.contourArea(cont)
# 单独计算候选前景轮廓中的洞的面积
hole_areas = [cv2.contourArea(contours[hole_idx]) for hole_idx in holes]
# 候选前景轮廓的面积减去洞的面积
a = a - np.array(hole_areas).sum()
# 如果候选前景轮廓减去洞的面积为0 ,那么这个不算前景轮廓
if a == 0: continue
# 如果候选前景轮廓的面积大于给定面积,则该候选前景轮廓为实际前景轮廓
# 只有实际前景轮廓中的洞才有资格是候选的后天人工误差洞
if tuple((filter_params['a_t'],)) < tuple((a,)):
filtered.append(cont_idx)
all_holes.append(holes)
foreground_contours = [contours[cont_idx] for cont_idx in filtered]
hole_contours = []
# 提取候选的后天人工误差洞
for hole_ids in all_holes:
unfiltered_holes = [contours[idx] for idx in hole_ids ]
# 计算候选的后天人工误差洞的面积
unfilered_holes = sorted(unfiltered_holes, key=cv2.contourArea, reverse=True)
# 注意候选的后天人工误差洞的个数是有限的,并且面积远大于组织天然洞,因此对所有的候选的后天人工误差洞的面积进行排序,筛选出最大的几个
unfilered_holes = unfilered_holes[:filter_params['max_n_holes']]
filtered_holes = []
# 如果候选的后天人工误差洞的面积大于给定阈值,则这些洞为真实的误差洞
for hole in unfilered_holes:
if cv2.contourArea(hole) > filter_params['a_h']:
filtered_holes.append(hole)
hole_contours.append(filtered_holes)
return foreground_contours, hole_contours
if filter_params: foreground_contours, hole_contours = _filter_contours(contours, hierarchy, filter_params)
WSI_object.contours_tissue = WSI_object.scaleContourDim(foreground_contours, scale)
WSI_object.holes_tissue = WSI_object.scaleHolesDim(hole_contours, scale)
#exclude_ids = [0,7,9]
if len(keep_ids) > 0:
contour_ids = set(keep_ids) - set(exclude_ids)
else:
contour_ids = set(np.arange(len(WSI_object.contours_tissue))) - set(exclude_ids)
WSI_object.contours_tissue = [WSI_object.contours_tissue[i] for i in contour_ids]
WSI_object.holes_tissue = [WSI_object.holes_tissue[i] for i in contour_ids]
参数初始化原则与上面的一样
import math
from PIL import Image
top_left = None
view_slide_only = False
seg_display = True
number_contours = False
color = (0, 255, 0) # 描边颜色
hole_color = (0, 0, 255) # 空洞颜色
custom_downsample = 1
max_size = None
line_thickness = 250 # 描边粗细
获取描边和空洞的可视化
这部分的代码几乎没的讲,自己看就行
downsample = WSI_object.level_downsamples[vis_level]
scale = [1/downsample[0], 1/downsample[1]]
if top_left is not None and bot_right is not None:
top_left = tuple(top_left)
bot_right = tuple(bot_right)
w, h = tuple((np.array(bot_right) * scale).astype(int) -
(np.array(top_left) * scale).astype(int))
region_size = (w, h)
else:
top_left = (0, 0)
region_size = WSI_object.level_dim[vis_level]
img = np.array(WSI_object.wsi.read_region(
top_left, vis_level, region_size).convert("RGB"))
if not view_slide_only:
offset = tuple(-(np.array(top_left) * scale).astype(int))
line_thickness = int(line_thickness * math.sqrt(scale[0] * scale[1]))
if WSI_object.contours_tissue is not None and seg_display:
if not number_contours:
img_number_contours = cv2.drawContours(img, WSI_object.scaleContourDim(WSI_object.contours_tissue, scale),
-1, color, line_thickness, lineType=cv2.LINE_8, offset=offset)
else: # add numbering to each contour
for idx, cont in enumerate(WSI_object.contours_tissue):
contour = np.array(WSI_object.scaleContourDim(cont, scale))
M = cv2.moments(contour)
cX = int(M["m10"] / (M["m00"] + 1e-9))
cY = int(M["m01"] / (M["m00"] + 1e-9))
# draw the contour and put text next to center
img_contour = cv2.drawContours(
img, [contour], -1, color, line_thickness, lineType=cv2.LINE_8, offset=offset)
cv2.putText(img, "{}".format(idx), (cX, cY),
cv2.FONT_HERSHEY_SIMPLEX, 2, (255, 0, 0), 10)
for holes in WSI_object.holes_tissue:
img_holes_tissue = cv2.drawContours(img, WSI_object.scaleContourDim(holes, scale),
-1, hole_color, line_thickness, lineType=cv2.LINE_8)
if WSI_object.contours_tumor is not None and annot_display:
img_contours_tumor = cv2.drawContours(img, WSI_object.scaleContourDim(WSI_object.contours_tumor, scale),
-1, annot_color, line_thickness, lineType=cv2.LINE_8, offset=offset)
img = Image.fromarray(img)
w, h = img.size
if custom_downsample > 1:
img = img.resize((int(w/custom_downsample), int(h/custom_downsample)))
if max_size is not None and (w > max_size or h > max_size):
resizeFactor = max_size/w if w > h else max_size/h
img = img.resize((int(w*resizeFactor), int(h*resizeFactor)))
plt.figure(figsize= (12,12))
plt.imshow(img);
如果我的博客您经过深度思考后仍然看不懂,可以根据以下方式联系我:
Best Regards,
Yuan.SH
---------------------------------------
School of Basic Medical Sciences,
Fujian Medical University,
Fuzhou, Fujian, China.
please contact with me via the following ways:
(a) e-mail :yuansh3354@163.com
