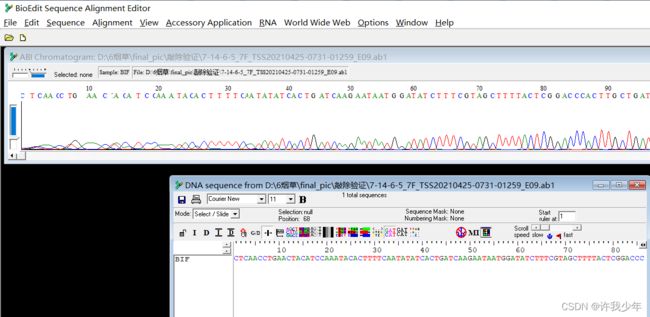
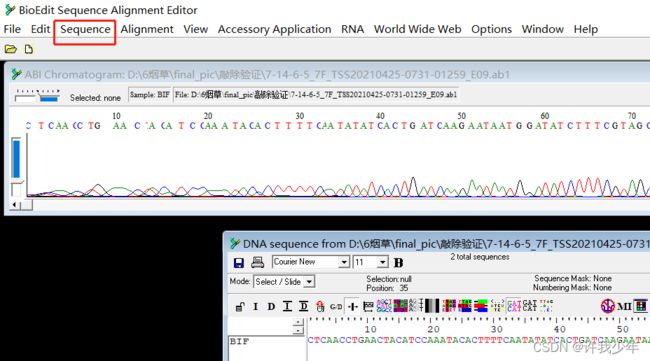
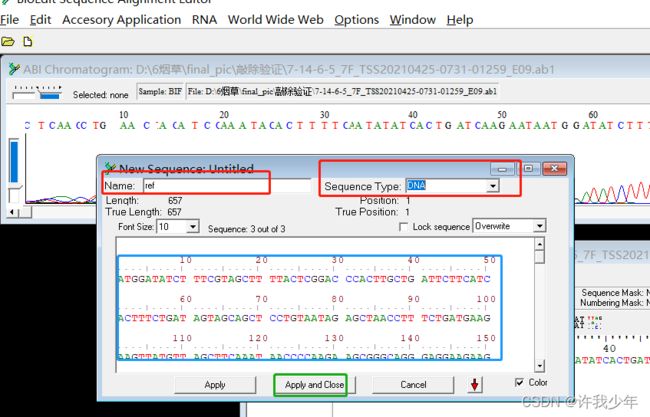
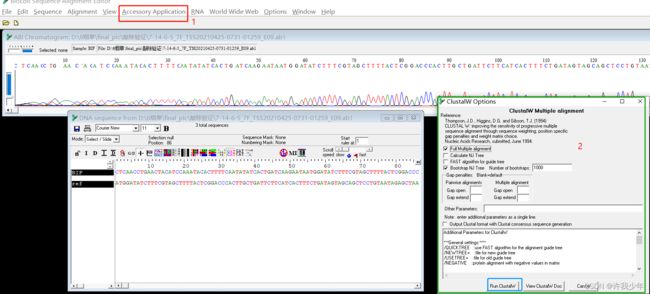
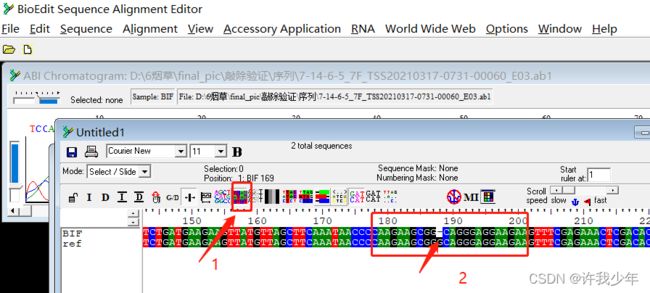
- 在生信分析中,处理vcf 比较好用的python包推荐
在生物信息学分析中,处理VCF(VariantCallFormat)文件的Python包有很多,以下是一些常用且好用的Python包,适合不同的分析需求:PyVCF(推荐)简介:PyVCF是一个专门为解析和操作VCF文件设计的Python库,支持读取、过滤和修改VCF文件。优点:简单易用,API直观。支持VCF4.0及以上版本。可以轻松访问变体的信息(如染色体、位置、参考碱基、变异碱基等)。安装:
- 在ARM46+KylinOS下安装配置Docker的详细步骤
Q_Daniooi
docker容器运维
目录一、安装前准备(一)环境检查(二)依赖准备二、Docker安装步骤(一)添加Docker官方源(以Debian分支银河麒麟为例,RPM系类似调整)(二)安装Docker引擎(三)启动与基础配置三、Docker优化配置(可选但推荐)(一)镜像加速(二)存储驱动优化四、注意事项(一)系统兼容性(二)网络与镜像源(三)权限与安全(四)ARM架构特殊点五、经常遇见的问题及解决方法六、学习经验分享一、前
- 微服务架构下的自动化测试策略调优经验分享
微服务架构下,自动化测试策略需针对分布式特性、服务自治性和高耦合风险进行针对性调整的关键调整方向及实施方法:一、测试策略重构:分层与契约驱动1.测试金字塔升级为钻石模型调整逻辑:传统金字塔中UI测试占比过高,而微服务需强化契约测试与组件测试,形成“钻石形”结构(契约测试占比20%-30%)。实施要点:契约测试层:通过消费者驱动契约(CDC)验证服务间API兼容性,使用Pact框架自动生成测试用例,
- Python综合应用学生管理系统
主要是复习使用,希望大佬提意见整体结构与核心数据结构importosimportsysstudents=[]上述代码引入了os和sys模块,os模块用于处理文件和目录相关操作,sys模块提供了对Python解释器相关变量和函数的访问。students列表作为核心数据结构,用于存储所有学生的信息,后续对学生信息的增删改查操作都围绕它展开。菜单功能实现defmenu():"""显示程序菜单"""pri
- 云原生信息提取系统:容器化流程与CI_CD集成实践
一、问题引出:自动化信息获取为何难以工程化?在实际开发中,我们经常需要对互联网页面进行结构解析与内容提取,但这些任务常常陷入以下困境:本地测试没问题,一旦部署到线上环境便频繁出错;环境配置不一致导致执行失败;内容接口更新频繁,人工维护成本高;无法做到自动更新与持续运行;对接口访问策略缺乏灵活适配手段。这说明,仅靠“能运行的脚本”远远不够,信息提取任务也需要标准化的开发、测试与交付机制。二、真实挑战
- 深度解析:SUSE Harvester私有云平台建设指南
关键词:SUSEHarvester,私有云,HCI,超融合,Kubernetes,KubeVirt,Longhorn,云原生,虚拟化,容器目录导航一、初识SUSEHarvester-私有云的新选择二、核心架构解析-揭开HCI的神秘面纱三、部署实战-从零到一搭建你的私有云四、存储与网络配置-数据的安全港湾五、虚拟机管理-让资源调度更智能六、监控与运维-保驾护航的守护者七、最佳实践-踩坑经验分享八、总
- java组件化设计_构建之路—谈谈组件化后端构建和实现
前言这一篇文章,准备了很久,构思了很久,草稿了很久。从个人编程至今,历经了C,C++,Java,到现如今的NodeJS。也后端到前端,再回到后端。更从学校里的学生信息管理系统到大型商业系统构建,是的,我曾一直以为编程也就是如此了,由瀑布模型,敏捷开发,设计模式等等组成的软件工程大致就是如此了。相信可能很多人也会有和我类似的想法,是否也都曾迷茫过?幸运的是,伴随着对前端的接触和深入,云雾散开。前端组
- 【2025B卷首发】华为OD机试真题+全流程解析+备考攻略+经验分享+Java最佳实现
专栏导读本专栏收录于《华为OD机试(JAVA)真题(E卷+D卷+A卷+B卷+C卷)》。刷的越多,抽中的概率越大,私信哪吒,备注华为OD,加入华为OD刷题交流群,每一题都有详细的答题思路、详细的代码注释、3个测试用例、为什么这道题采用XX算法、XX算法的适用场景,发现新题目,随时更新,全天CSDN在线答疑。2025年5月12日,华为官方已经将华为OD机试(A卷)切换为B卷。目前正在考的是B卷,按照华
- LeetCode 刷题:数据结构与算法的实战经验分享
LeetCode刷题:数据结构与算法的实战经验分享关键词:LeetCode、数据结构、算法、刷题经验、实战摘要:本文将围绕LeetCode刷题展开,深入探讨数据结构与算法在实际刷题过程中的应用。通过分享实战经验,帮助读者更好地理解和掌握数据结构与算法知识,提升解题能力。文章将从背景介绍入手,阐述刷题的目的和意义,接着详细解释核心概念,分析它们之间的关系,然后介绍核心算法原理和具体操作步骤,结合数学
- 深入解读 Qwen3 技术报告(一):引言
小爷毛毛(卓寿杰)
大模型AIGC深度学习基础/原理人工智能自然语言处理python语言模型深度学习
重磅推荐专栏:《大模型AIGC》《课程大纲》《知识星球》本专栏致力于探索和讨论当今最前沿的技术趋势和应用领域,包括但不限于ChatGPT和StableDiffusion等。我们将深入研究大型模型的开发和应用,以及与之相关的人工智能生成内容(AIGC)技术。通过深入的技术解析和实践经验分享,旨在帮助读者更好地理解和应用这些领域的最新进展1.引言:迎接大型语言模型的新纪元我们正处在一个由人工智能(AI
- 大一新生第一次参加蓝桥杯(C/C++组),只学C够吗?怎么备赛?个人经验分享
老虎0627
蓝桥杯
个人感受(唠叨唠叨)我是2023级的物联网工程专业的一名大一新生,在大一的下半学期有幸通过校赛,参加了第十五届蓝桥杯软件赛,其实我自己都没想到大一就能参加蓝桥杯,因为当时只会C语言,也很迷茫,到底该怎么备赛?剩的时间比较少到底要不要学习C++。到底要不要学C++?我在蓝桥杯正式比赛前特别纠结要不要学c++,因为当时省的时间比较少,而且会有担心学c++的一些语法会不会把它跟c语言搞混,到时候在考场忘
- 十分钟聊明白DDD领域驱动设计
roykingw
javajava架构DDD领域驱动
文章目录一、什么是领域?二、领域如何驱动设计?三、如何发挥DDD的价值最后十分钟聊明白DDD领域驱动设计--楼兰关于DDD,大部分朋友应该都听说过。全称DomainDrivenDesign,翻译过来就是领域驱动设计。这个神秘的架构思想,虽然远没有SpringBoot这类框架这么名声在外,但是却经常时不时冒出来,牵动一下大家的神经。美团、阿里每年的技术年会都会有关于DDD的经验分享,而另一方面,又有
- 【经验分享】分布式爬虫的优势与劣势分析
电商数据girl
跨境电商API接口电商项目API接口测试电商ERP项目接口经验分享分布式爬虫java数据库大数据python
分布式爬虫通过多节点协同工作实现数据采集,其设计初衷是解决单节点爬虫在大规模数据抓取场景中的性能瓶颈,但同时也因架构复杂度带来了新的挑战。以下从技术特性、应用场景适配性两个维度,系统分析其优势与劣势:一、分布式爬虫的核心优势高效突破大规模数据采集瓶颈并行处理能力:通过将任务拆分到多个节点并行执行,大幅提升数据抓取效率。例如,采集100万条电商商品数据时,单节点爬虫可能需要数天,而由10个节点组成的
- 华为OD机试真题——版本管理(2025B卷:100分)Java/python/JavaScript/C++最佳实现
纪元A梦
华为OD华为odjavapythonjavascriptc++
2025B卷100分题型本专栏内全部题目均提供Java、python、JavaScript、C++等多种语言的最佳实现方式;并且每种语言均涵盖详细的问题分析、解题思路、代码实现、代码详解、3个测试用例以及综合分析;本文收录于专栏:《2025华为OD真题目录+全流程解析+备考攻略+经验分享》华为OD机试真题《版本管理》:文章快捷目录题目描述及说明JavapythonJavaScriptC++题目名称
- Python指南:必备技巧与经验分享
master_chenchengg
pythonpython办公效率python开发IT
Python指南:必备技巧与经验分享一开场白:与Python共舞欢迎词:向Python爱好者们问好Python的魅力:为什么Python如此受欢迎个人经历:分享自己与Python的不解之缘二数据准备:磨刀不误砍柴工数据清洗:如何让数据焕然一新缺失值处理:填补或删除缺失数据的策略异常值检测:识别并处理异常值的方法数据转换:如何调整数据类型和格式类型转换:转换数据类型以适应需求标准化:使数据在同一尺度
- 学生上机管理系统设计与实现
AR新视野
本文还有配套的精品资源,点击获取简介:《学生上机管理系统》是一款专门用于教育领域的管理软件,通过VB开发实现学生和教师的信息化管理。系统包括学生管理模块和教师管理模块,提供详细的学生信息录入、查询、修改功能,成绩统计与展示,以及课程安排、监控和上机预约等功能。此外,系统支持作业提交和批改,以及基于角色的用户权限管理,确保信息安全性。该系统利用数据库技术和人机交互界面,旨在提高教学质量和管理效率。1
- 浅谈 Vue2 的 Mixin 混入和 Vue3 的 Hooks(组合式 API)
一个水瓶座程序猿.
Vue.js系列文章Vuevue.jsjavascriptecmascript
嘿,各位前端小伙伴!今天咱来好好唠唠Vue2里的Mixin混入和Vue3的Hooks(组合式API),这里面的门道可不少,我把自己的经验分享出来,希望能帮大家避避坑。一、Vue2的Mixin混入1.啥是Mixin混入Mixin混入就像是一个魔法口袋,你可以把一些通用的代码逻辑装进去,然后在多个组件里使用。简单来说,它就是一种代码复用的方式。比如说,你有多个组件都需要处理用户登录状态,那你就可以把这
- 高校招生服务平台小程序的设计与实现
一、选题简介、意义与背景(包括选题的理论价值和实践意义)选题简介:本课题旨针对高校招生数据错综复杂难以管理等问题,运用计算机技术实现一个高校招生服务平台,提供给专业且全面的招生服务,帮助用户快速的找到自己喜欢的专业以及提高招生效率。平台实现两类用户角色,分别是用户跟管理员角色。其中,用户将实现用户的登录注册、首页、招生信息、校园漫游、志愿填报等功能,管理员将实现招生信息管理、用户管理、缴费信息管理
- 40 岁想学中医怎么开始?过来人的经验分享 问止精一书院
2501_92067291
问止中医
零基础学中医学中医如何入门免费学中医!问止精一书院链接:https://tool.nineya.com/qrcode/1iv54b4ts不少人到了40岁,对中医产生浓厚兴趣,却不知该如何起步。作为一名从40岁开始学中医的过来人,我想分享一些实用经验,尤其推荐以问止中医的免费课程作为入门跳板。40岁学中医,最大的顾虑往往是“零基础怕跟不上”。问止中医的免费报名课程恰好解决了这个痛点,课程专为中医小白
- 新品|暴雨信创服务器震撼亮相2025 AI算力产业峰会
BAOYUCompany
人工智能服务器运维
4月9日,被誉为“中国AI算力风向标”的2025AI算力产业峰会在深圳会展中心盛大启幕。作为中国领先的服务器解决方案供应商,暴雨携信创新品亮相峰会,与行业伙伴展开深度交流与经验分享,旨在携手构建AI时代算力产业新范式,为数字未来的蓬勃发展贡献磅礴力量。步入2025年,AIGC技术呈爆发式增长,算力需求随之迎来深刻变革。在此关键节点,暴雨凭借其在软硬件协同研发领域长期深耕积累的雄厚实力,抢滩布局,率
- 大厂数分面试题
临近假期,又是一个找实习的时候,给大家分享一下最近找实习的一些面经,祝大家都能顺利找到满意的实习~目录面经分享1-游戏公司-乐信圣文-游戏数据分析实习生一面-技术面二面-HR面2-美团销售运营(数据分析方向)3-作业帮数据分析4-美团用户运营5-脉脉数据科学实习生反问环节反问环节很重要。为什么?技术面/业务面面试经验分享工具安利面经分享1-游戏公司-乐信圣文-游戏数据分析实习生一面-技术面1.自我
- C# WinForm 实现学生成绩信息管理系统,连接SQL数据库,完整代码
C#WinForm实现学生成绩信息管理系统《面向对象程序设计》实验功能介绍1、学生信息查询2、学生选课信息3、学生信息修改4、学生成绩录入实验分析遇到的问题解决方法主要问题嗨,我是射手座的程序媛,期待与大家更多的交流与学习,欢迎添加:3512724768《面向对象程序设计》实验一、实验目的1.熟练掌握C#开发,掌握建立Windows应用程序的步骤和方法。2.建立面向对象编程理念。3.熟练掌握控件及
- 自学黑客技术多长时间能达到挖漏洞的水平?
~小羊没烦恼~
网络安全黑客技术黑客网络安全web安全人工智能学习
作为一名白帽黑客,自学黑客技术是一种既刺激又实用的技能。然而,很多初学者都好奇,自学这门技术需要多长时间才能达到挖掘漏洞的水平。本文将从黑客的角度详细探讨这个问题,包括学习路径、实践方法和一些个人经验分享。自学路径概览黑客技术的自学可以分为几个阶段:基础知识学习、工具与技术掌握、实战演练和专业深造。每个阶段的时间长度可以根据个人的学习速度和投入时间的多少而有所不同。1.基础知识学习(1-3个月)初
- 数据结构与算法 第一章 绪论
noruta
408#数据结构与算法数据结构
1.1.数据结构的基本概念数据:对计算机来说,能被计算机程序识别和处理的符号的集合。(比如二进制0和1)数据元素:数据的基本单位,通常作为一个整体进行考虑和处理。(比如一个学生的信息是一个数据元素)数据项:构成数据元素的最小单位。(学生的学号,姓名,班级构成一个学生信息)要根据实际的业务需求来确定什么是数据元素、什么是数据项。数据结构:相互之间存在一种或多种特定关系的数据元素的集合。比如汉字有左右
- DolphinScheduler 6 个高频 SQL 操作技巧
数据库
摘要:ApacheDolphinScheduler系列4-后台SQL经验分享关键词:大数据、数据质量、数据调度整体说明在调研了DolphinScheduler之后,在项目上实际使用了一段时间,有了一些后台SQL实际经验,分享如下。进入DolphinScheduler后台数据库,我这里使用的是MySQL数据库。以任务名称包含“ods_xf_act”的任务为例。一、修改任务组操作UPDATEt_ds_
- 《48小时极速开发:Python+MySQL 学生信息管理系统架构实战揭秘》
Cyber4K
Python项目实践及实战pythonmysql架构
Python项目实践:学生信息管理系统1.项目概述1.1项目背景开发周期:2天(需求分析0.5天+开发1天+测试0.5天)技术栈:Python3.9+MySQL+面向对象编程核心价值:实现学生信息的全生命周期管理采用分层架构设计(表示层/业务层/数据层)数据库驱动的高效数据持久化方案1.2系统架构系统架构调用CRUD操作连接池业务逻辑层命令行界面数据访问层MySQL数据库2.核心模块实现2.1数据
- C++实现学生管理系统
lijiatu10086
C++c++
文章目录实验要求一、实验平台二、代码1.结构体以及相关变量2.相关函数实现过程(1)判断一个学生是否已经存在(2)从文件中读写学生信息(3)增加学生(4)删除学生(5)修改学生(6)查询学生(7)main函数的实现3.整体代码总结实验要求实验要求:将班上同学的信息(编号(001对应第一个,008对应第八个),姓名,性别,年龄,学校,年级,班级,爱好……),使用自己设计的结构体来存储。并在此基础上结
- ASP学生信息管理系统完整实施指南
带虾条酱
本文还有配套的精品资源,点击获取简介:ASP学生信息管理系统,基于ActiveServerPages技术,为学校优化学生信息管理,提高工作效率。系统功能涵盖班级管理、成绩跟踪、权限设置等。各组件如list_fenban.asp、xj_update.asp、xj_add.asp、admin_user.asp、funoperate.asp、list_check_upd.asp、list_check_a
- 生信分析用python还是r_生信分析利器:JupyterLab
weixin_39612726
生信分析用python还是r
生信分析对计算机的开发环境有诸多要求,随之而来的自然就是很多麻烦。不说别的,要兼顾Python和R的问题就有够头疼。一边想着用Python搭流程处理文本和分析结果,另一边还想着用R来做统计分析和画图,而且大多数时候生信分析还得在服务器上完成。Python你用Pycharm,R用Rstudio,一会这儿一会那儿的切来切去,还得设置服务器连接(Pycharm如果不是付费版本,要连服务器还挺麻烦)。完了
- 生信技能16 - 生信分析序列处理常用函数
生信与基因组学
生信分析项目实战技能合集pythonnumpy数据分析
生信分析序列处理常用函数生信分析经常需要对序列进行处理,下面的实现代码可用于个人练习,可以让我们更好地理解序列处理的原理,当然python也有更高效率的包可以实现以下功能。read_seq_file读取序列txt文件函数count_nucletotides计算各核苷酸数量函数dna2rnaDNA序列转RNA序列函数seq_reverseDNA序列转换为互补序列函数count_GC_ratio计算序
- html页面js获取参数值
0624chenhong
html
1.js获取参数值js
function GetQueryString(name)
{
var reg = new RegExp("(^|&)"+ name +"=([^&]*)(&|$)");
var r = windo
- MongoDB 在多线程高并发下的问题
BigCat2013
mongodbDB高并发重复数据
最近项目用到 MongoDB , 主要是一些读取数据及改状态位的操作. 因为是结合了最近流行的 Storm进行大数据的分析处理,并将分析结果插入Vertica数据库,所以在多线程高并发的情境下, 会发现 Vertica 数据库中有部分重复的数据. 这到底是什么原因导致的呢?笔者开始也是一筹莫 展,重复去看 MongoDB 的 API , 终于有了新发现 :
com.mongodb.DB 这个类有
- c++ 用类模版实现链表(c++语言程序设计第四版示例代码)
CrazyMizzz
数据结构C++
#include<iostream>
#include<cassert>
using namespace std;
template<class T>
class Node
{
private:
Node<T> * next;
public:
T data;
- 最近情况
麦田的设计者
感慨考试生活
在五月黄梅天的岁月里,一年两次的软考又要开始了。到目前为止,我已经考了多达三次的软考,最后的结果就是通过了初级考试(程序员)。人啊,就是不满足,考了初级就希望考中级,于是,这学期我就报考了中级,明天就要考试。感觉机会不大,期待奇迹发生吧。这个学期忙于练车,写项目,反正最后是一团糟。后天还要考试科目二。这个星期真的是很艰难的一周,希望能快点度过。
- linux系统中用pkill踢出在线登录用户
被触发
linux
由于linux服务器允许多用户登录,公司很多人知道密码,工作造成一定的障碍所以需要有时踢出指定的用户
1/#who 查出当前有那些终端登录(用 w 命令更详细)
# who
root pts/0 2010-10-28 09:36 (192
- 仿QQ聊天第二版
肆无忌惮_
qq
在第一版之上的改进内容:
第一版链接:
http://479001499.iteye.com/admin/blogs/2100893
用map存起来号码对应的聊天窗口对象,解决私聊的时候所有消息发到一个窗口的问题.
增加ViewInfo类,这个是信息预览的窗口,如果是自己的信息,则可以进行编辑.
信息修改后上传至服务器再告诉所有用户,自己的窗口
- java读取配置文件
知了ing
1,java读取.properties配置文件
InputStream in;
try {
in = test.class.getClassLoader().getResourceAsStream("config/ipnetOracle.properties");//配置文件的路径
Properties p = new Properties()
- __attribute__ 你知多少?
矮蛋蛋
C++gcc
原文地址:
http://www.cnblogs.com/astwish/p/3460618.html
GNU C 的一大特色就是__attribute__ 机制。__attribute__ 可以设置函数属性(Function Attribute )、变量属性(Variable Attribute )和类型属性(Type Attribute )。
__attribute__ 书写特征是:
- jsoup使用笔记
alleni123
java爬虫JSoup
<dependency>
<groupId>org.jsoup</groupId>
<artifactId>jsoup</artifactId>
<version>1.7.3</version>
</dependency>
2014/08/28
今天遇到这种形式,
- JAVA中的集合 Collectio 和Map的简单使用及方法
百合不是茶
listmapset
List ,set ,map的使用方法和区别
java容器类类库的用途是保存对象,并将其分为两个概念:
Collection集合:一个独立的序列,这些序列都服从一条或多条规则;List必须按顺序保存元素 ,set不能重复元素;Queue按照排队规则来确定对象产生的顺序(通常与他们被插入的
- 杀LINUX的JOB进程
bijian1013
linuxunix
今天发现数据库一个JOB一直在执行,都执行了好几个小时还在执行,所以想办法给删除掉
系统环境:
ORACLE 10G
Linux操作系统
操作步骤如下:
第一步.查询出来那个job在运行,找个对应的SID字段
select * from dba_jobs_running--找到job对应的sid
&n
- Spring AOP详解
bijian1013
javaspringAOP
最近项目中遇到了以下几点需求,仔细思考之后,觉得采用AOP来解决。一方面是为了以更加灵活的方式来解决问题,另一方面是借此机会深入学习Spring AOP相关的内容。例如,以下需求不用AOP肯定也能解决,至于是否牵强附会,仁者见仁智者见智。
1.对部分函数的调用进行日志记录,用于观察特定问题在运行过程中的函数调用
- [Gson六]Gson类型适配器(TypeAdapter)
bit1129
Adapter
TypeAdapter的使用动机
Gson在序列化和反序列化时,默认情况下,是按照POJO类的字段属性名和JSON串键进行一一映射匹配,然后把JSON串的键对应的值转换成POJO相同字段对应的值,反之亦然,在这个过程中有一个JSON串Key对应的Value和对象之间如何转换(序列化/反序列化)的问题。
以Date为例,在序列化和反序列化时,Gson默认使用java.
- 【spark八十七】给定Driver Program, 如何判断哪些代码在Driver运行,哪些代码在Worker上执行
bit1129
driver
Driver Program是用户编写的提交给Spark集群执行的application,它包含两部分
作为驱动: Driver与Master、Worker协作完成application进程的启动、DAG划分、计算任务封装、计算任务分发到各个计算节点(Worker)、计算资源的分配等。
计算逻辑本身,当计算任务在Worker执行时,执行计算逻辑完成application的计算任务
- nginx 经验总结
ronin47
nginx 总结
深感nginx的强大,只学了皮毛,把学下的记录。
获取Header 信息,一般是以$http_XX(XX是小写)
获取body,通过接口,再展开,根据K取V
获取uri,以$arg_XX
&n
- 轩辕互动-1.求三个整数中第二大的数2.整型数组的平衡点
bylijinnan
数组
import java.util.ArrayList;
import java.util.Arrays;
import java.util.List;
public class ExoWeb {
public static void main(String[] args) {
ExoWeb ew=new ExoWeb();
System.out.pri
- Netty源码学习-Java-NIO-Reactor
bylijinnan
java多线程netty
Netty里面采用了NIO-based Reactor Pattern
了解这个模式对学习Netty非常有帮助
参考以下两篇文章:
http://jeewanthad.blogspot.com/2013/02/reactor-pattern-explained-part-1.html
http://gee.cs.oswego.edu/dl/cpjslides/nio.pdf
- AOP通俗理解
cngolon
springAOP
1.我所知道的aop 初看aop,上来就是一大堆术语,而且还有个拉风的名字,面向切面编程,都说是OOP的一种有益补充等等。一下子让你不知所措,心想着:怪不得很多人都和 我说aop多难多难。当我看进去以后,我才发现:它就是一些java基础上的朴实无华的应用,包括ioc,包括许许多多这样的名词,都是万变不离其宗而 已。 2.为什么用aop&nb
- cursor variable 实例
ctrain
variable
create or replace procedure proc_test01
as
type emp_row is record(
empno emp.empno%type,
ename emp.ename%type,
job emp.job%type,
mgr emp.mgr%type,
hiberdate emp.hiredate%type,
sal emp.sal%t
- shell报bash: service: command not found解决方法
daizj
linuxshellservicejps
今天在执行一个脚本时,本来是想在脚本中启动hdfs和hive等程序,可以在执行到service hive-server start等启动服务的命令时会报错,最终解决方法记录一下:
脚本报错如下:
./olap_quick_intall.sh: line 57: service: command not found
./olap_quick_intall.sh: line 59
- 40个迹象表明你还是PHP菜鸟
dcj3sjt126com
设计模式PHP正则表达式oop
你是PHP菜鸟,如果你:1. 不会利用如phpDoc 这样的工具来恰当地注释你的代码2. 对优秀的集成开发环境如Zend Studio 或Eclipse PDT 视而不见3. 从未用过任何形式的版本控制系统,如Subclipse4. 不采用某种编码与命名标准 ,以及通用约定,不能在项目开发周期里贯彻落实5. 不使用统一开发方式6. 不转换(或)也不验证某些输入或SQL查询串(译注:参考PHP相关函
- Android逐帧动画的实现
dcj3sjt126com
android
一、代码实现:
private ImageView iv;
private AnimationDrawable ad;
@Override
protected void onCreate(Bundle savedInstanceState)
{
super.onCreate(savedInstanceState);
setContentView(R.layout
- java远程调用linux的命令或者脚本
eksliang
linuxganymed-ssh2
转载请出自出处:
http://eksliang.iteye.com/blog/2105862
Java通过SSH2协议执行远程Shell脚本(ganymed-ssh2-build210.jar)
使用步骤如下:
1.导包
官网下载:
http://www.ganymed.ethz.ch/ssh2/
ma
- adb端口被占用问题
gqdy365
adb
最近重新安装的电脑,配置了新环境,老是出现:
adb server is out of date. killing...
ADB server didn't ACK
* failed to start daemon *
百度了一下,说是端口被占用,我开个eclipse,然后打开cmd,就提示这个,很烦人。
一个比较彻底的解决办法就是修改
- ASP.NET使用FileUpload上传文件
hvt
.netC#hovertreeasp.netwebform
前台代码:
<asp:FileUpload ID="fuKeleyi" runat="server" />
<asp:Button ID="BtnUp" runat="server" onclick="BtnUp_Click" Text="上 传" />
- 代码之谜(四)- 浮点数(从惊讶到思考)
justjavac
浮点数精度代码之谜IEEE
在『代码之谜』系列的前几篇文章中,很多次出现了浮点数。 浮点数在很多编程语言中被称为简单数据类型,其实,浮点数比起那些复杂数据类型(比如字符串)来说, 一点都不简单。
单单是说明 IEEE浮点数 就可以写一本书了,我将用几篇博文来简单的说说我所理解的浮点数,算是抛砖引玉吧。 一次面试
记得多年前我招聘 Java 程序员时的一次关于浮点数、二分法、编码的面试, 多年以后,他已经称为了一名很出色的
- 数据结构随记_1
lx.asymmetric
数据结构笔记
第一章
1.数据结构包括数据的
逻辑结构、数据的物理/存储结构和数据的逻辑关系这三个方面的内容。 2.数据的存储结构可用四种基本的存储方法表示,它们分别是
顺序存储、链式存储 、索引存储 和 散列存储。 3.数据运算最常用的有五种,分别是
查找/检索、排序、插入、删除、修改。 4.算法主要有以下五个特性:
输入、输出、可行性、确定性和有穷性。 5.算法分析的
- linux的会话和进程组
网络接口
linux
会话: 一个或多个进程组。起于用户登录,终止于用户退出。此期间所有进程都属于这个会话期。会话首进程:调用setsid创建会话的进程1.规定组长进程不能调用setsid,因为调用setsid后,调用进程会成为新的进程组的组长进程.如何保证? 先调用fork,然后终止父进程,此时由于子进程的进程组ID为父进程的进程组ID,而子进程的ID是重新分配的,所以保证子进程不会是进程组长,从而子进程可以调用se
- 二维数组 元素的连续求解
1140566087
二维数组ACM
import java.util.HashMap;
public class Title {
public static void main(String[] args){
f();
}
// 二位数组的应用
//12、二维数组中,哪一行或哪一列的连续存放的0的个数最多,是几个0。注意,是“连续”。
public static void f(){
- 也谈什么时候Java比C++快
windshome
javaC++
刚打开iteye就看到这个标题“Java什么时候比C++快”,觉得很好笑。
你要比,就比同等水平的基础上的相比,笨蛋写得C代码和C++代码,去和高手写的Java代码比效率,有什么意义呢?
我是写密码算法的,深刻知道算法C和C++实现和Java实现之间的效率差,甚至也比对过C代码和汇编代码的效率差,计算机是个死的东西,再怎么优化,Java也就是和C