基因组代谢网络(GEMS)与全基因组模型从入门到实践系列-----(2)文献案例详细讲解
全文目录
- 0 前言说明
- 系列后续更新内容(11.23更新)
- 1. 案例文献
- 2. 研究背景
- 3. 文献中代谢模型框架流程
- 4. 文献code分步讲解
-
- 1)导入目标底盘细胞模型
- 2)将导入的模型进行预处理
- 3)加入反应
- 4)统计参与反应的通路与代谢物
- 5)结果汇总
- 6)计算结果
- 5.中文注释后完整代码
-
- 6.运行常见问题
- 后续更新
- 0 前言说明
0 前言说明
本系列内容说明: 本系列是针对生物研究中的模型种类进行分析介绍,作为**微生物群落相互建模系列的组成一部分。分阶段的介绍代谢网络模型从入门的环境的搭建,到指导湿实验过程全流程分享。探讨基于生物模型对群落中相互作用方式进行,探究挖掘不同生物间的进化关系。
系列的目录文章地址:https://blog.csdn.net/qq_33980829/article/details/118958463
系列后续更新内容(11.23更新)
- 如何完成构建一个乳酸菌的多胺生成网络模型 2021.8.15
- 如何基于代谢网络模型探究群落中的相互作用模式 2021.9.15
- 整理常用的代谢网络构建工具网站。 2021.8.3
1. 案例文献
- 文献题目:Engineering yeast metabolism for the discovery and production of polyamines and polyamine analogues
- 发表时间:2021.4
- 通讯作者:Jens Nielsen(丹麦技术大学)
- 期 刊:Nature Catalysis
- 文章简介分享
- 文献代码中代码地址: https://github.com/foodgeoff2010/Polyamine_platform
2. 研究背景
1.多胺和多胺类似物在工业上作为化工的化学原料同时特定结构的多胺药物价值。含酰胺键的天然多胺类似物,如作为kukoamines,一组植物多胺类似物聚甲基多胺主链(例如,腐胺,亚精胺或亚精胺(SPM)),以及至少一种二氢咖啡酸酸性片段,近年来作为功能性片段受到了关注因其临床和农业特性而成为候选食品和药物。此外,非天然多胺类似物,如N1-鸟苷基-1,7-二氨基庚烷和N1,N11-二乙基降精胺,代表一类具有潜在应用价值的重要抗病毒药物。二元胺作为一种重要的聚合单体,广泛应用于聚酯、聚氨酯、聚酰胺等高分子材料的合成中。
详细综述可以参考文献:王昕, 王静, 陈可泉, 欧阳平凯. 合成生物技术制备脂肪族二元胺的研究进展[J]. 合成生物学, 2020, 1(1): 71-83
3. 文献中代谢模型框架流程
-
尝试本文提高的代码前建议安装系列文章安装对应软件
-
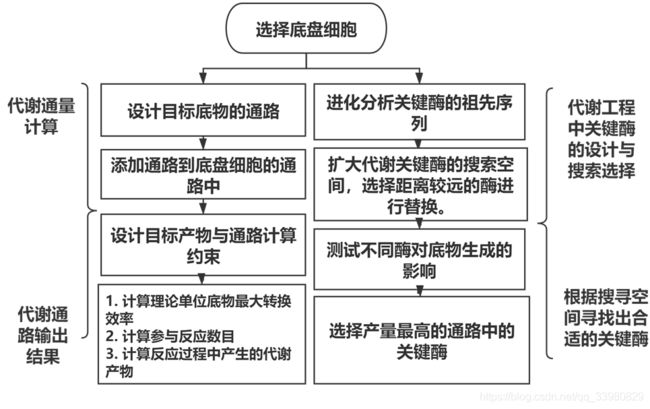
本文献中代谢模型只是作为起始通路设计与产量预测的一小部分内容,核心内容是围绕酵母中多胺和多胺类似物的代谢通路的构建与改造。通过引用祖先酶序列的进化关系进行扩展关键核心酶的选择范围,提高代谢改造的求解空间。
-
文章模型框架
4. 文献code分步讲解
1)导入目标底盘细胞模型
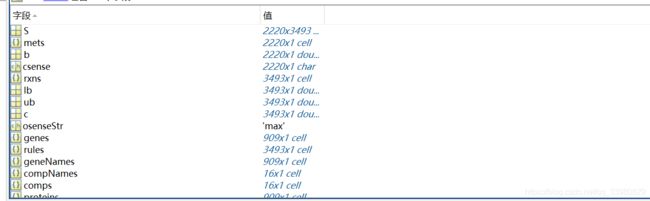
model = readCbModel('yeast_7.6_COBRA.xml');
导入的目标模型中包含了相关代谢通路信息与代谢底物信息。其生成可以通过相关网站的下载(详细见系列(3)),或者本来构建表格利于xls2model进行生成。
2)将导入的模型进行预处理
model = changeObjective(model,'r_2051',1);
% add secretion of 5'-methylthioadenosine
model = addReaction(model,'Sec_s_0303','reactionFormula','s_0303[c_03] -> ');
该过程主要将要加入的代谢产物和通路进行预先修改,将其中的通路中引入多胺,设定生成前提物质来源,对应代谢改造过程对底盘细胞的操作
changeObjective 设定目标产物
3)加入反应
model_1 = model;
model_1 = changeRxnBounds(model_1,'r_0817',0,'b');
model_1 = addReaction(model_1,'M1_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_1 = addReaction(model_1,'M1_2','reactionFormula','agmatine + s_0803[c_03] -> s_1389[c_03] + s_1552[c_03]');
printRxnFormula(model_1,'rxnAbbrList',model_1.rxns(end-1:end),'metNameFlag',1);
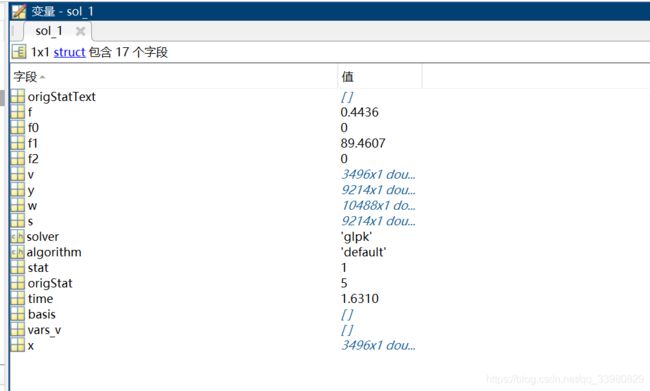
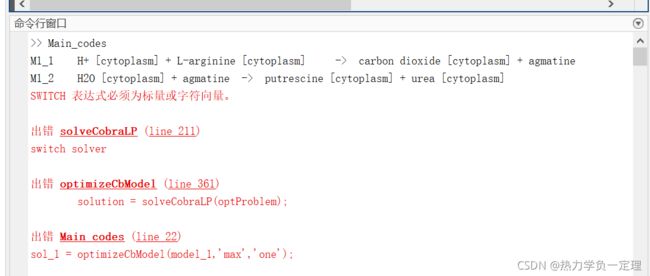
sol_1 = optimizeCbModel(model_1,'max','one');
在第一步改造的基础模型上引入设计的代谢通路,多胺的生成过程,本处主要引入了2个反应
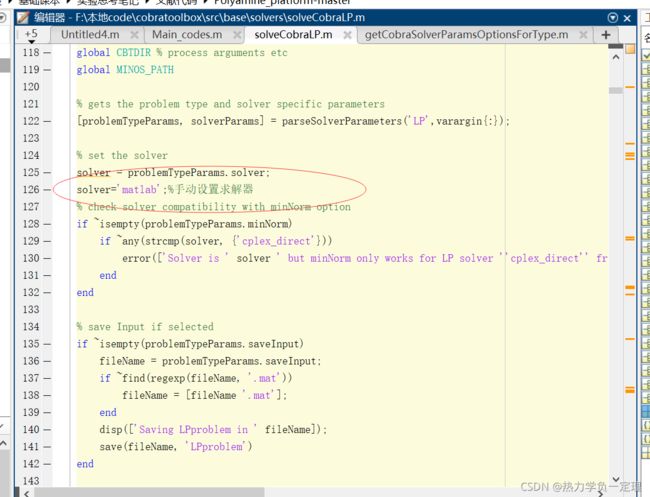
optimizeCbModel步骤是计算底物预测结果 3个参数分布代表 计算的模型 计算的目标要求 计算的约束(具体介绍参考help)
4)统计参与反应的通路与代谢物
[num_rxns_1, num_mets_1] = countActive(model_1,sol_1);
5)结果汇总
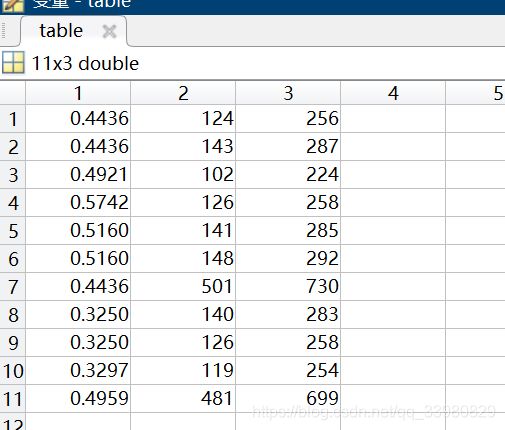
代谢转化效率 参与反应种类 反应代谢物
table = [sol_1.f, num_rxns_1, num_mets_1];
6)计算结果
5.中文注释后完整代码
%% 调用工具包
% cd ../../../GitHub/cobratoolbox;
% initCobraToolbox;
%% Model modification
model = readCbModel('yeast_7.6_COBRA.xml');
% change obj to spermidine production
model = changeObjective(model,'r_2051',1);
% add secretion of 5'-methylthioadenosine
model = addReaction(model,'Sec_s_0303','reactionFormula','s_0303[c_03] -> ');
% printRxnFormula(model,'rxnAbbrList',model.rxns);
%% Calculate yield for different pathways
% Pathway1
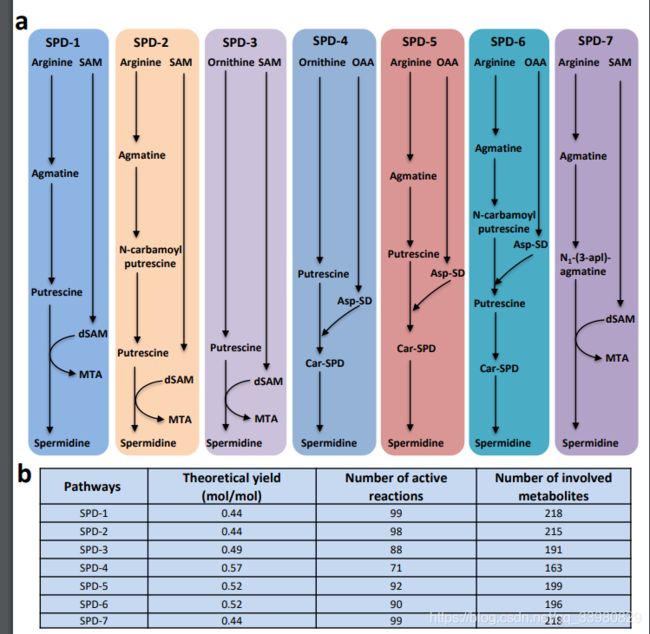
% add arginine decarboxylase(EC 4.1.1.19) and Agmatinase(EC 3.5.3.11)
model_1 = model;
model_1 = changeRxnBounds(model_1,'r_0817',0,'b');
model_1 = addReaction(model_1,'M1_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_1 = addReaction(model_1,'M1_2','reactionFormula','agmatine + s_0803[c_03] -> s_1389[c_03] + s_1552[c_03]');
printRxnFormula(model_1,'rxnAbbrList',model_1.rxns(end-1:end),'metNameFlag',1);
sol_1 = optimizeCbModel(model_1,'max','one');
% Pathway2
% add arginine decarboxylase(EC 4.1.1.19), agmatine deaminase(EC 3.5.3.12)
% and N-carbamoylputrescine amidohydrolase(EC 3.5.1.53)
model_2 = model;
model_2 = changeRxnBounds(model_2,'r_0817',0,'b');
model_2 = addReaction(model_2,'M2_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_2 = addReaction(model_2,'M2_2','reactionFormula','agmatine + s_0803[c_03] -> s_0419[c_03] + N_carbamoylputrescine');
model_2 = addReaction(model_2,'M2_3','reactionFormula','N_carbamoylputrescine + s_0803[c_03] + 2 s_0794[c_03] -> s_1389[c_03] + s_0456[c_03] + s_0419[c_03]');
printRxnFormula(model_2,'rxnAbbrList',model_2.rxns(end-2:end),'metNameFlag',1);
sol_2 = optimizeCbModel(model_2,'max','one');
% Pathway3
% native
model_3 = model;
sol_3 = optimizeCbModel(model_3,'max','one');
% Pathway4
% add carboxyspermidine dehydrogenase (EC 1.5.1.43) and carboxyspermidine
% decarboxylase(EC 4.1.1.-).
model_4 = model;
model_4 = changeRxnBounds(model_4,'r_1001',0,'b');
model_4 = addReaction(model_4,'M4_1','reactionFormula','s_1389[c_03] + s_0978[c_03] + s_0794[c_03] + s_1212[c_03] -> Carboxyspermidine + s_1207[c_03] + s_0803[c_03]');
model_4 = addReaction(model_4,'M4_2','reactionFormula','Carboxyspermidine + s_0794[c_03] -> s_1439[c_03] + s_0456[c_03]');
printRxnFormula(model_4,'rxnAbbrList',model_4.rxns(end-1:end),'metNameFlag',1);
sol_4 = optimizeCbModel(model_4,'max','one');
% Pathway5
% add arginine decarboxylase(EC 4.1.1.19), Agmatinase(EC 3.5.3.11),
% carboxyspermidine dehydrogenase (EC 1.5.1.43) and carboxyspermidine
% decarboxylase(EC 4.1.1.-).
model_5 = model;
model_5 = changeRxnBounds(model_5,'r_0817',0,'b');
model_5 = changeRxnBounds(model_5,'r_1001',0,'b');
model_5 = addReaction(model_5,'M5_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_5 = addReaction(model_5,'M5_2','reactionFormula','agmatine + s_0803[c_03] -> s_1389[c_03] + s_1552[c_03]');
model_5 = addReaction(model_5,'M5_3','reactionFormula','s_1389[c_03] + s_0978[c_03] + s_0794[c_03] + s_1212[c_03] -> Carboxyspermidine + s_1207[c_03] + s_0803[c_03]');
model_5 = addReaction(model_5,'M5_4','reactionFormula','Carboxyspermidine + s_0794[c_03] -> s_1439[c_03] + s_0456[c_03]');
printRxnFormula(model_5,'rxnAbbrList',model_5.rxns(end-3:end),'metNameFlag',1);
sol_5 = optimizeCbModel(model_5,'max','one');
% Pathway6
% add arginine decarboxylase(EC 4.1.1.19), agmatine deaminase(EC 3.5.3.12),
% N-carbamoylputrescine amidohydrolase(EC 3.5.1.53), carboxyspermidine
% dehydrogenase(EC 1.5.1.43), carboxyspermidine decarboxylase(EC 4.1.1.-).
model_6 = model;
model_6 = changeRxnBounds(model_6,'r_0817',0,'b');
model_6 = changeRxnBounds(model_6,'r_1001',0,'b');
model_6 = addReaction(model_6,'M6_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_6 = addReaction(model_6,'M6_2','reactionFormula','agmatine + s_0803[c_03] -> s_0419[c_03] + N_carbamoylputrescine');
model_6 = addReaction(model_6,'M6_3','reactionFormula','N_carbamoylputrescine + s_0803[c_03] + 2 s_0794[c_03] -> s_1389[c_03] + s_0456[c_03] + s_0419[c_03]');
model_6 = addReaction(model_6,'M6_4','reactionFormula','s_1389[c_03] + s_0978[c_03] + s_0794[c_03] + s_1212[c_03] -> Carboxyspermidine + s_1207[c_03] + s_0803[c_03]');
model_6 = addReaction(model_6,'M6_5','reactionFormula','Carboxyspermidine + s_0794[c_03] -> s_1439[c_03] + s_0456[c_03]');
printRxnFormula(model_6,'rxnAbbrList',model_6.rxns(end-4:end),'metNameFlag',1);
sol_6 = optimizeCbModel(model_6,'max','one');
% Pathway7
% add arginine decarboxylase(EC 4.1.1.19), agmatine aminopropyl
% transferase(EC 2.5.1.104) and N1-aminopropylagmatine ureohydrolase.
model_7 = model;
model_7 = changeRxnBounds(model_7,'r_0817',0,'b');
model_7 = changeRxnBounds(model_7,'r_1001',0,'b');
model_7 = addReaction(model_7,'M7_1','reactionFormula','s_0965[c_03] + s_0794[c_03] -> s_0456[c_03] + agmatine');
model_7 = addReaction(model_7,'M7_2','reactionFormula','agmatine + s_1420[c_03] -> N1_3_aminopropyl_agmatine + s_0303[c_03] + s_0794[c_03]');
model_7 = addReaction(model_7,'M7_3','reactionFormula','N1_3_aminopropyl_agmatine + s_0803[c_03] -> s_1439[c_03] + s_1552[c_03]');
printRxnFormula(model_7,'rxnAbbrList',model_7.rxns(end-2:end),'metNameFlag',1);
sol_7 = optimizeCbModel(model_7,'max','one');
clear model;
%% New tasks
model = readCbModel('yeast_7.6_COBRA.xml');
% Pathway8
% Theoretical yield of Spermine
model_8 = model;
% change obj to spermine exchange
model_8 = changeObjective(model_8,'r_2052',1);
sol_8 = optimizeCbModel(model_8,'max','one');
% Pathway9
% Theoretical yield of Thermospermine
% add thermospermine synthase(EC 2.5.1.79) and export of thermospermine
model_9 = model;
model_9 = addReaction(model_9,'M9_1','reactionFormula','s_1420[c_03] + s_1439[c_03] -> s_0303[c_03] + thermospermine + s_0794[c_03]');
model_9 = addReaction(model_9,'M9_2','reactionFormula','thermospermine + s_0796[c_06] -> s_0794[c_03] + thermospermine_e');
model_9 = addReaction(model_9,'M9_3','reactionFormula','thermospermine_e -> ');
printRxnFormula(model_9,'rxnAbbrList',model_9.rxns(end-2:end),'metNameFlag',1);
model_9 = changeObjective(model_9,'M9_3',1);
sol_9 = optimizeCbModel(model_9,'max','one');
% Pathway10
% Theoretical yield of sym-homospermidine (biosynthesis I)
% add homospermidine synthase(EC 2.5.1.45) and export of sym-homospermidine
% and trimethylenediamine
model_10 = model;
model_10 = addReaction(model_10,'M10_1','reactionFormula','s_1439[c_03] + s_1389[c_03] -> sym-homospermidine + s_1526[c_03]');
model_10 = addReaction(model_10,'M10_2','reactionFormula','sym-homospermidine -> sym-homospermidine_e');
model_10 = addReaction(model_10,'M10_3','reactionFormula','sym-homospermidine + s_0796[c_06] -> s_0794[c_03] + sym-homospermidine_e');
model_10 = addReaction(model_10,'M10_4','reactionFormula','sym-homospermidine_e -> ');
model_10 = addReaction(model_10,'M10_5','reactionFormula','s_1526[c_03] -> trimethylenediamine_e');
model_10 = addReaction(model_10,'M10_6','reactionFormula','s_1526[c_03] + s_0796[c_06] -> s_0794[c_03] + trimethylenediamine_e');
model_10 = addReaction(model_10,'M10_7','reactionFormula','trimethylenediamine_e -> ');
printRxnFormula(model_10,'rxnAbbrList',model_10.rxns(end-6:end),'metNameFlag',1);
model_10 = changeObjective(model_10,'M10_4',1);
sol_10 = optimizeCbModel(model_10,'max','one');
% Pathway11
% Theoretical yield of sym-homospermidine (biosynthesis II)
% add homospermidine synthase(EC 2.5.1.44) and export of sym-homospermidine
model_11 = model;
model_11 = addReaction(model_11,'M11_1','reactionFormula','2 s_1389[c_03] -> sym-homospermidine + s_0419[c_03]');
model_11 = addReaction(model_11,'M11_2','reactionFormula','sym-homospermidine -> sym-homospermidine_e');
model_11 = addReaction(model_11,'M11_3','reactionFormula','sym-homospermidine + s_0796[c_06] -> s_0794[c_03] + sym-homospermidine_e');
model_11 = addReaction(model_11,'M11_4','reactionFormula','sym-homospermidine_e -> ');
printRxnFormula(model_11,'rxnAbbrList',model_11.rxns(end-3:end),'metNameFlag',1);
model_11 = changeObjective(model_11,'M11_4',1); %求解的产物,设定需要观察的目标产物, M11_4为底物编号
sol_11 = optimizeCbModel(model_11,'max','one');%求解目标产物的最大转换率,设置求解参数,
clear ans;
%% Active reactions and metabolites
[num_rxns_1, num_mets_1] = countActive(model_1,sol_1);
[num_rxns_2, num_mets_2] = countActive(model_2,sol_2);
[num_rxns_3, num_mets_3] = countActive(model_3,sol_3);
[num_rxns_4, num_mets_4] = countActive(model_4,sol_4);
[num_rxns_5, num_mets_5] = countActive(model_5,sol_5);
[num_rxns_6, num_mets_6] = countActive(model_6,sol_6);
[num_rxns_7, num_mets_7] = countActive(model_7,sol_7);
[num_rxns_8, num_mets_8] = countActive(model_8,sol_8);
[num_rxns_9, num_mets_9] = countActive(model_9,sol_9);
[num_rxns_10, num_mets_10] = countActive(model_10,sol_10);
[num_rxns_11, num_mets_11] = countActive(model_11,sol_11);
%输出结果 代谢转化率 代谢路径长度 通路代谢种类产物
table = [sol_1.f, num_rxns_1, num_mets_1
sol_2.f, num_rxns_2, num_mets_2
sol_3.f, num_rxns_3, num_mets_3
sol_4.f, num_rxns_4, num_mets_4
sol_5.f, num_rxns_5, num_mets_5
sol_6.f, num_rxns_6, num_mets_6
sol_7.f, num_rxns_7, num_mets_7
sol_8.f, num_rxns_8, num_mets_8
sol_9.f, num_rxns_9, num_mets_9
sol_10.f, num_rxns_10, num_mets_10
sol_11.f, num_rxns_11, num_mets_11];
clear num_rxns_1 num_mets_1 num_rxns_2 num_mets_2;
clear num_rxns_3 num_mets_3 num_rxns_4 num_mets_4;
clear num_rxns_5 num_mets_5 num_rxns_6 num_mets_6;
clear num_rxns_7 num_mets_7 num_rxns_8 num_mets_8;
clear num_rxns_9 num_mets_9 num_rxns_10 num_mets_10;
clear num_rxns_11 num_mets_11;
%%
model_tmp = model_11;
sol_tmp = sol_11;
idx = sol_tmp.x ~= 0;
z1_tmp = model_tmp.rxns(idx);
z2_tmp = printRxnFormula(model_tmp,'rxnAbbrList',z1_tmp,'metNameFlag',1);
z3_tmp = sol_tmp.x(idx);
% Count active reactions and metabolites
function [num_rxns, num_mets] = countActive(model,sol)
flux = sol.x;
idx = flux ~= 0;
activeRxns = model.rxns(idx);
num_rxns = length(activeRxns);
activeMets_tmp = {};
for i = 1:num_rxns
met_list = model.mets(model.S(:,i) ~= 0);
for j = 1:length(met_list)
met_id = met_list{j};
if contains(met_id,'[c')
met_id_new = met_id(1:6);
else
met_id_new = met_id;
end
activeMets_tmp = [activeMets_tmp;met_id_new];
end
end
activeMets = unique(activeMets_tmp);
num_mets = length(activeMets);
6.运行常见问题
后续更新
- 如何完成构建一个乳酸菌的多胺生成网络模型
- 如何基于代谢网络模型探究群落中的相互作用模式
- 整理常用的代谢网络构建工具网站。
0 前言说明
本系列内容说明: 本系列是针对生物研究中的模型种类进行分析介绍,作为**微生物群落相互建模系列的组成一部分。分阶段的介绍代谢网络模型从入门的环境的搭建,到指导湿实验过程全流程分享。探讨基于生物模型对群落中相互作用方式进行,探究挖掘不同生物间的进化关系。
系列的目录文章地址:https://blog.csdn.net/qq_33980829/article/details/118958463
系列目的: 前期学习中网上缺少相关文章、文献、资料对生物模型的应用从基础到实践的进行总结。同时由于领域中真正掌握相关知识技巧的不多且请教学习时经常碰壁,因此将学习过程的积累分享总结,文中的定义、分析是根据文献收集和自身知识背景描述只供参考。对文中内容存在疑问或是指正的可以私信于我,或是添加qq:1602480875 进行联系说明。
相关系列后续也会提前发布在我的github :