筛选数据库_筛选TCGA数据库中低级神经胶质瘤微环境中的预后基因
Ann Transl Med
IF:3.297
Published in 2020.3.8
Introduction
肿瘤微环境(TME)是肿瘤灶所在的细胞环境。它由内皮细胞,炎性介质,间充质细胞以及免疫细胞和基质细胞组成。其中,免疫细胞和基质细胞是两个主要的非肿瘤成分,在癌症的诊断和预后中具有重要意义。LGG肿瘤细胞形成了肿瘤微环境的复杂环境,最终促进了肿瘤细胞转录组的适应性和疾病进展。另一方面,研究表明TME可通过调控基因表达对肿瘤恶性程度及临床预后产生重要影响。
为了在LGG中通过ESTIMATE评估观察到TME特性,作者通过使用LGG人群的TCGA数据库和从ESTIMATE算法获得的免疫评分,提取了一系列预测LGG患者预后不良的微环境相关基因,更重要的是,作者开发了一个风险评分系统评估中心基因的预后价值。此外,还探索了中心基因与免疫浸润之间的相关性。
 Result0 1免疫评分和基质评分与LGG亚型,肿瘤等级和生存结果相关
Result0 1免疫评分和基质评分与LGG亚型,肿瘤等级和生存结果相关
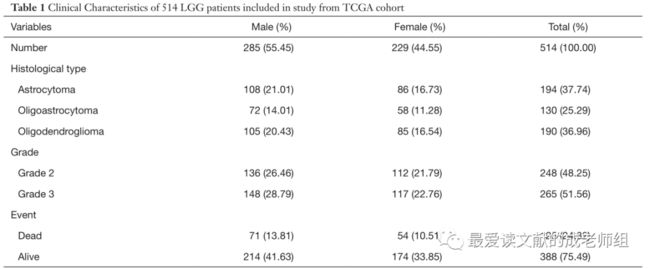
表1
TCGA共收集530条LGG肿瘤数据。然后,作者筛查了516例以-01结尾的样本,代表原发实体瘤。其中,作者删除了样本TCGA-CS-5390-01和TCGA-R8-A6YH-01,因为作者无法获得这些样本的生存信息。
最终从TCGA数据库中提取LGG样本514份,其中男性285份(55.45%),女性229份(44.55%)。LGG患者的临床特征见表1。
图1
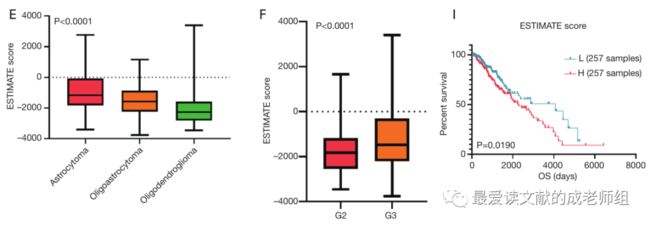
免疫评分、基质评分和估计值分别在:
http://cdn.amegroups.cn/static/application/954ad2d4809e5d6e033eaf346ed86465/atm.2020.01.73-t1.pdf上列出。免疫评分、基质评分和ESTIMATE评分在不同组织学类型和分级中的分布如图1A、B、C、D、E、F所示。结果显示,评分越高与组织学类型和肿瘤分级相关(P<0.0001)。
此外,K-M曲线显示,免疫评分(P=0.0167)、基质评分(P=0.0035)和估计值(P=0.0190)得分越高,生存结局越差(图1G,H,I)。
0 2LGG患者基因表达谱与免疫评分和基质评分的比较图2
DEGs在火山图中得到了反映(图2A)。据统计,684个上调和892个下调的差异表达基因被选在免疫评分组:
(http://cdn.amegroups.cn/static/application/09167c99881221da5e479a7b81a141af/atm.2020.01.73-t2.docx),而409个上调和个下调的差异表达基因被选在基质评分组(http://cdn.amegroups.cn/static/application/4847a27dac216bb9faa00192b15e5428/atm.2020.01.73-t3.docx)。
然后在venn图中发现了785个上调的交叠基因和357个下调的交叠基因(图2B,C)。
0 3功能富集分析图3
GO分析结果显示,BP(图3A)、CC(图3B)和MF(图3C)三类中,有1142个交叠基因与免疫应答、炎症应答、质膜和受体活性相关。
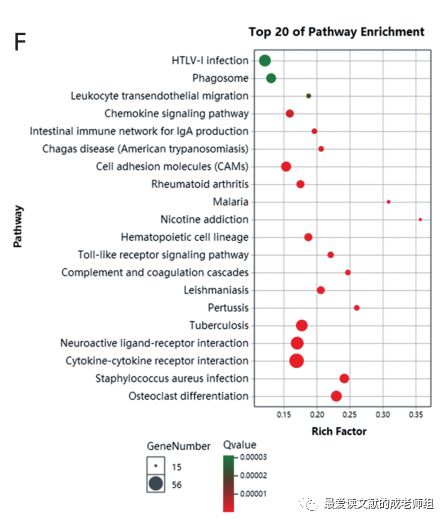
分析KEGG pathway注释,如图3D所示。作者分别计算了交叠基因在生物系统、人类疾病、环境信息处理、细胞过程代谢和遗传信息处理等方面的富集。
从结果中作者发现交叠基因在免疫系统通路、感染性疾病通路、信号转导通路、信号分子与相互作用通路、细胞转运与分解代谢通路、gloable和overview map通路以及折叠、分类和降解通路中最为丰富。
KEGG富集barplot如图3E所示。前20个pathway富集分析如图3F所示,气泡大小代表基因数量,颜色代表Q值。
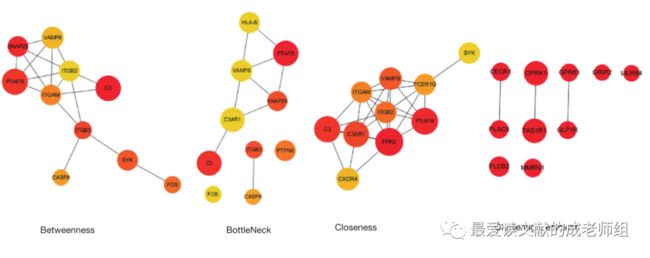
0 4PPI图S4
通过 STRING工具进行PPI网络,探索识别出的DEGs之间的相互作用。
该网络包含1,103个节点和4,070条边。然后通过Cytoscape对STRING数据进行进一步分析,通过cytoHubba对hub基因进行鉴定,算法结果如图S4所示。
25个与肿瘤免疫相关的hub基因分别为:HRH3、APLNR、FCER1G、SYK、GNG12、GNG13、GNG5、PTPN6、GPR183、CXCL11、CXCL9、HLA-B、VAMP8、SSTR1、CCR5、PTAFR、C3、TAS1R1、ANXA1、OPRK1、ITGB2、CCL5、CXCR3、CXCR4、GNGT2。
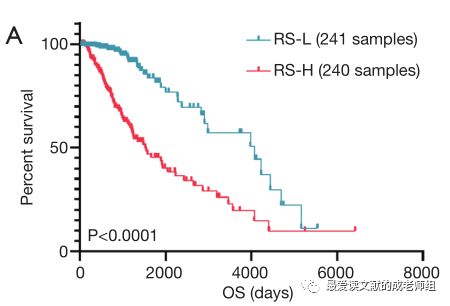
0 5RS和生存分析图4
基于所选择的基因中心,RS=HRH3*(−0.045)+APLNR*0.063+FCER1G*0.03+SYK*(−0.535)+GNG12*0.269+GNG13*0.024+GNG5*0.344+PTPN6*(−0.523)+GPR183*0.346+CXCL11*0.181+CXCL9*(−0.13)+HLA-B*(−0.026)+VAMP8*(−0.302)+SSTR1*(−0.174)+CCR5*0.093+PTAFR*0.23+C3*(−0.37)+TAS1R1*0.171+ANXA1*0.158+OPRK1*0.147+ITGB2*0.381+CCL5*−0.371+CXCR3*0.245+CXCR4*(−0.119)+GNGT2*0.597。
作者从514例样本中选择了481例生存期为30天的患者。然后计算481例患者的RS数据并在网上显示:
http://cdn.amegroups.cn/static/application/e64541dfb3b1cd2b8e9900fe0c982188/atm.2020.01.73-t4.pdf。
RS范围为3.99 ~ 3.03,中位数为0.5。根据中位RS,共481例符合条件的LGG患者被分为低RS组和高RS组。构建生存分析,结果显示高RS组的总生存期比低RS组差(P<0.0001,图4A)。
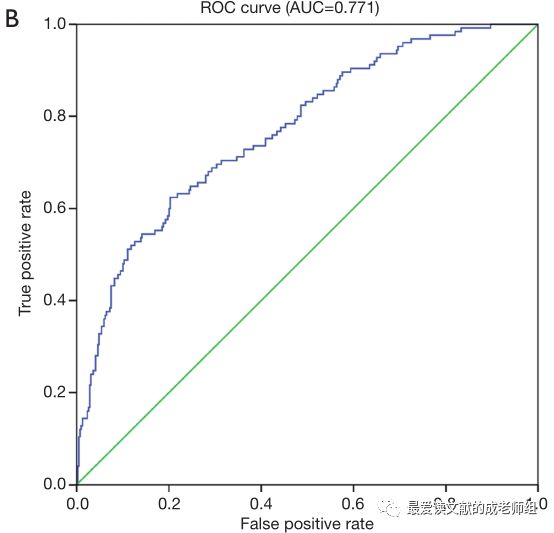
此外,ROC曲线用于评估RS的预后价值。AUC计算为0.771,这表明总生存期的预测准确性更高(图4B)。
另外,25个hub基因的存活曲线如图S5所示。从K-M曲线上看,GNG13、HRH3、OPRK1、SSTR1表达水平低与预后不良相关。相比之下,作者发现LGG患者中其他hub基因的高表达与不良预后相关。
0 6Hub基因和免疫细胞浸润 图S6利用TIMER数据库进一步探讨LGG TME中25个hub基因与免疫浸润的关系。
结果显示,16个Hub基因(ANAX1、C3、CCL5、CCR5、CXCL11、CXCR3、CXCR4、FCER1G、GNG5、GNG12、GNGT2、GPR183、HLA-B、OPRK1、PTAFR、SYK)与B细胞、CD4+T细胞、CD8+T细胞、巨噬细胞、中性粒细胞、树突状细胞浸润相关(P<0.05,图S6)。
Conclusion作者从TCGA数据库中鉴定了25个与TME相关的基因,这些基因与中枢神经系统LGG患者的总体存活率显着相关。
Reference:Screening TCGA database for prognostic genes in lower grade glioma microenvironment
PMID:32309356
DOI:10.21037/atm.2020.01.73