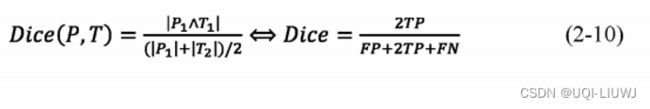论文笔记:CTSpine1K: A Large-Scale Dataset for Spinal Vertebrae Segmentation in Computed Tomography
1 abstract& introduction
由于缺乏大规模的带注释的脊柱图像数据集,基于深度学习的主流分割方法(data-driven的)受到严重限制。
这篇论文介绍了一个名为 CTSpine1K 的大规模脊柱 CT 数据集,该数据集由多个椎体分割的数据源中汇集而成。其中包含 1,005 个 CT volume,其中超过 11,100 个标记的、属于不同的脊柱状况的椎骨。
这个大规模的数据集将促进许多与脊柱相关的图像分析任务的进一步研究,包括但不限于椎骨分割、标记、双平面射线照片的 3D 脊柱重建、图像超分辨率和增强。
2 关于脊柱的一些医学知识
脊柱是肌肉骨骼系统的重要组成部分,维持身体活动性并保护脊髓,脊髓是人体最重要的神经通路 。 人体躯干从上到下,脊柱由7块颈椎(C1-C7)、12块胸椎(T1-T12)、5块腰椎(L1-L5)、1块骶椎和1块尾椎组成 . 请注意,有些人含有 L6(由于骶骨腰椎化)或失去 L5(由于腰椎骶骨化),但这在人群中很少发生。
由于承受人体的负荷,每一个椎骨都有患病的风险。 脊柱相关疾病,包括退行性改变、脊柱炎症、脊柱肿瘤、脊柱结核、脊柱感染等,发病率高,造成巨大的社会成本负担。 在临床实践中,常见的脊柱疾病主要包括脊柱退行性疾病,以及椎间盘突出症。 老年人常见的脊柱退行性疾病包括颈椎病、腰椎病和胸椎管狭窄症。 椎间盘突出症包括涉及颈椎的颈椎间盘突出症、涉及胸椎的胸椎间盘突出症和常见的腰椎间盘突出症,其中以腰椎间盘突出症最为常见。 腰椎退行性疾病也是临床上最常见的,有腰椎管狭窄症、腰椎滑脱、腰椎不稳等。 治疗因疾病实体而异,临床情况可能是非特异性的。
通过计算机断层扫描 (CT)、磁共振成像 (MRI)、射线照相、超声、正电子发射断层扫描 (PET) 和其他放射成像方式进行的脊柱成像对于无创可视化和评估脊柱病理学至关重要。 计算方法支持和增强医生利用这些成像技术进行诊断、无创治疗和临床实践干预的能力。 在计算机视觉、计算机图形学、信号处理和机器学习领域开发的分析算法已适用于分析脊柱图像。
传统上,由于骨软组织对比度高,CT 是研究脊柱的首选方式。 由于椎体位置、金属伪影和脊柱疾病等不同,图像外观变化多样,对分析算法提出了挑战。 图 1 给出了这些不同条件的一些示例。
CT 中的脊柱或椎骨图像分割是所有关于脊柱形态和病理学自动量化的应用中的关键步骤。近年来,深度学习在各种医学成像应用中取得了显著成功,并提出了许多自动脊柱图像分割方法。然而,所有这些方法都依赖于数据,并且只在私有数据集或小型公共数据集上得到验证。考虑到 SpineWeb,一个流行的多模式脊柱数据存档,它只列出了两个 CT 数据集:CSI201410 和 xVertSeg11,这两个数据集都只包含几十个 CT 扫描。因此,这些方法受到严格限制。
为了解决大规模数据可用性的问题,Sekuboyina 等人结合 2019 年医学图像计算和计算机辅助干预国际会议 (MICCAI) 和 MICCAI 2020 组织了大规模椎骨分割基准 (VerSe) 作为一项挑战。通过 VerSe'19,他们向公共领域发布了一个多样化的数据集160 次脊柱多排 CT 扫描,包括 1,735 块椎骨(120 次可见 CT 扫描和 40 次隐藏 CT 扫描)。对于 VerSe'19 的升级版 VerSe'20,他们发布了 300 次 CT 扫描,这是迄今为止最大的公共脊柱 CT 数据集。这两个数据集都提供了每个椎骨的ground truth,并且是目前最常用的椎骨分割数据集。尽管如此,这些数据集仍然很小。此外,来自 VerSe'19 和 VerSe'20 数据集的大多数 CT 扫描都是“裁剪”的,仅包含脊柱的一小部分区域,而放弃了有关周围器官的其他信息。
为了推进脊柱图像分析的研究,我们在此展示了一个大规模的综合数据集:CTSpine1K。 我们收集和注释来自多个领域和不同制造商的大规模脊椎 CT 数据集,总计 1,005 个不同外观变化的 CT volume(超过 500,000 个标记切片和超过 11,000 个椎骨)。 我们精心设计了精致统一的标注流水线,保证标注的质量。 据我们所知,我们的 CTSpine1K 数据集是最大的公开可用注释脊柱 CT 数据集。 我们通过进行椎骨分割的基准实验来评估数据集的质量。
3 方法
3.1 数据采集
为了构建一个能够复制实际外观变化的综合脊柱图像数据集,我们从以下四个开源资源中创建了一个大规模的脊柱椎骨 CT 数据集。
- COLONOG。该子数据集来自与 CT 结肠造影试验相关的 CT COLONOGRAPHY 数据集。我们为我们的数据集随机选择每个患者的两个位置之一,这些位置具有相似的信息。有 825 次 CT 扫描,采用医学数字成像和通信 (DICOM) 格式。
- HNSCC-3DCT-RT。该子数据集包含使用西门子 16 层 CT 扫描仪在治疗前、治疗中和治疗后收集的三维 (3D) 高分辨率扇形束 CT 扫描图像,该扫描仪用于 31 个头部和颈部的鳞状细胞癌 (HNSCC) 患者 。这些图像采用 DICOM 格式。
- MSD T10。该子数据集来自第十届医学分割十项全能比赛。为了获得更多包含脊椎的切片,我们选择了由 201 个案例组成的 task03_liver 数据集。这些图像采用神经影像信息学技术倡议 (NIfTI) 格式。
- COVID-19。该子数据集包含来自 632 名 COVID-19 感染患者的胸部 CT。这些图像是从经逆转录聚合酶链反应 (RT-PCR) 确认存在 SARS-CoV-215 的患者那里获取的。我们挑选了 40 次扫描,图像以 NIfTI 格式存储。
我们将所有 DICOM 图像重新格式化为 NIfTI,以简化数据处理和去识别图像。我们的数据集概览以及与 VerSe Challenge 数据集的全面比较见表 1。
3.2 数据标注
考虑到医学图像标注是一项非常耗时且主观的任务,我们在标注前设计了统一、严格的标注标准和流程。
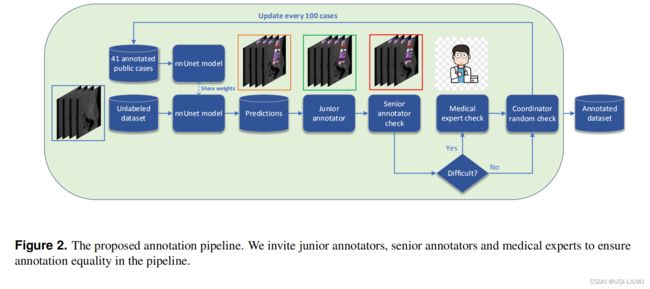
标注流水线如图 2 所示。 为了减少注释工作量,我们首先使用nnUnet 算法分割来自 VerSe'19 和 VerSe'20 Challenges 的公共数据集。
如前所述,大部分 VerSe 的 Challenge 样本都是“裁剪”的,放弃了器官等周围结构的信息。因此,我们选择具有完整 CT 图像(未裁剪),总共可以使用 41 个案例。
然后,对于要标注的图像,我们调用经过训练的分割模型来预测,并邀请一些初级注释者根据预测结果细化标签。所有这些由初级注释者细化的标签都由两名高级注释者检查以进一步细化。
如果高级注释者发现难以确定注释,这些数据将被发送给一位训练有素的脊柱外科医生,他的图像阅读经验平均为 12 年。
最后,所有这些带注释的标签都经过协调员的随机双重检查,以确保注释的最终质量
如果复查中存在错误的情况,则由注释者进行更正。 然后将人工校正的注释及其相应的图像添加到训练数据中,以重新训练更强大的模型。
为了加快注释过程,我们每 100 个案例更新一次数据库并重新训练深度学习模型。 该过程是迭代的,直到注释任务完成。
整个注释过程使用 ITK-SNAP软件进行操作。 以 NIfTI 格式保存。 我们总共有 1005 个 CT volumn 的注释。 该数据集可在 CTSpine 1K 上获得。
4 数据存储形式
- CTSpine1K 数据集由三个子文件夹、两个 xlsx 文件和一个自述文件组成,即 trainset、test_public、test_private、metadata_colonog.xlsx、metadata_neck.xlsx 和 readme.txt。
- 每个子文件夹包含两个名为 data 和 gt 的子文件夹,其中存储了 CT 图像和相应的ground truth。
- metadata.xlsx 文件记录了有关此数据集的详细信息,例如患者 ID、制造商、患者性别、肿瘤位置(如果适用)等(子数据集 COVID19 和 MSD T10 没有此 metadata.xlsx 文件。)
- readme .txt 文件记录了这些病理病例的 ID。
- 所有 CT volumn和ground truth均采用 NIfTI 格式。
数据集结构如下。 ID 1.3.6.1.4.1.9328.50.4.0xxx、HN_Pxxx、liver_xxx 和 volume-covid19-A-0xxx 分别表示来自 COLONOG 数据集、HNSCC-3DCT-RT 数据集、MSD T10 数据集和 COVID-19 数据集的患者。
5 技术验证
基于 CTSpine1K,我们使用完全监督的方法训练深度网络进行脊椎分割,以建立基准。 近年来,nnUnet 模型在许多医学图像分割任务中取得了比其他方法更好的结果,并已成为医学图像分割中公认的基线。
nnUnet本质上是一个U-Net,但具有自适应数据集本身特性的特定网络架构、设计参数和训练参数,以及强大的数据增强。 因此,我们选择 nnUnet 作为椎骨分割的基准模型。 由于我们的数据集中有大量的高分辨率 3D 图像,我们使用 3D 全分辨率 U-net 架构。
5.1 实验配置
5.1.1 数据划分
数据集包含 1005 个 3D CT volumn(平均而言,每次扫描有 504 个切片和 11 个标记的椎骨),有超过 500,000 个标记的椎骨切片(大小为 512x512)。
CTSpine1K 数据集分为训练数据集(610)、公共测试数据集(197)和私密测试数据集(198)
为了比较我们的注释数据集和 VerSe 挑战数据集之间的域差异,我们使用来自 VerSe 挑战的 41 个公共案例作为 Test_VerSe。
5.1.2 评价标准
每个椎骨(从 C1 到 L6)都标有从 1 到 25 的整数值。我们在这里使用医学图像分割领域中普遍的两个指标:
(i)Dice系数(DSC)。
Dice系数是计算两个样本之间的相似度,即考察两个样本之间重叠的范围。Dice系数值范围通常在0-1之间。若为1,则证明两个样本完全重合;若为0,则证明两个样本没有相同的像素。
TP(True Positive)为判定为正样本,事实上也是正样本;
TN(True Negative) 为判定为负样本,事实上也是负样本;
FP(False Positive)为判定为正样本,事实上负样本;
FN(False Negative)为判定为负样本,事实上为正样本。
(ii) Hausdorff 表面距离 95 (HD),单位为毫米。
HD 测量由ground truth和预测分割图构成的两个表面之间的局部最大距离。
6 结果
6.1 定量结果
我们计算了每个椎骨的两个指标,结果如表 2 所示。
一方面,我们的实验结果与使用相同模型(nnUnet)的参考文献 2 中报告的结果接近,验证了我们注释的高质量。
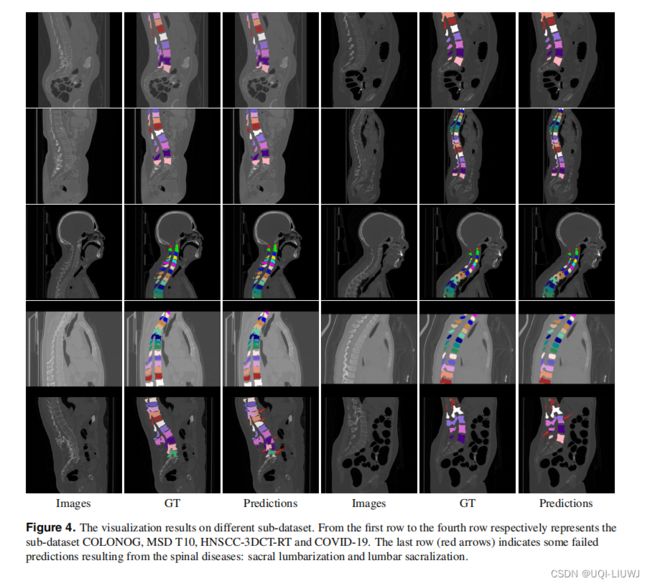
另一方面,表2显示病变椎骨难以分割(L6的DSC几乎为0)。 具体来说,L6 的存在会混淆模型,导致预测错位(参见图 4 中的最后一行)。 因此,我们包含许多 L6 案例的标记数据集对于病变椎骨分割非常有价值
表 2 还说明了使用我们的注释训练的模型可以在我们的 CTSpine1K 数据集上实现良好的性能,但在 VerSe 挑战数据集上的性能要差得多,这说明我们的注释数据集和公共数据集之间存在明显的域差距。 我们推断原因是 COlONOG 数据集是在空腹和结肠上获得的,通过 CT 图像包含的信息少于全胃和结肠(见图 3)。 因此,我们的注释是对现有数据集的一个很好的补充
6.2 定性结果
一些可视化结果如图 4 所示,我们可以观察到基线模型可以实现出色的分割结果。 然而,当存在脊柱疾病,特别是骶椎腰椎化和腰椎骶骨化时,会出现一些失败的预测。
此外,图像 Z 方向的分辨率与结果密切相关,分辨率越低,结果越差。 如何在低分辨率下保持合理的性能是一项研究挑战。 图像超分辨率可能值得探索。