CADD计算机辅助药物设计+先导化合物筛选+薛定谔专题+分子动力学模拟
计算机辅助药物设计是通过计算机模拟、计算和预算药物与受体生物大分子之间的关系,设计和优化先导化合物的方法;近年来,计算机辅助药物设计已经取得一定成就,特别是在新冠肺炎爆发后,很多研究机构和企业采用计算机辅助药物设计方法辅助进行了很多研究工作。在新型冠状病毒的治疗方案中,通过同源建模方法发布了新冠病毒的三维结构,通过药物靶标预测、筛选出针对新型冠状病毒的药物,通过一系列计算机辅助药物生物计算的方法发现一大类药物分子可以有效阻止新冠病毒的侵染,为治疗新冠提供了新机理和新思路。应新老客户的培训需求,北京软研国际信息技术研究院特举办“计算机辅助药物设计技术与实践”系列专题培训班,本次培训由互动派(北京)教育科技有限公司具体承办,具体相关事宜通知如下:
NO.1特色
1、本次计算机辅助药物设计系列课程共分三个专题,《基础班:CADD专题》、《进阶班:先导化合物筛选专题》、《进阶班:薛定谔专题》。每个专题单独小班授课,运用不同的专业软件对知识进行由浅入深,层层递进分阶段系统讲解,配合案例边讲边练,让学员能运用专业软件针对每个技术点进行上机操作;
2、帮助学员掌握和运用计算机辅助药物设计专业软件工具应用于先导化合物发现和优化的药物分子设计过程,提高药物设计水平、速度和成功率,使药物设计从基于偶然性趋向于定向化和合理化。课堂上建立班级交流平台,学员学完后可以继续与老师同学交流问题,巩固学习内容。
大纲
生物分子互作基础:
1.生物分子互作用研究方法
1.1蛋白-小分子、蛋白-蛋白相互作用原理
1.2 分子对接研究生物分子相互作用
1.3 蛋白蛋白对接研究分子相互作用
蛋白数据库:
1. PDB 数据库介绍
1.1 PDB蛋白数据库功能
1.2 PDB蛋白数据可获取资源
1.3 PDB蛋白数据库对药物研发的重要性
2.PDB 数据库的使用
2.1 靶点蛋白结构类型、数据解读及下载
2.2 靶点蛋白结构序列下载
2.3 靶点蛋白背景分析及相关数据资源获取途径
2.4 批量下载蛋白晶体结构
蛋白结构分析
1. Pymol 软件介绍
1.1 软件安装及初始设置
1.2 基本知识介绍(如氢键等)
2.Pymol 软件使用
2.1蛋白小分子相互作用图解
2.2 蛋白蛋白相互作用图解
2.3 蛋白及小分子表面图、静电势表示
2.4蛋白及小分子结构叠加及比对
2.5绘相互作用力
2.6 Pymol动画制作
3.实例讲解与练习:
(1)尼洛替尼与靶点的相互作用,列出相互作用的氨基酸,并导出结合模式图
(2)制作结合口袋表面图
(3)Bcr/Abl靶点的PDB结构叠合
(4)制作蛋白相互作用动画
(5)针对ACE2和新冠病毒Spike的蛋白晶体复合物,制作蛋白-蛋白相互作用图
微信公众号:科研记
Cartoon Surface Surface of binding site
图一:Pymol 软件制作蛋白表面图
同源建模:
1. 同源建模原理介绍
1.1 同源建模的功能及使用场景
1.2 同源建模的方法
2. Swiss-Model 同源建模;
2.1 同源蛋白的搜索(blast等方法)
2.2 蛋白序列比对
2.3 蛋白模板选择
2.4 蛋白模型搭建
2.5 模型评价(蛋白拉曼图)
2.6 蛋白模型优化
微信公众号:科研记
图二:最佳模型结果Spike: Model_03 3.实例讲解与练习:用2019-nCoV spike蛋白序列建模,根据相应参数和方法评价模型
小分子构建:
1. ChemDraw软件介绍
1.1小分子结构构建
1.2 小分子理化性质(如分子量、clogP等)计算
2.实例讲解与练习:分别构建大环、氨基酸、DNA、RNA等分子小分子化合物库:
1. 小分子数据库
1.1 DrugBank、ZINC、ChEMBL等数据库介绍及使用
1.2 天然产物、中药成分数据库介绍及使用
生物分子互作用ⅠAutodock:
1.分子对接基础
1.1分子对接原理及对接软件介绍
2. 分子对接软件(Autodock) 使用
2.1半柔性对接
2.1.1 小分子配体优化准备
2.1.2 蛋白受体优化及坐标文件准备
2.1.3 蛋白受体格点计算
2.1.4 半柔性对接计算
2.2对接结果评价
2.2.1 晶体结构构象进行对比
2.2.2 能量角度评价对接结果
2.2.3 聚类分析评价对接结果
2.2.4 最优结合构象的选择
2.2.5 已知活性化合物对接结果比较
微信公众号:科研记
图三:PDB 1IEP
3.实例讲解与练习:针对1IEP完成半柔性对接、柔性对接 虚拟筛选:
2.3柔性对接
2.3.1 小分子配体优化准备
2.3.2 蛋白受体优化及坐标文件准备
2.3.3 蛋白受体格点计算
2.3.4 柔性对接计算
2.3.5 柔性对接结果评价
2.3.6 半柔性对接与柔性对接比较与选择
3. 分子对接用于虚拟筛选(Autodock)
3.1 虚拟筛选定义、流程构建及演示
3.2 靶点蛋白选择
3.3化合物库获取
3.4虚拟筛选
3.5 结果分析(打分值、能量及相互作用分析)
3.实例讲解与练习:对1IEP晶体复合物完成虚拟筛选,并对结果进行分析
答疑:针对前两天学习问题的答疑
生物分子互作用IIZDOCK:
1.预测蛋白-蛋白相互作用(ZDOCK)
1.1 受体和配体蛋白前期优化准备
1.2 载入受体和配体分子
1.3蛋白蛋白相互作用对接位点设定
1.4蛋白蛋白对接结果分析与解读
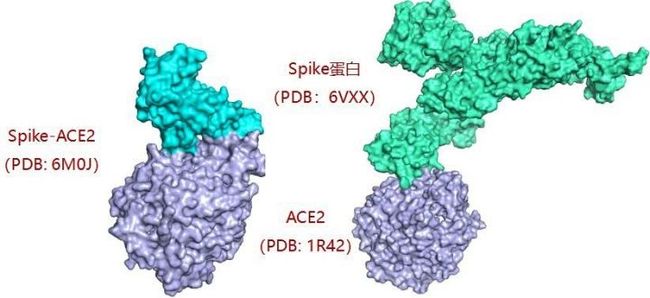
2. 实例讲解与练习:ZDOCK模拟2019-nCoV的spike蛋白6VXX和ACE2蛋白晶体结构的相互作用
微信公众号:科研记
图四:Spike-ACE2和ZDOCK results的比较
构效关系分析:
1. 3D-QSAR模型构建(Sybyl软件)
1.1 小分子构建
1.2 创建小分子数据库
1.3 小分子加电荷及能量优化
1.4 分子活性构象确定及叠合
1.5 创建3D-QSAR模型
1.6 CoMFA和CoMSIA模型构建
1.7 测试集验证模型
1.8模型参数分析
1.9模型等势图分析
1.10 3D-QSAR模型指导药物设计
2. 实例讲解与练习:搜集Imatinib或Bcr/Abl同靶点抑制剂小分子的类似物结构及其活性值,分别构建COMFA与COMSIA模型,并调整参数,获得最佳预测模型
基于碎片药物设计:
1. 基于碎片药物设计
1.1基于碎片的药物设计与发现
1.2 基于碎片化合物库构建
1.2.1 骨架替换
1.2.2 碎片连接
1.2.3 碎片生长
1.3 基于药效团的化合物库生成
1.4 基于蛋白结合口袋的化合物库生成
1.5 基于分子描述符的化合物库生成
1.6 基于BREED规则的化合物库构建
1.7 基于碎片的化合物库筛选
2. 实例讲解与练习:利用碎片替换、碎片链接、碎片生长以及BREED对晶体复合物中的小配体进行碎片化合物的生成,并对结果进行分析
微信公众号:科研记
图五:生成小配体碎片库
2. 实例讲解与练习:利用碎片替换、碎片链接、碎片生长以及BREED对晶体复合物中的小配体进行碎片化合物的生成,并对结果进行分析
分子动力学模拟:
1. 分子动力学模拟简介
1.1 分子动力学基本原理
1.2 分子动力学模拟在药物设计中的作用
系列课程(二)进阶班:先导化合物筛选线上培训课表
| 第一天上午 |
新药先导筛选新策略 |
1. 运用组合筛选全新方法发现新药先导化合物1.1 基于药效团的虚拟筛选方法1.2 基于分子对接的虚拟筛选方法 1.3 组合筛选的基本原理1.4 组合筛选全新方法-用于先导化合物的快速发现 |
| 数据库构建和类药性筛选 |
2. 数据库的构建和类药性筛选2.1 分子结构及小分子数据库的构建2.2 多肽数据库的构建2.3 类药性分析2.4 小分子数据库的类药性筛选实例讲解与练习: (1) 小分子数据库构建和类药性分析(2) 多肽数据库构建 |
2.1 分子结构及小分子数据库的构建 2.2 多肽数据库的构建 2.3 类药性分析 2.4 小分子数据库的类药性筛选 实例讲解与练习:
|
| 第一天下午 |
基于受体的药效团建模实践 |
3. 基于受体的药效团建模流程及结果分析3.1 建模操作流程3.1.1 靶点蛋白-配体复合物的选择3.1.2 靶点蛋白-配体复合物前期优化准备3.1.3 靶点蛋白与配体的相互作用分析3.1.4 药效团模型构建3.1.5 测试集的建立和药效团模型的验证3.2 虚拟筛选3.3 结果分析实例讲解与练习:(1) 基于受体药效团模型及虚拟筛选方法发现新型PDE4抑制剂(2) 基于PARP-1活性位点构建三维药效团模型及先导化合物筛选 |
| 第二天上午 |
基于配体的药效团建模实践 |
4. 基于配体的药效团建模流程及结果分析4.1 建模操作流程4.1.1 配体的选择与处理4.1.2 多构象小分子数据库的构建4.1.3 基于配体的药效团模型构建4.1.4 测试集的建立和药效团模型的验证4.2 虚拟筛选4.3 结果分析实例讲解与练习:(1) 基于配体的药效团建模和虚拟筛选方法发现新型MDM2抑制剂(2) 基于配体药效团模型从数据库中发现全新的微管蛋白抑制剂 |
| 第二天下午 |
组合筛选方法的实例分析 |
5. 基于组合筛选方法发现全新新药先导化合物目标:从数据库中发现微管蛋白、PARP-1、MDM2和PLK1等靶点的全新新药先导化合物实例讲解与练习(举一反三-学以致用):(1) 运用组合筛选方法发现全新的MDM2抑制剂(2) 运用组合筛选方法发现全新的PLK1抑制剂(3) 运用组合筛选方法发现全新的新冠病毒PLpro抑制剂(4) 运用组合筛选方法发现全新的PD-L1靶点抑制剂 |
系列课程(三)进阶班:薛定谔专题线上培训课表
| 时间 |
内容 |
课程内容 |
| 第一天上午 |
蛋白-配体相互作用 |
1. 分子对接基础、原理及对接软件介绍2. 分子对接软件(薛定谔) 使用 2.1半柔性对接 2.1.1 小分子配体优化准备 2.1.2 蛋白受体优化及坐标文件准备2.1.3 蛋白受体格点计算2.1.4 半柔性对接计算 2.2 对接结果评价2.2.1 晶体结构构象比较2.2.2 打分角度评价对接结果2.2.3 构象角度评价对接结果2.2.4 最优结合构象选择2.2.5 已知活性化合物对接结果比较实例讲解与练习:Bcr/Abl靶点抑制剂的分子对接 |
| 第一天下午 |
柔性对接及基于结构的虚拟筛选 |
2.3 诱导契合柔性对接 2.3.1 小分子配体优化准备2.3.2 蛋白受体优化及坐标文件准备2.3.3 蛋白受体格点计算2.3.4 柔性对接计算2.3.5 柔性对接结果评价2.3.6 半柔性对接与柔性对接比较与选择3. 基于结构的虚拟筛选(薛定谔) 3.1 虚拟筛选定义、流程构建及演示3.2 靶点蛋白选择3.3 化合物库获取3.4 虚拟筛选3.5 结果分析(打分值、能量项分解及相互作用)4. 共价对接(薛定谔)4.1 蛋白结构准备4.2 小分子结构准备4.3 共价对接4.4 共价对接结果分析实例讲解与练习:1)Bcr/Abl靶点抑制剂的虚拟筛选2)EGFR靶点抑制剂共价对接 |
| 第二天上午 |
基于药效团虚拟筛选 |
5. 药效团构建虚拟筛选(薛定谔)及结果分析5.1 配体和受体结构准备及优化5.2 基于多配体共同特征构建药效团5.3 基于蛋白-配体复合物构建药效团5.4 药效团的特征修饰5.5 药效团的富集分析5.6 基于药效团的虚拟筛选5.7 基于药效团虚拟筛选结果分析实例讲解与练习:基于药效团的Bcr/Abl抑制剂虚拟筛选 |
| 第二天下午 |
构效关系模型预测分子活性 |
6. 构效关系模型(3D-QSAR)构建及化合物活性分析6.1 构效关系基本理论介绍6.2 分子数据集准备6.3 3D-QSAR模型构建6.4 3D-QSAR模型结果分析6.5 3D-QSAR模型预测新化合物活性实例讲解与练习:Bcr/Abl抑制剂3D-QSAR构建及分子活性预测 |
其它相关:《Amber分子动力学技术与应用专题培训》《Gromacs分子动力学技术与应用专题培训》《人工智能药物设计技术与应用专题培训》具体介绍请咨询联系人!
问答:
课程讲师是否专业?
专题课程(一)、(三)讲师:由全国重点大学、国家“双一流”、“211工程”重点建设医药类高校,医药信息学博士、副教授,硕士生导师、哈佛访问学者。在国内外学术期刊发表研究论文二十余篇,曾先后主持和参与国家自然科学基金和省自然科学基金数项。拥有十余年的抗肿瘤、抗病毒领域新药分子设计开发经验,主要擅长计算机辅助药物设计、人工智能药物设计方法等。
专题课程(二)讲师:全国重点大学、国家“双一流”、“211工程”重点建设医药类高校,药物分析系博士、副教授,硕士生导师。在国际著名学术期刊发表论文十余篇、申请专利4项、单篇论文影响因子最高超过15(第一通讯作者),影响因子累计大于50。曾先后主持和参与国家自然科学基金、省自然科学基金和中央高校基本科研业务费专项基金。拥有十余年的抗肿瘤、抗病毒领域新药分子设计开发经验,主要擅长运用人工智能和计算机辅助设计技术,针对疾病和药物分子作用的关键靶点进行靶向识别、分子探针设计、合成及检测肿瘤标志物、建立多肽数据库库,筛选候选多肽药物等。
专题课程(四)讲师:山东省属重点高校特聘教授、省优青,研究团队在《J. Am. Chem. Soc.》、《Phys. Chem. Chem. Phys.》、《Nanoscale》、《J. Chem. Phys.》、《Appl. Phys. Lett.》、《J. Phys. Chem. B》等国际知名期刊发表SCI论文50余篇。其中以第一作者发表的两篇JACS属于本领域顶级TOP期刊(一区论文,IF: 14.612)。h-index为15。主持国家自然科学基金4项目,省部级课题多项。主要从事生物分子的量化计算和分子动力学模拟研究,在动力学模拟、量化计算、结合自由能计算等积累了丰富的经验。
详细链接如下 可咨询:
https://mp.weixin.qq.com/s/ojtFxNjMG2dzkQY8EoKALA