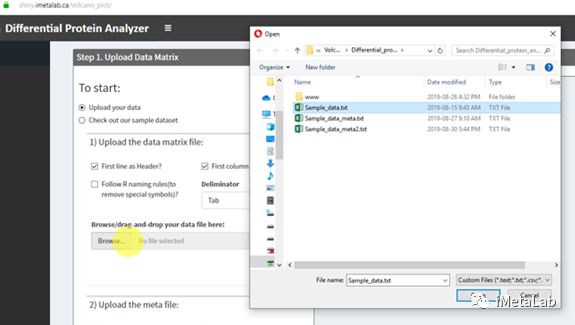
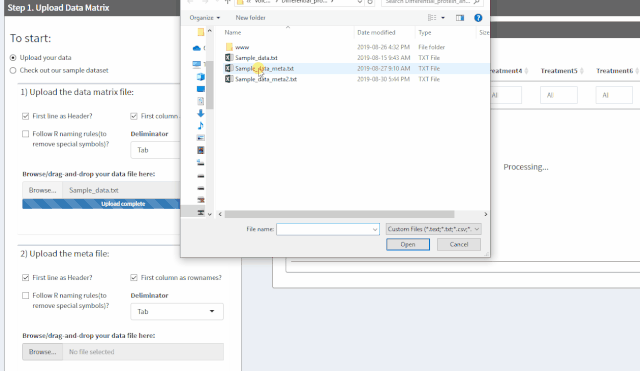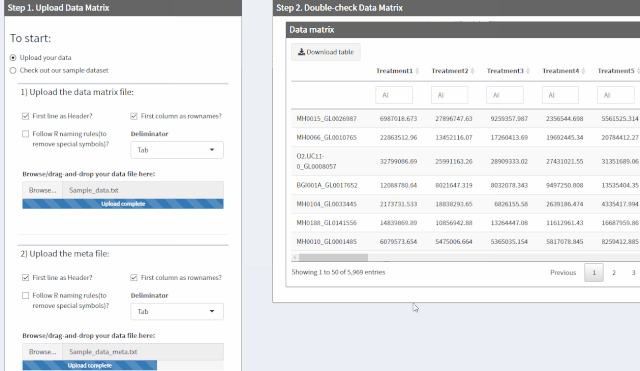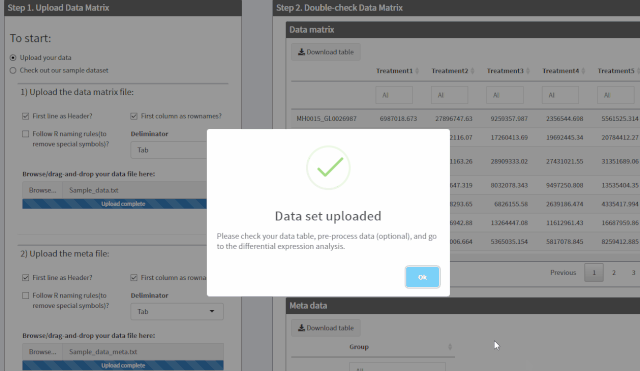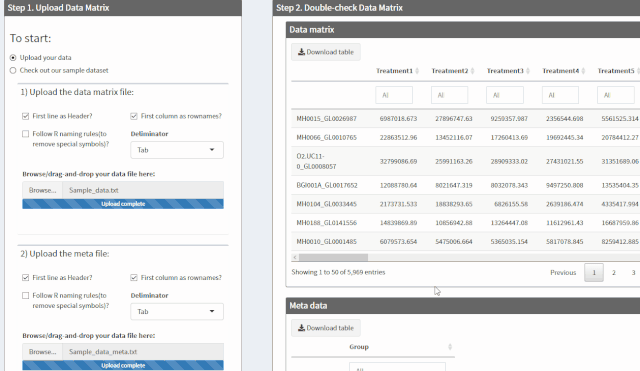
- 如何在YashanDB数据库中实现数据模型的简化
数据库
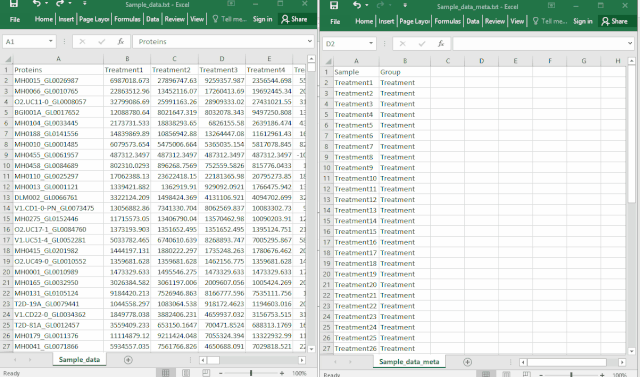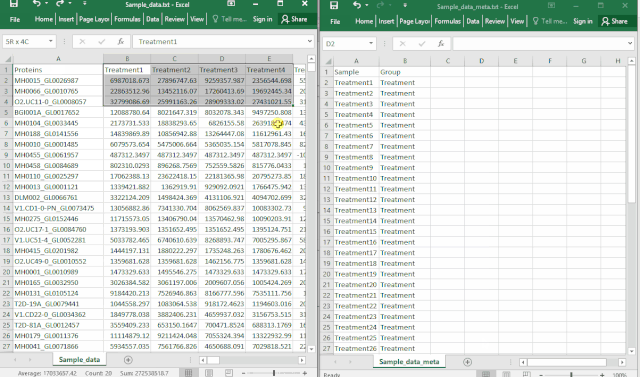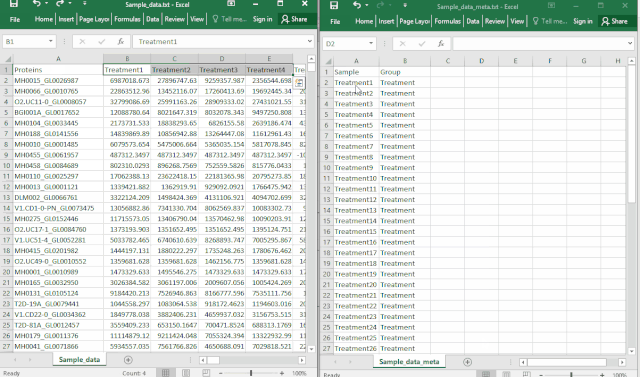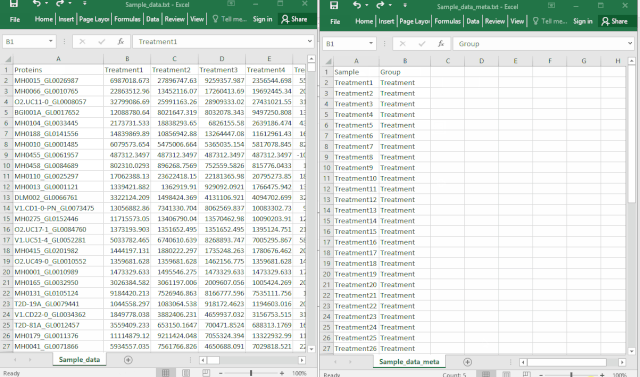
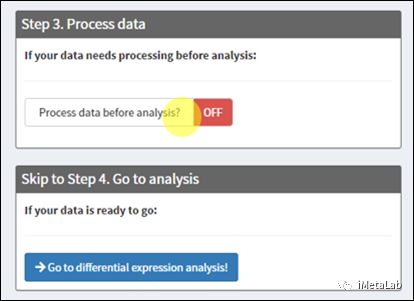
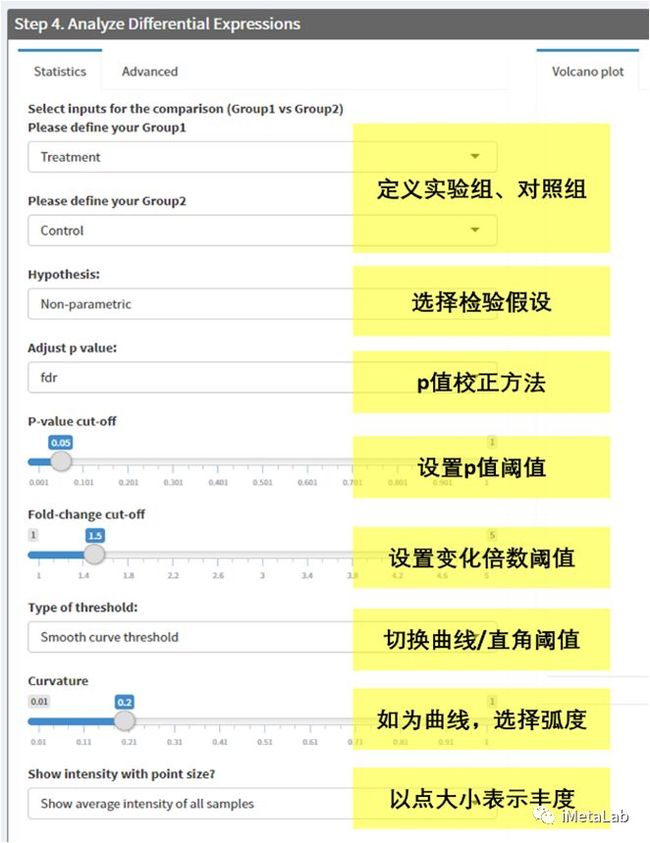
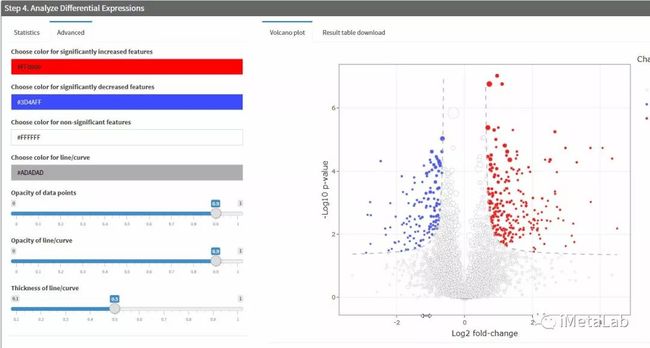
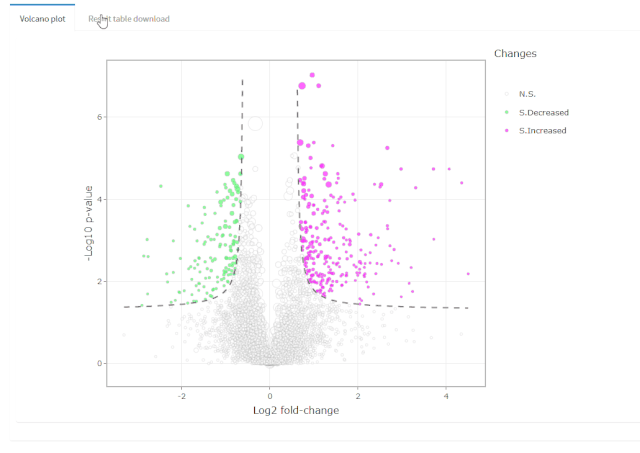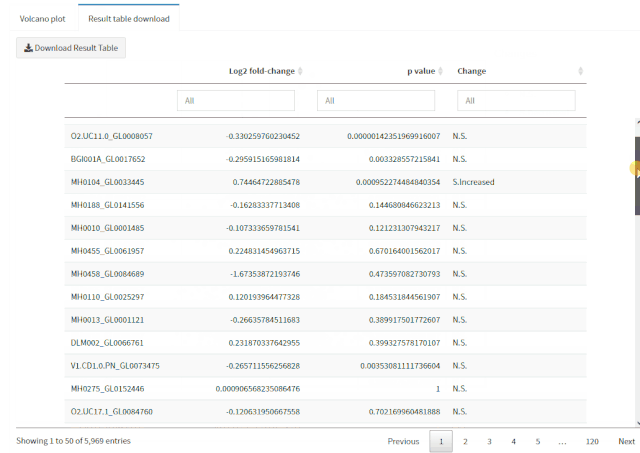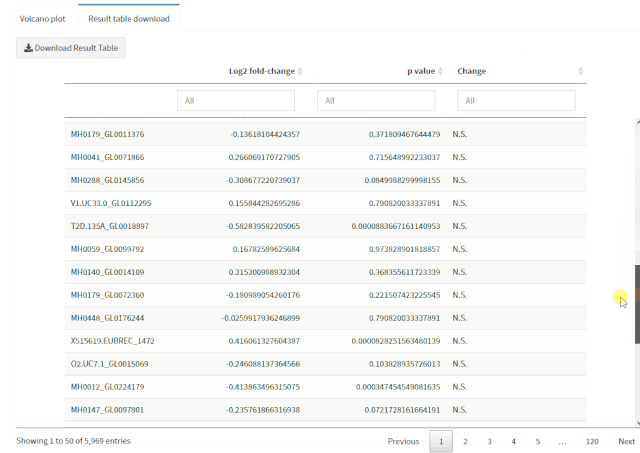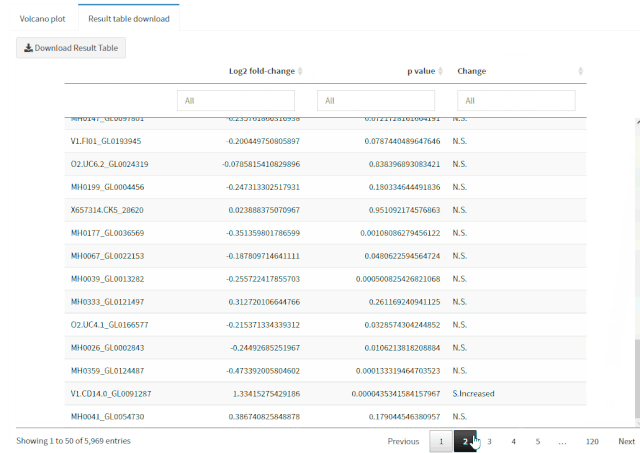
在现代数据库技术领域,数据模型的复杂性经常导致性能瓶颈和维护困惑。随着数据规模的增长和业务诉求的增加,复杂的数据结构、冗余的存储和不必要的关联关系都会影响整体数据库的性能和可维护性。特别是在面对动态变化的业务需求时,灵活性和扩展性成为关键因素。YashanDB提供了一系列功能强大的工具和机制,能够有效简化数据模型,提升数据库性能,并增强数据操作的灵活性。本文章旨在为数据库开发者和架构师提供技术洞见
- Git 常用命令指南:从入门到精通
Takumilovexu
小技巧git
文章目录前言1.初始化一个Git仓库2.克隆远程仓库3.查看仓库状态4.添加文件到暂存区5.提交代码6.推送到远程仓库7.拉取远程仓库的更改8.分支管理9.查看提交历史10.回退到某个版本结语前言如果你是一位开发者或者对代码感兴趣,那么你一定听说过Git。Git是目前最流行的版本控制系统,可以帮助你管理代码,跟踪历史变化,甚至还能协作开发项目。虽然Git功能强大,但对初学者来说,刚开始使用时可能会
- 设计模式系列(10):结构型模式 - 桥接模式(Bridge)
系列导读:在学习了接口适配后,我们来看如何处理抽象与实现的分离问题。桥接模式解决的是"多维度变化"的设计难题。解决什么问题:将抽象部分与实现部分分离,使它们都可以独立变化。避免在多个维度上变化时出现类爆炸问题。想象一下,你要设计一个图形绘制系统,既要支持不同的形状(圆形、矩形),又要支持不同的绘制方式(Windows绘制、Linux绘制)。如果用继承,你需要WindowsCircle、LinuxC
- PTP 与 gPTP 的对比解析
ftdlk
人形机器人机器人自动驾驶
PTP与gPTP的对比解析PTP(PrecisionTimeProtocol)和gPTP(generalizedPrecisionTimeProtocol)均为高精度时间同步协议,但设计目标、应用场景及技术实现存在显著差异。以下是两者的核心区别:1.协议标准与目标特性PTP(IEEE1588)gPTP(IEEE802.1AS)标准版本IEEE1588-2008(主流)/IEEE1588-2019I
- 推荐算法(推广搜)——广告和推荐有什么不同?
导语近几年新兴起一个行业:推广搜。即推荐、广告、搜索算法的简称。各大厂都隐隐将其作为公司核心技术来发展。此文将带领大家探秘广告和推荐有什么区别以及其相似处。再此强调一下,广告算法里面的推荐广告和自然推荐结果里的推荐系统进行对比,但因为广告算法里面还有“搜索广告”,搜索广告和推荐系统差异性就太大了,这里不做讨论。一、不同点1.1本质不同推荐广告和自然推荐本质中要处理的群体和衡量的利益完全不一样。(图
- MySQL与SQLite区别
GoKu~
mysqlsqlite
MySQL和SQLite都是关系型数据库管理系统(RDBMS),它们都使用SQL(结构化查询语言)作为标准查询语言。然而,尽管它们共享许多共同点,但它们在语法、功能、性能和存储机制方面存在一些差异。以下是一些主要的差异:1.存储引擎:-MySQL:支持多种存储引擎,如InnoDB、MyISAM、Memory等,每种存储引擎都有不同的特性,如事务支持、索引类型、数据存储方式等。-SQLite:只有一
- 2025年渗透测试面试题总结-2025年HW(护网面试) 31(题目+回答)
独行soc
2025年护网面试职场和发展安全linux护网渗透测试
安全领域各种资源,学习文档,以及工具分享、前沿信息分享、POC、EXP分享。不定期分享各种好玩的项目及好用的工具,欢迎关注。目录2025年HW(护网面试)311.自我介绍2.渗透测试流程(五阶段模型)3.技术栈与开发经历4.自动化挖洞实践5.信息搜集方法论6.深度漏洞挖掘案例8.SQL注入实战技巧9.AWVS扫描与防御10.CSRFvsSSRF核心差异11.SSRF正则绕过技术12.虚拟主机识别原
- 从0到1打造创始人IP:创客匠人如何用内容构建商业护城河
创客匠人老蒋
创始人IP创客匠人IP变现大数据知识付费
创始人IP为何成为企业破局的关键引擎?在知识付费赛道竞争白热化的当下,创客匠人创始人老蒋以“IP新商业架构师”的身份,将个人IP与企业品牌深度绑定,走出了一条差异化路径。当传统企业还在纠结流量成本时,老蒋通过输出“成事心法”“商业认知”等干货内容,在公众号、短视频等平台积累精准用户,其“正确的事做长期”理念,正是创客匠人9年深耕行业的缩影。这种将创始人个人影响力转化为企业信任背书的模式,让创客匠人
- 深度解析:venv和conda如何解决依赖冲突难题
咕咕日志
condapython
文章目录前言一、虚拟环境的核心价值1.1依赖冲突的典型场景1.2隔离机制实现原理二、venv与conda的架构对比2.1工具定位差异2.2性能基准测试(以创建环境+安装numpy为例)三、venv的配置与最佳实践3.1基础工作流3.2多版本Python管理四、conda的进阶应用4.1环境创建与通道配置4.2混合使用conda与pip的风险控制4.3跨平台环境导出五、工具选型决策树5.1场景化推荐
- SVN简介
Bu Sir
SVN初体验svn
svn介绍:1.项目管理中的版本控制问题:①解决代码冲突困难;②容易引发bug;③难于恢复至以前正确的版本;④无法进行权限控制;⑤项目版本发布困难;2.什么是版本控制:版本控制是维护工程蓝图的标准做法,能追踪工程蓝图从诞生一直到定案的过程。是一种记录若干文件内容变化,以便将来查阅特定版本修订情况的系统。3.svn是什么:SVN是版本管理工具,在当前的开源项目里(J2EE),几乎都会使用SVN。Su
- SVN笔记之SVN启动模式
SVN开源代码的版本控制系统一、生命周期创建版本库→检出→更新→执行变更→复查变化→修复错误→解决冲突→提交更改二、SVN启动模式首先,在服务端进行SVN版本库的相关配置手动新建版本库目录mkdir/opt/svn利用svn命令创建版本库svnadmincreate/opt/svn/runoob使用命令svnserve启动服务svnserve-d-r目录--listen-port端口-r:配置方式
- STM32-内存运行原理与RAM执行实战
东方少爷
内存地址单片机嵌入式硬件arm开发硬件工程stm32
一、底层原理深度解析(先懂“为什么要拷贝”)1.存储介质本质差异(ROM/FlashvsRAM)ROM(以STM32内部Flash为例):物理特性:电可擦写非易失性存储(虽叫ROM,实际可通过编程改写),擦写次数有限(一般万次级别),读速度慢(STM32F1系列Flash读取周期约30-50ns)。存储内容:程序代码(指令)、只读常量(const修饰的全局变量、字符串字面量)、初始化的全局变量(R
- 高效沟通05-FFC赞美法则
古城码农
工作记实录高效沟通沟通技巧
效沟通专栏–组织运转的命脉与个人成功的基石目录1.概念1.1底层逻辑1.2优势1.3适用场景1.4技巧2.示例1.概念 FFC是由三个英文单词首字母组成的结构化赞美模型,旨在让赞美真实可信、打动人心,避免空洞敷衍。其核心是通过三个层次构建逻辑闭环:要素含义作用关键要点F(Feeling)感受表达主观情感反应用情绪词传递真诚(如“感动”“佩服”)例:“你的方案让我非常惊喜!”F(Fact)事实提供
- Vue动态绑定Class与Style
一、动态绑定Class1.1对象语法v-bind:class指令是Vue.js中用于动态绑定CSS类的指令。它可以根据Vue实例中的数据来动态添加或移除HTML元素的类。这样可以根据数据的变化来动态改变元素的样式,实现更灵活的样式控制。语法class-name:要绑定的CSS类名condition:一个表达式,当为true时,class-name会被添加;当为false时,class-name会被
- 项目管理中,范围管理和需求管理有什么区别
易成技术团队
即时通讯工具
范围管理与需求管理在项目管理中存在以下五大核心区别:1、关注层级不同、2、管理目标不同、3、输出成果不同、4、涉及角色不同、5、所用工具与流程不同。其中,“关注层级不同”是二者的根本分野。需求管理聚焦于收集和分析利益相关者的真实需求,回答“用户想要什么”;而范围管理则是在项目层面明确要交付的全部工作,回答“我们做什么、不做什么”。这种层级差异决定了需求是制定范围的输入源,范围是实现需求的边界框架,
- 爆改RAG!用强化学习让你的检索增强生成系统“开挂”——从小白到王者的实战指南
许泽宇的技术分享
人工智能
“RAG不准?RL来救场!”——一位被RAG气哭的AI工程师前言:RAG的烦恼与AI炼丹师的自我修养在AI圈混久了,大家都知道RAG(Retrieval-AugmentedGeneration,检索增强生成)是大模型落地的“万金油”方案。无论是企业知识库、智能问答,还是搜索引擎升级,RAG都能插上一脚。但你用过RAG就知道,理想很丰满,现实很骨感。明明知识库里啥都有,问个“量子比特的数学表达式”,
- DTW 动态时间规整:时间序列的柔性桥梁
在时间的长河中,数据如浪花般不断涌现,而时间序列数据更是其中璀璨的存在。当我们试图比较两段时间序列时,常常会遇到一个棘手的问题:就像两位舞者,他们演绎着相同的舞蹈,却有着不同的节奏与速度,直接对比难以判断二者的相似度。而DTW(DynamicTimeWarping,动态时间规整)算法,就像一座神奇的柔性桥梁,能够跨越时间节奏的差异,精准度量时间序列间的相似性,在众多领域发挥着不可或缺的作用。一、D
- 【干货】深度解析个人IP打造:从定位到变现的全维度运营指南
老蒋新思维
创始人IP
在短视频浪潮席卷的当下,越来越多人意识到“个人IP”的商业价值。但许多人将其简单等同于“真人出镜发内容”或“企业找员工代言”,这种浅层认知往往导致运营陷入瓶颈。事实上,打造个人IP是一项系统性工程,需要从定位、内容、平台到商业体系的全链条规划。本文将聚焦「定位」这一核心基石,结合实战案例与趋势洞察,为创业者提供可落地的操作框架。一、定位本质:构建差异化价值坐标个人IP的定位绝非“选赛道”这么简单,
- Vue 3 中使用路由参数跳转时 watch 触发重复请求问题详解
Vue3中使用路由参数跳转时watch触发重复请求问题详解收藏+点赞+关注,掌握Vue3路由参数监听中的隐藏陷阱,避免详情页、嵌套路由页误触发重复请求!一、问题背景在Vue3项目中,常见需求是:在列表页点击跳转到详情页,传递id参数详情页通过watch(()=>route.query.id)监听路由变化,自动请求数据例如:watch(()=>route.query.id,(newId)=>{fet
- Django5.1(91)—— 如何删除一个 Django 应用
小天的铁蛋儿
djangoPythondjangopython后端
如何删除一个Django应用Django提供了将一组功能组织成名为应用程序的Python包的能力。当需求发生变化时,应用程序可能会变得过时或不再需要。以下步骤将帮助你安全地删除一个应用程序。删除所有与该应用程序相关的引用(导入、外键等)。从相应的models.py文件中删除所有模型。通过运行makemigrations来创建相关的迁移。这一步会生成一个迁移,用于删除已删除模型的表,以及与这些模型相
- Linux: perf: debug问题一例,cpu使用率上升大约2%;多线程如何细化cpu及perf数据分析
mzhan017
kernel系统性能linux服务器网络
文章目录前提面临的问题内核级别函数的差别继续debug总结根据pid前提一个进程安置在一个CPU上,新功能上线之后,固定量的业务打起来,占用的CPU是42%。之前没有新功能的情况下,CPU占用是40%。差了大约2%。而且这个进程里的线程数非常多,有50多个线程。从差距看变化不大,没有别的办法,只能使用perf来抓取数据来看。但是使用perf也要面临很多的问题。面临的问题面临的问题有一堆:两次per
- Python之聚合函数
_AndyLau
手把手学pythonpython
Python聚合函数文章目录Python聚合函数聚合函数使用多个聚合函数结合`annotate`和`values`进行分组聚合注意事项F表达式和Q表达式F表达式Q表达式注意事项视图HTML中的表单概述Django中表单概述ModelForm关键点使用示例创建ModelForm在视图中使用ModelForm模板总结Cookie和SessionDjango中的Cookie操作Django中的Sessi
- Vue - 深入理解 Vue 3 中的 ref 和 reactive
深入理解Vue3中的ref和reactiveVue3引入了一个全新的响应式系统,使得数据的变化能够自动反映到视图中。对于开发者来说,理解如何使用响应式API是掌握Vue3的关键。本文将重点介绍Vue3中的两个核心响应式API—ref和reactive,并理解它们的区别、使用场景以及如何在实际项目中有效利用它们。1.Vue3响应式系统概述Vue3的响应式系统基于Proxy对象实现,它使得我们可以轻松
- VUE2双向绑定的原理
许先森森
VUE2javascript前端vue.jsvue双向绑定vue
文章目录VUE2双向绑定的原理1.什么是双向绑定2.双向绑定的原理2.1ViewModel的重要作用2.2双向绑定的流程3.双向绑定的实现3.1data响应化处理3.2Compile编译3.3依赖收集VUE2双向绑定的原理1.什么是双向绑定讲双向绑定先讲单项绑定,啥叫单项绑定,就是一句话就是通过Model去改变View,再直白点,就是通过js代码把数据改变后,html视图也跟着变化那双项绑定就很好
- ABC363 题解
ABC363题解A-PilingUp(模拟)题意:输入一个数字,数字介于111-999999显示了一次^,100100100-199199199显示了^两次…增加显示的所需的最小的评分增幅分析:算比次数字大且为100100100的倍数的最小值减此数字代码:voidsolve(){intn;cin>>n;intt=n/100;cout<<(t+1)*100-n;}B-JapaneseCursedDo
- Oracle基础包之DBMS_LOGMNR(十二)
夜未央,流年殇
Oracle数据库oracledbms
概述通过使用包DBMS_LOGMNR和DBMS_LOGMNR_D,可以分析重做日志和归档日志所记载的事务变化,最终确定误操作(例如DROPTABLE)的时间,跟踪用户事务操作,跟踪并还原表的DML操作.回到顶部包的组成dbms_logmnr.add_logfile作用:用于为日志分析列表增加或删除日志文件,或者建立日志分析列表。语法:dbms_logmnr.add_logfile(LogFileN
- Linux与Windows切换使用Obsidian,出现 unexplained changes 问题的解决
CS-Polaris
业务能力技术栈git
如果你的Obsidian文档在Linux与Windows间来回切换,可能会涉及到文件的保存换行符问题,但这样的话就容易导致一个问题,那就是内容无差异,Obsidian却提示unexplainedchanges,Windows系统下的解决方法如下,找到.git/config文件,配置[core]bare=falseautocrlf=falsetrustctime=false一、参考文章或视频链接[1
- CentOS-7的“ifupdown“与Debian的“ifupdown“对比 笔记250706
kfepiza
OS操作系统WindowsLinux等#控制台命令行Shellbashcmd等网络通讯传输协议IPTCPUDP物联centosdebian笔记linux网络
CentOS-7的"ifupdown"与Debian的"ifupdown"对比笔记250706CentOS7和Debian的ifupdown工具名称相同,但在实现机制、配置文件语法和系统集成上存在显著差异。以下是核心对比分析:⚙️一、核心差异概览对比维度CentOS7Debian工具定位network-scripts套件的一部分,依赖传统ifcfg文件独立包(ifupdown),使用/etc/ne
- React与Vue的区别?
扎西_德勒
vue.jsreact.jsjavascript
一、区别:1.语法Vue采用自己特有的模板语法;React是单向的,采用jsx语法创建react元素。2.监听数据变化的实现原理不同Vue2.0通过Object.defineproperty()方法的getter/setter属性,实现数据劫持,每次修改完数据会触发diff算法(双端对比)React默认是通过shouldComponentUpdata生命周期来决定是否需要渲染更新,再触发它的dif
- 代码随想录算法训练营第十一天
天天开心(∩_∩)
算法
LeetCode.150逆波兰表达式求值题目链接逆波兰表达式求值题解classSolution{publicintevalRPN(String[]tokens){Stackcstack=newStackset=newHashSetdeque=newLinkedListdeque.getLast()){deque.removeLast();}deque.add(val);}intpeek(){ret
- knob UI插件使用
换个号韩国红果果
JavaScriptjsonpknob
图形是用canvas绘制的
js代码
var paras = {
max:800,
min:100,
skin:'tron',//button type
thickness:.3,//button width
width:'200',//define canvas width.,canvas height
displayInput:'tr
- Android+Jquery Mobile学习系列(5)-SQLite数据库
白糖_
JQuery Mobile
目录导航
SQLite是轻量级的、嵌入式的、关系型数据库,目前已经在iPhone、Android等手机系统中使用,SQLite可移植性好,很容易使用,很小,高效而且可靠。
因为Android已经集成了SQLite,所以开发人员无需引入任何JAR包,而且Android也针对SQLite封装了专属的API,调用起来非常快捷方便。
我也是第一次接触S
- impala-2.1.2-CDH5.3.2
dayutianfei
impala
最近在整理impala编译的东西,简单记录几个要点:
根据官网的信息(https://github.com/cloudera/Impala/wiki/How-to-build-Impala):
1. 首次编译impala,推荐使用命令:
${IMPALA_HOME}/buildall.sh -skiptests -build_shared_libs -format
2.仅编译BE
${I
- 求二进制数中1的个数
周凡杨
java算法二进制
解法一:
对于一个正整数如果是偶数,该数的二进制数的最后一位是 0 ,反之若是奇数,则该数的二进制数的最后一位是 1 。因此,可以考虑利用位移、判断奇偶来实现。
public int bitCount(int x){
int count = 0;
while(x!=0){
if(x%2!=0){ /
- spring中hibernate及事务配置
g21121
Hibernate
hibernate的sessionFactory配置:
<!-- hibernate sessionFactory配置 -->
<bean id="sessionFactory"
class="org.springframework.orm.hibernate3.LocalSessionFactoryBean">
<
- log4j.properties 使用
510888780
log4j
log4j.properties 使用
一.参数意义说明
输出级别的种类
ERROR、WARN、INFO、DEBUG
ERROR 为严重错误 主要是程序的错误
WARN 为一般警告,比如session丢失
INFO 为一般要显示的信息,比如登录登出
DEBUG 为程序的调试信息
配置日志信息输出目的地
log4j.appender.appenderName = fully.qua
- Spring mvc-jfreeChart柱图(2)
布衣凌宇
jfreechart
上一篇中生成的图是静态的,这篇将按条件进行搜索,并统计成图表,左面为统计图,右面显示搜索出的结果。
第一步:导包
第二步;配置web.xml(上一篇有代码)
建BarRenderer类用于柱子颜色
import java.awt.Color;
import java.awt.Paint;
import org.jfree.chart.renderer.category.BarR
- 我的spring学习笔记14-容器扩展点之PropertyPlaceholderConfigurer
aijuans
Spring3
PropertyPlaceholderConfigurer是个bean工厂后置处理器的实现,也就是BeanFactoryPostProcessor接口的一个实现。关于BeanFactoryPostProcessor和BeanPostProcessor类似。我会在其他地方介绍。
PropertyPlaceholderConfigurer可以将上下文(配置文件)中的属性值放在另一个单独的标准java
- maven 之 cobertura 简单使用
antlove
maventestunitcoberturareport
1. 创建一个maven项目
2. 创建com.CoberturaStart.java
package com;
public class CoberturaStart {
public void helloEveryone(){
System.out.println("=================================================
- 程序的执行顺序
百合不是茶
JAVA执行顺序
刚在看java核心技术时发现对java的执行顺序不是很明白了,百度一下也没有找到适合自己的资料,所以就简单的回顾一下吧
代码如下;
经典的程序执行面试题
//关于程序执行的顺序
//例如:
//定义一个基类
public class A(){
public A(
- 设置session失效的几种方法
bijian1013
web.xmlsession失效监听器
在系统登录后,都会设置一个当前session失效的时间,以确保在用户长时间不与服务器交互,自动退出登录,销毁session。具体设置很简单,方法有三种:(1)在主页面或者公共页面中加入:session.setMaxInactiveInterval(900);参数900单位是秒,即在没有活动15分钟后,session将失效。这里要注意这个session设置的时间是根据服务器来计算的,而不是客户端。所
- java jvm常用命令工具
bijian1013
javajvm
一.概述
程序运行中经常会遇到各种问题,定位问题时通常需要综合各种信息,如系统日志、堆dump文件、线程dump文件、GC日志等。通过虚拟机监控和诊断工具可以帮忙我们快速获取、分析需要的数据,进而提高问题解决速度。 本文将介绍虚拟机常用监控和问题诊断命令工具的使用方法,主要包含以下工具:
&nbs
- 【Spring框架一】Spring常用注解之Autowired和Resource注解
bit1129
Spring常用注解
Spring自从2.0引入注解的方式取代XML配置的方式来做IOC之后,对Spring一些常用注解的含义行为一直处于比较模糊的状态,写几篇总结下Spring常用的注解。本篇包含的注解有如下几个:
Autowired
Resource
Component
Service
Controller
Transactional
根据它们的功能、目的,可以分为三组,Autow
- mysql 操作遇到safe update mode问题
bitray
update
我并不知道出现这个问题的实际原理,只是通过其他朋友的博客,文章得知的一个解决方案,目前先记录一个解决方法,未来要是真了解以后,还会继续补全.
在mysql5中有一个safe update mode,这个模式让sql操作更加安全,据说要求有where条件,防止全表更新操作.如果必须要进行全表操作,我们可以执行
SET
- nginx_perl试用
ronin47
nginx_perl试用
因为空闲时间比较多,所以在CPAN上乱翻,看到了nginx_perl这个项目(原名Nginx::Engine),现在托管在github.com上。地址见:https://github.com/zzzcpan/nginx-perl
这个模块的目的,是在nginx内置官方perl模块的基础上,实现一系列异步非阻塞的api。用connector/writer/reader完成类似proxy的功能(这里
- java-63-在字符串中删除特定的字符
bylijinnan
java
public class DeleteSpecificChars {
/**
* Q 63 在字符串中删除特定的字符
* 输入两个字符串,从第一字符串中删除第二个字符串中所有的字符。
* 例如,输入”They are students.”和”aeiou”,则删除之后的第一个字符串变成”Thy r stdnts.”
*/
public static voi
- EffectiveJava--创建和销毁对象
ccii
创建和销毁对象
本章内容:
1. 考虑用静态工厂方法代替构造器
2. 遇到多个构造器参数时要考虑用构建器(Builder模式)
3. 用私有构造器或者枚举类型强化Singleton属性
4. 通过私有构造器强化不可实例化的能力
5. 避免创建不必要的对象
6. 消除过期的对象引用
7. 避免使用终结方法
1. 考虑用静态工厂方法代替构造器
类可以通过
- [宇宙时代]四边形理论与光速飞行
comsci
从四边形理论来推论 为什么光子飞船必须获得星光信号才能够进行光速飞行?
一组星体组成星座 向空间辐射一组由复杂星光信号组成的辐射频带,按照四边形-频率假说 一组频率就代表一个时空的入口
那么这种由星光信号组成的辐射频带就代表由这些星体所控制的时空通道,该时空通道在三维空间的投影是一
- ubuntu server下python脚本迁移数据
cywhoyi
pythonKettlepymysqlcx_Oracleubuntu server
因为是在Ubuntu下,所以安装python、pip、pymysql等都极其方便,sudo apt-get install pymysql,
但是在安装cx_Oracle(连接oracle的模块)出现许多问题,查阅相关资料,发现这边文章能够帮我解决,希望大家少走点弯路。http://www.tbdazhe.com/archives/602
1.安装python
2.安装pip、pymysql
- Ajax正确但是请求不到值解决方案
dashuaifu
Ajaxasync
Ajax正确但是请求不到值解决方案
解决方案:1 . async: false , 2. 设置延时执行js里的ajax或者延时后台java方法!!!!!!!
例如:
$.ajax({ &
- windows安装配置php+memcached
dcj3sjt126com
PHPInstallmemcache
Windows下Memcached的安装配置方法
1、将第一个包解压放某个盘下面,比如在c:\memcached。
2、在终端(也即cmd命令界面)下输入 'c:\memcached\memcached.exe -d install' 安装。
3、再输入: 'c:\memcached\memcached.exe -d start' 启动。(需要注意的: 以后memcached将作为windo
- iOS开发学习路径的一些建议
dcj3sjt126com
ios
iOS论坛里有朋友要求回答帖子,帖子的标题是: 想学IOS开发高阶一点的东西,从何开始,然后我吧啦吧啦回答写了很多。既然敲了那么多字,我就把我写的回复也贴到博客里来分享,希望能对大家有帮助。欢迎大家也到帖子里讨论和分享,地址:http://bbs.csdn.net/topics/390920759
下面是我回复的内容:
结合自己情况聊下iOS学习建议,
- Javascript闭包概念
fanfanlovey
JavaScript闭包
1.参考资料
http://www.jb51.net/article/24101.htm
http://blog.csdn.net/yn49782026/article/details/8549462
2.内容概述
要理解闭包,首先需要理解变量作用域问题
内部函数可以饮用外面全局变量
var n=999;
functio
- yum安装mysql5.6
haisheng
mysql
1、安装http://dev.mysql.com/get/mysql-community-release-el7-5.noarch.rpm
2、yum install mysql
3、yum install mysql-server
4、vi /etc/my.cnf 添加character_set_server=utf8
- po/bo/vo/dao/pojo的详介
IT_zhlp80
javaBOVODAOPOJOpo
JAVA几种对象的解释
PO:persistant object持久对象,可以看成是与数据库中的表相映射的java对象。最简单的PO就是对应数据库中某个表中的一条记录,多个记录可以用PO的集合。PO中应该不包含任何对数据库的操作.
VO:value object值对象。通常用于业务层之间的数据传递,和PO一样也是仅仅包含数据而已。但应是抽象出的业务对象,可
- java设计模式
kerryg
java设计模式
设计模式的分类:
一、 设计模式总体分为三大类:
1、创建型模式(5种):工厂方法模式,抽象工厂模式,单例模式,建造者模式,原型模式。
2、结构型模式(7种):适配器模式,装饰器模式,代理模式,外观模式,桥接模式,组合模式,享元模式。
3、行为型模式(11种):策略模式,模版方法模式,观察者模式,迭代子模式,责任链模式,命令模式,备忘录模式,状态模式,访问者
- [1]CXF3.1整合Spring开发webservice——helloworld篇
木头.java
springwebserviceCXF
Spring 版本3.2.10
CXF 版本3.1.1
项目采用MAVEN组织依赖jar
我这里是有parent的pom,为了简洁明了,我直接把所有的依赖都列一起了,所以都没version,反正上面已经写了版本
<project xmlns="http://maven.apache.org/POM/4.0.0" xmlns:xsi="ht
- Google 工程师亲授:菜鸟开发者一定要投资的十大目标
qindongliang1922
工作感悟人生
身为软件开发者,有什么是一定得投资的? Google 软件工程师 Emanuel Saringan 整理了十项他认为必要的投资,第一项就是身体健康,英文与数学也都是必备能力吗?来看看他怎么说。(以下文字以作者第一人称撰写)) 你的健康 无疑地,软件开发者是世界上最久坐不动的职业之一。 每天连坐八到十六小时,休息时间只有一点点,绝对会让你的鲔鱼肚肆无忌惮的生长。肥胖容易扩大罹患其他疾病的风险,
- linux打开最大文件数量1,048,576
tianzhihehe
clinux
File descriptors are represented by the C int type. Not using a special type is often considered odd, but is, historically, the Unix way. Each Linux process has a maximum number of files th
- java语言中PO、VO、DAO、BO、POJO几种对象的解释
衞酆夼
javaVOBOPOJOpo
PO:persistant object持久对象
最形象的理解就是一个PO就是数据库中的一条记录。好处是可以把一条记录作为一个对象处理,可以方便的转为其它对象。可以看成是与数据库中的表相映射的java对象。最简单的PO就是对应数据库中某个表中的一条记录,多个记录可以用PO的集合。PO中应该不包含任何对数据库的操作。
BO:business object业务对象
封装业务逻辑的java对象