libsvm安装使用及网格搜索法参数寻优
LIBSVM 是台湾大学林智仁( Chih-Jen Lin)教授开发的。
说明:本教程仅针对电脑为
64位的计算机,如果是32位的计算机需要下载C语言编辑器进行手动编译。
1.下载libsvm
①下载地址在其官网:https://www.csie.ntu.edu.tw/~cjlin/libsvm/
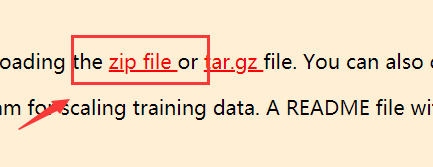
注意:这里需要右击“在新标签页中打开链接”
2.安装libsvm
① 将解压好的文件夹粘贴到Matlab的安装路径下的toolbox中
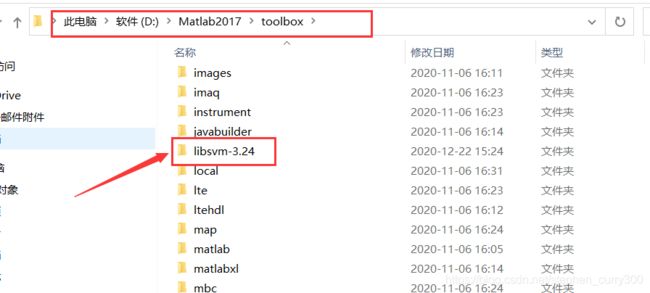
② 打开matlab设置工具箱

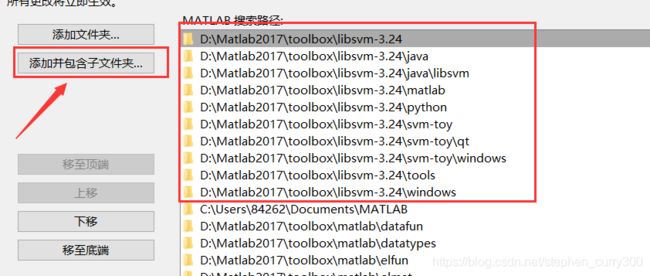
③添加工具箱文件
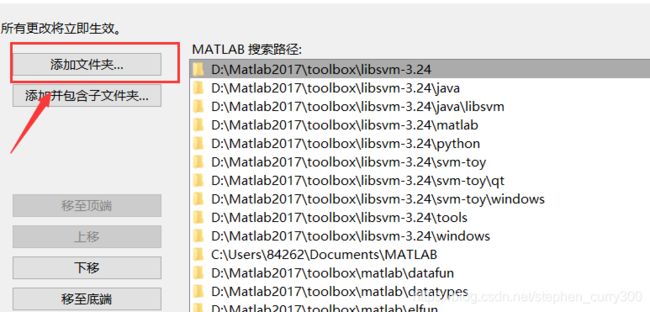
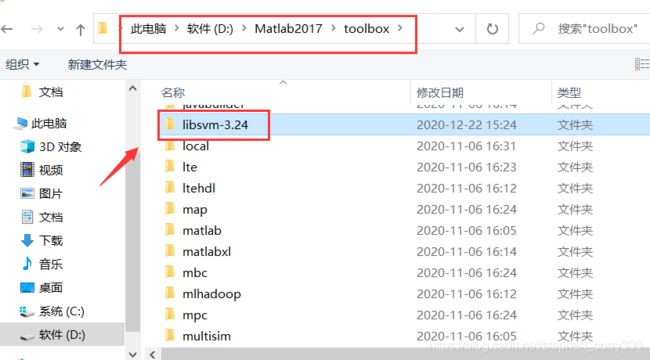
选择“添加并包含子文件夹”,选择刚才相同的libsvm-3.24
3.验证是否安装成功
下载测试数据集:https://share.weiyun.com/5LGqTGa
创建脚本输入:
clc;clear;
load heart_scale.mat
model = svmtrain(heart_scale_label,heart_scale_inst, '-c 1 -g 0.07');
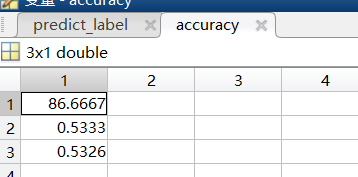
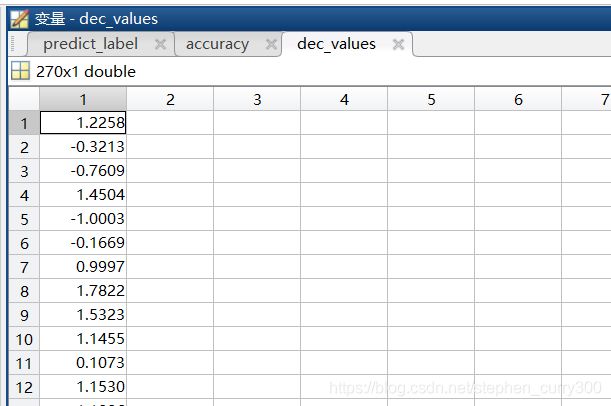
[predict_label, accuracy, dec_values] = svmpredict(heart_scale_label, heart_scale_inst, model);
4.libsvm参数说明
这里首先推荐大家看一下官方给的参考文档:https://www.csie.ntu.edu.tw/~cjlin/papers/guide/guide.pdf
如果英文文档看着不舒服,丢到网易有道翻译器中翻译下
这里参考唱一半的歌歌转载的文章进行说明:
4.1训练参数
model = libsvmtrain(training_label, training_label ['libsvm_options']);
training_label:训练集的标签y
training_data:训练集数据x
可选参数libsvm_options选项如下:
-s svm类型:SVM设置类型(默认0)
0 — C-SVC; 1 –v-SVC; 2 – 一类SVM; 3 — e-SVR; 4 — v-SVR
-t 核函数类型:核函数设置类型(默认2)
0 – 线性核函数
1 – 多项式核函数poly
2 – RBF(径向基)核函数
3 – sigmoid核函数
-d degree:核函数中的degree设置(针对多项式核函数)(默认3)
-g r(gamma):核函数中的gamma函数设置(针对多项式/rbf/sigmoid核函数)(默认1/k,k为总类别数)
-r coef0:核函数中的coef0设置(针对多项式/sigmoid核函数)((默认0)
-c cost:设置C-SVC,e -SVR和v-SVR的参数(损失函数)(默认1)
-n nu:设置v-SVC,一类SVM和v- SVR的参数(默认0.5)
-p p:设置e -SVR 中损失函数p的值(默认0.1)
-m cachesize:设置cache内存大小,以MB为单位(默认40)
-e eps:设置允许的终止判据(默认0.001)
-h shrinking:是否使用启发式,0或1(默认1)
-wi weight:设置第几类的参数C为weight*C (C-SVC中的C) (默认1)
-v n: n-fold交互检验模式,n为fold的个数,必须大于等于2
4.2 训练返回参数
-Parameters: 一个5 x 1的矩阵,从上到下依次表示:
-s SVM类型(默认0);
-t 核函数类型(默认2)
-d 核函数中的degree设置(针对多项式核函数)(默认3);
-g 核函数中的r(gamma)函数设置(针对多项式/rbf/sigmoid核函数) (默认类别数目的倒数);
-r 核函数中的coef0设置(针对多项式/sigmoid核函数)((默认0)
-nr_class: 表示数据集中有多少类别,比如二分类时这个值即为2。
-totalSV: 表示支持向量的总数。
-rho: 决策函数wx+b中的常数项的相反数(-b)。
-Label: 表示数据集中类别的标签,比如二分类常见的1和-1。
-ProbA: 使用-b参数时用于概率估计的数值,否则为空。
-ProbB: 使用-b参数时用于概率估计的数值,否则为空。
-nSV: 表示每类样本的支持向量的数目,和Label的类别标签对应。如Label=[1; -1],nSV=[63; 67],则标签为1的样本有63个支持向量,标签为-1的有67个。
-sv_coef: 表示每个支持向量在决策函数中的系数。
-SVs: 表示所有的支持向量,如果特征是n维的,支持向量一共有m个,则为m x n的稀疏矩阵。
4.3 预测参数
[predict_label, accuracy, dec_values] = svmpredict(test_label, test_data, model);
test_label:验证集的标签y
test_data:验证集数据x
4.4 预测返回的参数
-predicted_label:第一个返回值,表示样本的预测类标号。
-accuracy:第二个返回值,一个3 x 1的数组,表示分类的正确率、回归的均方根误差、回归的平方相关系数。
-decision_values/prob_estimates:第三个返回值,一个矩阵包含决策的值或者概率估计。对于n个预测样本、k类的问题,如果指定“-b 1”参数,则n x k的矩阵,每一行表示这个样本分别属于每一个类别的概率;如果没有指定“-b 1”参数,则为n x k*(k-1)/2的矩阵,每一行表示k(k-1)/2个二分类SVM的预测结果。
5.参数寻优
官方大大给了我们明确的提示,可以利用python和gunplot来进行参数寻优~

翻译如下:
我们现在为不了解SVM的用户提供了一个简单的脚本(easy.py)。它使一切变得自动化-从数据缩放到参数选择。
参数选择工具grid.py生成以下交叉验证精度轮廓。要使用此工具,您还需要安装python 和 gnuplot。
5.1 下载python和gnuplot
下载python过程略
下载gnuplot:
两种方式:
①官网下载(速度贼慢,外网你懂的)

②某盘链接提供给你们,就是不是最新版本的gnuplot
链接:https://pan.baidu.com/s/1VE72XqOErGFQqJeuI3lIug
提取码:wel5
5.2 安装gnuplot
找到下载好的安装包进行安装(可以默认路径,但是你一定要记得)
一般软件安装的默认路径为:C;\ProgramFiles\gnuplot
5.3 测试寻优过程
①找到自己装的libsvm路径下的tools文件夹下的grid.py
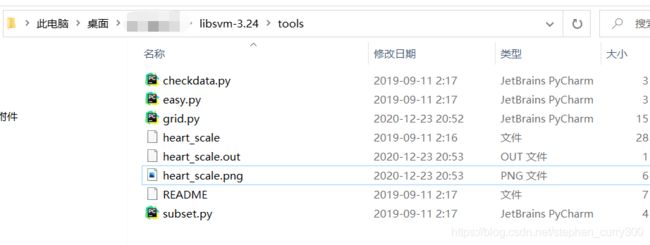
②打开grid.py(这里我是使用的Pycharm2020)
修改两个地方:
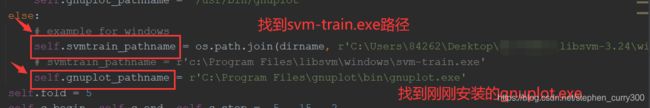
- self.svmtrain_pathname
在你的libsvm-3.24文件下windows文件夹下游svm-train.exe
将他的路径复制到self.svmtrain_pathname后面
- self.gnuplot_pathname
在你刚刚安装的gnuplot文件下的bin目录下会有gnuplot.exe
将他的路径复制到self.gnuplot_pathname后面
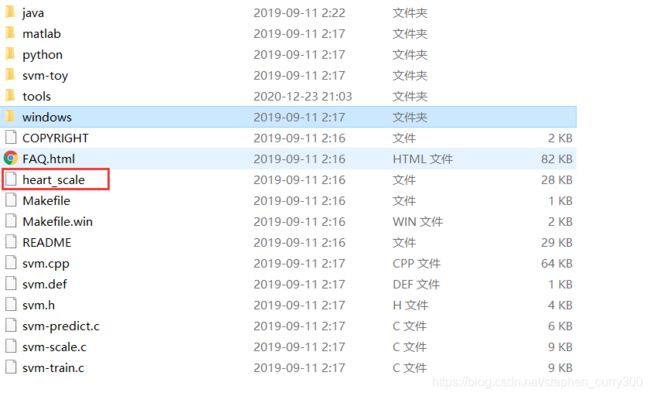
③将libsvm-3.24文件夹自带的测试案例heart-scale复制到与grid.py同级目录下(即tools文件夹下)
④打开cmd命令控制窗口,进入grid.py文件目录下
输入命令:
python grid.py heart_scale
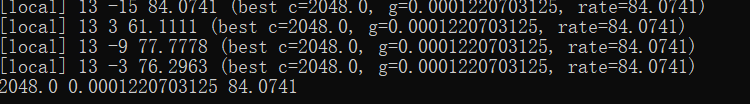
结果为:C=2048.0;γ=0.0001220703125;交叉验证精度CV Rate=84.0741%
⑤打开刚刚的tools文件,会发现多出来两个文件

一个是寻优过程,一个是寻优图像
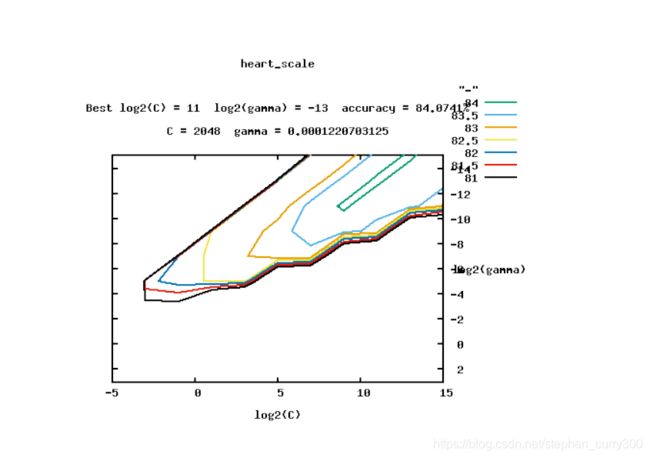
当然如果是你熟悉python代码,可以它的源码库进行代码修改,把图片的标题,坐标轴改成你喜欢的样式~
5.4 自己的数据文件寻优
同样的方法,将数据文件放在grid.py文件夹下,然后在cmd中执行
python grid.py xxx(文件名)即可
另外,如果不知道怎么将数据格式转化为libsvm格式的文件,见第六章
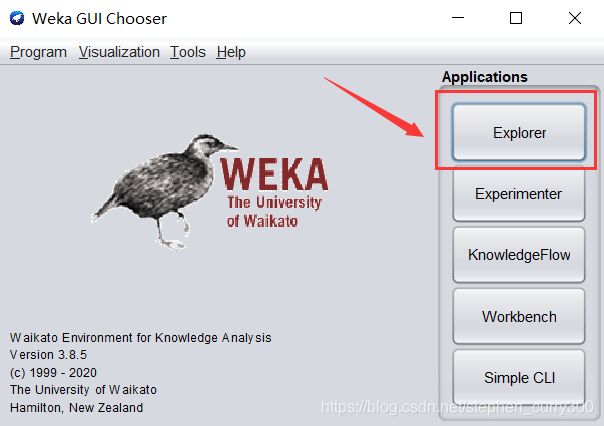
6.Weka软件的使用
Weka的全名是怀卡托智能分析环境(Waikato Environment for Knowledge Analysis),是一款免费的,非商业化(与之对应的是SPSS公司商业数据挖掘产品–Clementine )的,基于JAVA环境下开源的机器学习(machine learning)以及数据挖掘(data mining)软件。【摘自百度百科】
6.1 weka下载
其官网位置:https://www.cs.waikato.ac.nz/ml/weka/
① 点击Download

② 选择计算机操作系统进行下载
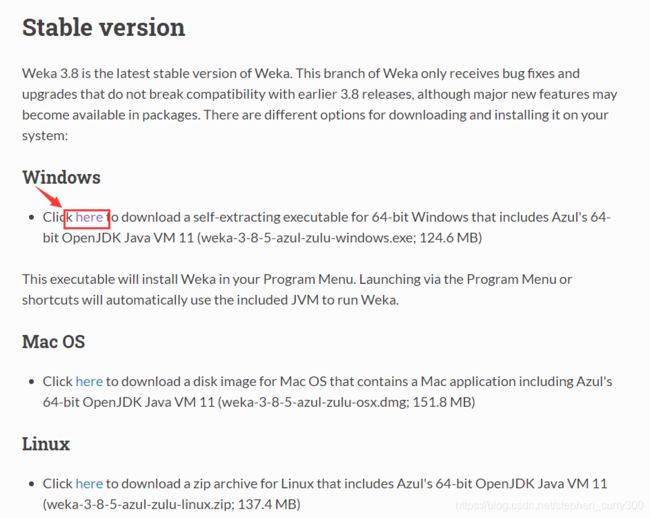
③ 会自动跳转到下载页进行下载
(这是下载结束后的)
6.2 weka软件的安装
安装非常简单,选择你的安装目录进行“下一步安装大法”就行。
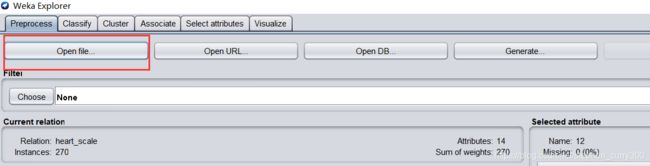
6.3 数据文件转libsvm
- 最后一列为标签列
- 最后一列前面的都是数据维度
- 第一行需要加索引序号
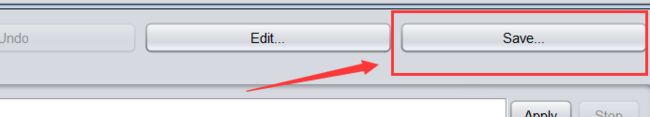
将xlsx的excel文件保存为csv格式
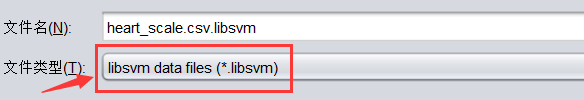
③ 保存libsvm格式的文件