i6mA-DNC:基于深度学习的二核苷酸表示预测水稻基因组DNA n6 -甲基腺苷位点
i6mA-DNC:基于深度学习的二核苷酸表示预测水稻基因组DNA n6 -甲基腺苷位点
- 摘要
- 一、简介
- 二、材料和方法
-
- 1.基准数据集
- 2.提出的模型
- 3.绩效评估
-
- 3.1交叉验证
- 三、结果与讨论
- 四、预测器模型的web服务器
摘要
DNA甲基化是一个重要的表观遗传过程。DNA n6 -甲基腺嘌呤与DNA复制、转录、修复和细胞防御等多种生物过程密切相关。在基因组中,N6甲基腺嘌呤 (6mA)位点不均匀分布;因此,为了更好地理解其生物学功能,需要确定6mA的基因组位置。虽然各种实验程序已被用于鉴定6mA位点并产生阳性结果,这些生化技术是昂贵和费时的。为了解决这一问题并为今后的研究提供便利,必须建立一个稳健、准确的n6 -甲腺嘌呤位点的计算模型。为此,我们引入了一种基于深度学习的计算模型i6mA-DNC来检测水稻基因组中的n6 -甲基烯胺位点。我们将DNA序列分解成二核苷酸成分,并将它们输入模型。该模型使用卷积神经网络(CNN)从预处理数据中自动提取最优特征。我们提出的模型i6mA-DNC获得了89.20%的特异性、88.01%的敏感性、88.60%的准确性和0.772的MCC。这些结果证明我们的智能模型在所有评价指标上都比现有的方法取得了更好的成功率。我们的模型i6mA-DNC有望成为n6 -甲腺嘌呤位点识别的学术研究的有用工具。
一、简介
DNA甲基化是指一个重要的表观遗传过程,其中甲基被添加到DNA碱基。它经常在不改变序列的情况下改变基因的活性。在DNA的四个碱基中,胞嘧啶和腺嘌呤碱基可以被甲基化。在真核生物中,5-甲基胞嘧啶(5 mC)是最常见的DNA修饰类型。然而,在原核生物中,6-甲腺嘌呤(6ma)是最主要的DNA修饰。DNA n6 -甲基腺嘌呤(6 mA)作为一种非经典的DNA修饰,已在细菌、古细菌和真核生物三界中被检测到。在基因组中,6ma位点的分布并不均匀。因此,为了充分了解6ma的生物学功能,准确地确定其基因组位置是十分必要的。
我们要利用已有的实验数据设计一个强大的、精确的、快速的计算模型来检测n6 -甲基腺嘌呤位点。
为了确定DNAN6甲基腺嘌呤位点,提出了利用基于卷积神经网络(CNN)的二核苷酸组成来鉴定6mA的i6mA-DNC模型。将原始DNA序列划分为二核苷酸成分,并将其输入到CNN模型中。与现有的深度学习模型相比,我们提出的新型深度学习模型具有更好的预测效果。
二、材料和方法
1.基准数据集
首先,我们选择一个有效的基准数据集,对我们提出的模型进行有效的预测和测试。所有样本有41个核苷酸,中心位置为6mA。因此,数据集表示为:
![]()
其中R表示包含1760个样本的整个数据集。R+是880个带有6mA位点的序列组成,R-由880个带有非6ma位点序列组成。U表示两个子集的并集。
2.提出的模型

图一给出了模型的框架。该模型利用卷积神经网络(CNN)对6mA位点进行预测。
首先,该模型只输入一个DNA样本。给出的序列S可以表示为S={N1,N2…Ni},这里N1表示序列的第一个核苷酸,N2表示第二个核苷酸,以此类推。每个核苷酸都属于这四种核苷酸中的一种:A;C;G;T.。i =41表示样本的长度。
将原始基因组序列转换为深度学习可以识别的载体形式是很重要的。在将序列数据转换为矢量形式时,也需要考虑模式序列信息的丢失,因为这主要发生在离散模型中。为了避免模式丢失,许多技术被引入,包括伪氨基酸组成(pseudo amino acid composition, PseAAC),它在蛋白质组学中得到了广泛的应用。
为了将DNA序列转化为载体形式,该模型将所有序列都进行划分,它是通过在序列之间滑动大小为2的窗口来实现的。例如,序列AGTTCA将导致以下的二聚体的子序列:AG, GT, TT, TC, CA。因为序列长度为L的序列有L-K+1的k聚体,所有长度为41的样本产生40个二聚类的成分。然后,这些2聚类成分被嵌入16维向量空间,因为所有四个单个核苷酸(A、C、G和T)可以相互组合,产生4²=16种可能的双核苷酸组合。如图1所示,AA, AC, TT,例如,分别被表示为(1,0,0,0,0,0,0,0,0,0,0,0,0,0,0,0],[0,1,0,0,0,0,0,0,0,0,0,0,0,0,0,0),和(0,0,0,0,0,0,0,0,0,0,0,0,0,0,0,1]。
所提出的模型将DNA样本转换成具有2聚类表示的矢量形式后,将其输入CNN去检测6个mA位点(CNN,即卷积神经网络是一种广泛应用于生物信息学各个研究领域的方法。在训练过程中,CNN自动从输入数据中提取出主要特征。link)
卷积神经网络由卷积层、池化层、ReLU层、全连通层等层组成。每一层都包含不同的超参数,需要在学习时进行调整。基于网格搜索方法,选择各层超参数的最佳组合。调优的超参数是卷积层的个数,卷积层中的过滤器个数,卷积层中的过滤器大小,卷积层后的dropout值。表1为模型的超参数选择。

表2显示了所提议的模型的具体配置。我们提出的模型使用了Keras框架。 link
link。Conv1D(f,s)中f是一维卷积层过滤器的数量、s是过滤器的大小。每个卷积层后面是一个非线性激活函数,称为矫正线性单元(ReLU)。dropout§用作算子的概率p防止模型过度拟合。Maxpool1D (m,d)降低维度通过选择窗口内的最大值m, m是池大小和d是步幅。至于Dense(n),n代表一个完全连接层中的节点数量。最后一个是乙状结肠函数,它的输出值在0和1之间。我们用这一层来预测输入序列是否有m6A位点。
3.绩效评估
为了评估训练模型的成功率,我们选择并进行了适当的交叉验证。预测系统的性能计算通常采用四个指标。分别是准确度(ACC)、Mathew’s correlation coefficient (MCC)、特异性(Sp)和敏感性(Sn)。

3.1交叉验证
选择一种精确的交叉验证方法也是评估模型性能的重要部分。有三种基本的交叉验证方法:独立数据集检验、k-fold交叉验证和折刀检验。其中,k-fold交叉验证是最健壮的用于预测器性能评价的方法。在这项工作中,我们使用了10倍交叉验证,通过将整个数据集随机分为10个分区,10个子集的大小大约相等。使用单个子集作为测试数据集来评估所提出的模型,而保留剩下的9个子集作为训练数据集。交叉验证过程重复10次,每个子集使用一次作为测试数据。然后我们计算所有折叠的10个结果的平均值。
三、结果与讨论
在这一部分,我们通过k-fold交叉验证测试,讨论了使用基准数据集实现所提出的模型。表3显示了i6mA-DNC模型与现有最先进的模型6 mA-Pred、iDNA6mA(5步规则)和iN6-methylat(5步)在水稻基因组中鉴定n6 -甲基腺苷位点的成功率比较。
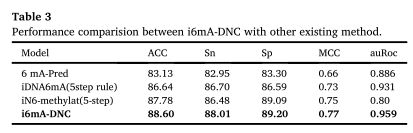
该模型获得0.77的MCC,比iN6methylat(5步)模型增加2%。准确度为86.60,比最新方法提高1.18%。Sp、Sn依次为89.20和88.01,分别比iN6methylat(5步)增加0.11%和1.5% 3。
接受者工作特征曲线下面积(auROC)用于评价分类器的性能质量。图2为i6mA-DNC的auROC值,为95.93。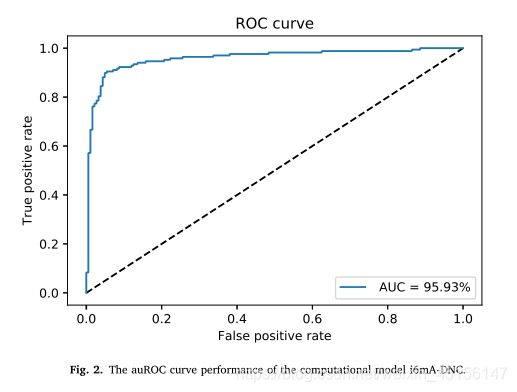
图3是四种预测系统:i6mA-DNC、iN6-methylat(5步)、iDNA6mA和i6mA-Pred。从图中可以看出,我们提出的模型得到了最好的结果。

图4显示了i6mA-DNC的混淆矩阵性能的可视化。
为了进行综合评价,我们尝试在一个独立的数据集上对模型进行测试。在这个数据集中,我们选择了关联修改分数(ModQV)大于30的序列,它们与基准数据集中的序列相似度小于60%。结果,我们获得221个6 mA阳性序列。另一方面,选择了221条以A为中心的阴性序列,但未被SMRT-seq检测到。Acc、Sn、Sp、MCC分别达到88.64%、89.09%、88.18%、77.28%。

link.
最后,我们在基准数据集中对核苷酸进行计算突变,并研究突变对最终预测结果的影响。例如;对于每个长度为L的序列,我们有L x 3个可能的突变。对于每一个突变,我们都保留了绝对差异。然后,我们对基准数据集中由于整个序列的突变而产生的预测得分修改取平均值。从图5的热图可以看出,序列中心的突变比序列两边的突变对最终预测结果的影响更大。

四、预测器模型的web服务器
最后一步是为所提出的模型开发一个易于访问的web服务器。许多研究人员在他们的出版物中采用了这一步骤,以使每个人,特别是生物学家,能够操作一个友好的计算预测模型。由于这些面向web的计算工具,生物信息学对医学科学的影响大大提高了。为了方便起见,还在 link.上建立了i6mA-DNC predictor的web服务器。