R语言实战-第十七章 R in action-chapter17
本章目的:基于一组预测变量预测一个分类结果
(如:根据关键词、图像、来源等判断一份邮件是否是病毒邮件)
本章用到的package:
#rpart rpart.plot party 实现决策树模型及其可视化
#randomForest包拟合随机森林
#e1071包构造支持向量机
#基本函数glm实现逻辑回归
library(rpart)
library(rpart.plot)
library(party)
library(randomForest)
library(e1071)
本章数据来源:UCI机器学习数据库中威斯康星州乳腺癌数据
目的:根据组织细胞针抽吸活检所反映的特征,来判断备件沿着是否患有乳腺癌
#数据准备
loc <- "http://archive.ics.uci.edu/ml/machine-learning-databases/"
ds <- 'breast-cancer-wisconsin/breast-cancer-wisconsin.data'
url <- paste(loc,ds,sep="")
breast <- read.table(url,sep=",",header=FALSE,na.strings="?")
names(breast) <- c("ID","clumpThickness","sizeUniformity",
"shapeUniformity","maginalAshesion",
"singleEpithelialCellSize","bareNuclei",
"blandChromatin","normalNucleoli","mitosis","class")df <- breast[-1] #去掉ID列
df$class <- factor(df$class,levels = c(2,4),
labels = c("benign","malignant")) #良性 恶性
str(df)
set.seed(1234)
#从699行中随机取489行用做训练集df.train,train为提取的行数号
train <- sample(nrow(df),0.7*nrow(df))
str(train)
df.train <- df[train,]
df.validate <- df[-train,]
table(df.train$class)
table(df.validate$class)
17.2 逻辑回归
#使用glm()进行逻辑回归
fit.logit <- glm(class~.,data = df.train,family = binomial())
summary(fit.logit)
prob <- predict(fit.logit,newdata =df.validate, type = "response" )
logit.pred <- factor(prob>0.5,levels = c(FALSE,TRUE),
labels = c("benign","malignant"))
logit.perf <- table(df.validate$class,logit.pred,
dnn = c("actual","predicted"))
logit.perf
17.3 决策树
17.3.1 经典决策树
#使用rpart()函数创建分类决策树
library(rpart)
set.seed(1234)
#生成树
dtree <- rpart(class~.,data=df.train,method = "class",
parms = list(split="information"))
dtree$cptable
#CP nsplit rel error xerror xstd
#1 0.81764706 0 1.00000000 1.0000000 0.06194645
#2 0.04117647 1 0.18235294 0.1823529 0.03169642
#3 0.01764706 3 0.10000000 0.1588235 0.02970979
#4 0.01000000 4 0.08235294 0.1235294 0.02637116
plotcp(dtree)
#根据复杂度参数减掉最不重要的枝
#依据dtree$cptable可以发现xerror最小值为0.1235294,对应xstd为0.02637116
#则最优的树为xerror在0.124±0.0264(0.0976,0.1504)之间,且值最小的树(对应nsplit=4 CP=0.01的树)
dtree.pruned <- prune(dtree,cp=0.01)
library(rpart.plot)
rpart.plot::prp(dtree.pruned,type=2,extra = 104,
fallen.leaves = TRUE,main="决策树")#extra = 104可画出每一类的概率以及每个节点处的样本占比
#从树的顶端开始,若满足条件,则从左枝往下,否则从右枝往下,该终端节点(nsplit+1)即为这一观测点所属类别
rpart.plot::prp(dtree.pruned,type=2,extra = 104,
fallen.leaves = FALSE,main="决策树") #左图#将传统推断树结果展现形式变成条件推断树
library(partykit)
plot(as.party(dtree.pruned)) #右图dtree.pred <- predict(dtree.pruned,newdata = df.validate,type = "class")
dtree.pref <- table(df.validate$class,dtree.pred,
dnn=c("actual","predicted"))
dtree.pref# predicted
# actual benign malignant
# benign 129 10
# malignant 2 69
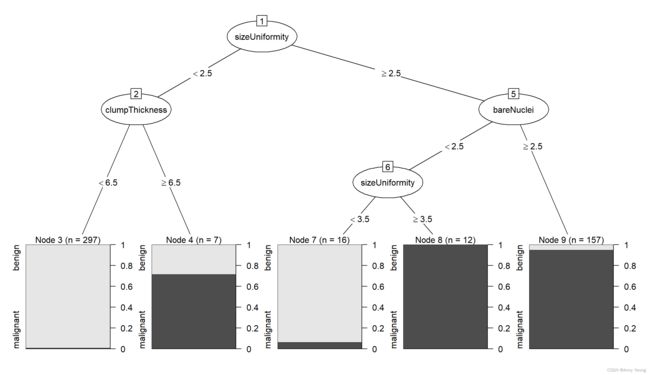
17.3.2 条件推断树 传统决策树的一种变体
library(party)
fit.ctree <- party::ctree(class~.,data=df.train)
plot(fit.ctree,main="条件推断树")
#值得注意的是,对于条件推断树来说,剪枝不是必需的, 其生成过程相对更自动化一些。
#另外, party 包也提供了许多图像参数。每个节点中的阴影区域代表这个节点对应的恶性肿瘤比例
ctree.pred <- predict(fit.ctree,newdata = df.validate,type = "response")
ctree.pref <- table(df.validate$class,ctree.pred,
dnn=c("actual","predicted"))
dtree.pref# predicted
# actual benign malignant
# benign 129 10
# malignant 2 69
17.4 随机森林
随机森林算法的实质是基于决策树的分类器集成算法,其中每一棵树都依赖于一个随机向量,随机森林的所有向量都是独立同分布的。随机森林就是对数据集的列变量和行观测进行随机化,生成多个分类数,最终将分类树结果进行汇总。
随机森林相比于神经网络,降低了运算量的同时也提高了预测精度,而且该算法对多元共线性不敏感以及对缺失数据和非平衡数据比较稳健,可以很好地适应多达几千个解释变量数据集。
随机森林的一个明显缺点是分类方法难以理解和表达。另外,我们需要存储整个随机森林以对新样本单元分类。
library(randomForest)
set.seed(1234)
#基于经典决策树的随机森林 默认生成500棵树 importance用于计算模型变量的重要性
fit.forest <- randomForest(class~.,data = df.train,
importance=TRUE,na.action = na.roughfix)#na.action=na.roughfix参数可以将数值变量中的缺失值替换成对应列的中位数;类别变量中的缺失值替换成对应列的众数类
fit.forestimportance(fit.forest,type = 2)
#type可以是1,也可以是2,用于判别计算变量重要性的方法,
#1表示使用精度平均较少值作为度量标准;2表示采用节点不纯度的平均减少值最为度量标准值越大说明变量的重要性越强forest.pred <- predict(fit.forest,newdata = df.validate)
forest.pref <- table(df.validate$class,forest.pred,
dnn=c("actual","predicted"))
forest.pref
17.5 支持向量机
#支持向量机(SVM)旨在在多维空间找到一个能将全部样本单元分成两类的最优平面的过程。在R中SVM可以通过kernlab包的ksvm()函数和e1071包中的svm()函数实现,前者功能更强大,后者相对简单。
library(e1071)
set.seed(1234)
fit.svm <- e1071::svm(class~.,data=df.train)
fit.svm
svm.pred <- predict(fit.svm,newdata = na.omit(df.validate))#svm函数默认在生成模型前对每个变量标准化,在预测新样本单元时不允许有缺失值出现
svm.pref <- table(na.omit(df.validate)$class,svm.pred,
dnn=c("actual","predicted"))
svm.pref#选择调和参数
set.seed(1234)
#对不同的gamma和cost拟合一个带RBF核的SVM模型
tuned <- tune.svm(class~.,data = df.train,
gamma = 10^(-6:1),cost=10^(-10:10))
tuned
#- best parameters:
# gamma cost
# 0.1 10
fit.svm1 <- e1071::svm(class~.,data=df.train,gamma=0.1,cost=10)
svm.pred1 <- predict(fit.svm1,newdata = na.omit(df.validate))#svm在预测新样本单元时不允许有缺失值出现
svm.pref1 <- table(na.omit(df.validate)$class,svm.pred1,
dnn=c("actual","predicted"))
svm.pref1
17.6 选择预测效果最好的解
#预测准确性度量:敏感度sensitivity 特异性specificity 正例命中率positive predictive power 负例命中率negative predictive power 准确率accuracy
#评估二分类准确性 构建代码
performance <- function(table,n=2){
if(!all(dim(table)==c(2,2)))
stop("must be a 2*2 table")
tn=table[1,1] #提取负类正确 良性被判断为良性
fp=table[1,2] #提取负类错误 恶性被判断为良性
fn=table[2,1] #提取正类错误 良性被判断为恶性
tp=table[2,2] #提取正类正确 恶性被判断为恶性
sensitivity=tp/(tp+fn)
specificity=tn/(tn+fp)
ppp=tp/(tp+fp)
npp=tn/(tn+fn)
hitrate=(tp+tn)/(tp+fn+fp+fn)
result <- paste("sensitivity=",round(sensitivity,n),
"\nspecificity=",round(specificity,n),
"\npositive predictive power=",round(ppp,n),
"\nnegative predictive power=",round(npp,n),
"\naccuracy=",round(hitrate,n),"\n",sep="")
cat(result)
}#对本章提到的五个分类器进行性能比较
performance(logit.pref)
performance(dtree.pref)
performance(ctree.pref)
performance(forest.pref)
performance(svm.pref)#本案例本次结果逻辑回归表现最佳