《利用Python进行数据分析·第2版》 附录B 更多关于IPython的内容(完)
第1章 准备工作
第2章 Python语法基础,IPython和Jupyter
第3章 Python的数据结构、函数和文件
第4章 NumPy基础:数组和矢量计算
第5章 pandas入门
第6章 数据加载、存储与文件格式
第7章 数据清洗和准备
第8章 数据规整:聚合、合并和重塑
第9章 绘图和可视化
第10章 数据聚合与分组运算
第11章 时间序列
第12章 pandas高级应用
第13章 Python建模库介绍
第14章 数据分析案例
附录A NumPy高级应用
附录B 更多关于IPython的内容(完)
第2章中,我们学习了IPython shell和Jupyter notebook的基础。本章中,我们会探索IPython更深层次的功能,可以从控制台或在jupyter使用。
B.1 使用命令历史
Ipython维护了一个位于磁盘的小型数据库,用于保存执行的每条指令。它的用途有:
- 只用最少的输入,就能搜索、补全和执行先前运行过的指令;
- 在不同session间保存命令历史;
- 将日志输入/输出历史到一个文件
这些功能在shell中,要比notebook更为有用,因为notebook从设计上是将输入和输出的代码放到每个代码格子中。
搜索和重复使用命令历史
Ipython可以让你搜索和执行之前的代码或其他命令。这个功能非常有用,因为你可能需要重复执行同样的命令,例如%run命令,或其它代码。假设你必须要执行:
In[7]: %run first/second/third/data_script.py
运行成功,然后检查结果,发现计算有错。解决完问题,然后修改了data_script.py,你就可以输入一些%run命令,然后按Ctrl+P或上箭头。这样就可以搜索历史命令,匹配输入字符的命令。多次按Ctrl+P或上箭头,会继续搜索命令。如果你要执行你想要执行的命令,不要害怕。你可以按下Ctrl-N或下箭头,向前移动历史命令。这样做了几次后,你可以不假思索地按下这些键!
Ctrl-R可以带来如同Unix风格shell(比如bash shell)的readline的部分增量搜索功能。在Windows上,readline功能是被IPython模仿的。要使用这个功能,先按Ctrl-R,然后输入一些包含于输入行的想要搜索的字符:
In [1]: a_command = foo(x, y, z)
(reverse-i-search)`com': a_command = foo(x, y, z)
Ctrl-R会循环历史,找到匹配字符的每一行。
输入和输出变量
忘记将函数调用的结果分配给变量是非常烦人的。IPython的一个session会在一个特殊变量,存储输入和输出Python对象的引用。前面两个输出会分别存储在 _(一个下划线)和 __(两个下划线)变量:
In [24]: 2 ** 27
Out[24]: 134217728
In [25]: _
Out[25]: 134217728
输入变量是存储在名字类似_iX的变量中,X是输入行的编号。对于每个输入变量,都有一个对应的输出变量_X。因此在输入第27行之后,会有两个新变量_27 (输出)和_i27(输入):
In [26]: foo = 'bar'
In [27]: foo
Out[27]: 'bar'
In [28]: _i27
Out[28]: u'foo'
In [29]: _27
Out[29]: 'bar'
因为输入变量是字符串,它们可以用Python的exec关键字再次执行:
In [30]: exec(_i27)
这里,_i27是在In [27]输入的代码。
有几个魔术函数可以让你利用输入和输出历史。%hist可以打印所有或部分的输入历史,加上或不加上编号。%reset可以清理交互命名空间,或输入和输出缓存。%xdel魔术函数可以去除IPython中对一个特别对象的所有引用。对于关于这些魔术方法的更多内容,请查看文档。
警告:当处理非常大的数据集时,要记住IPython的输入和输出的历史会造成被引用的对象不被垃圾回收(释放内存),即使你使用del关键字从交互命名空间删除变量。在这种情况下,小心使用xdel %和%reset可以帮助你避免陷入内存问题。
B.2 与操作系统交互
IPython的另一个功能是无缝连接文件系统和操作系统。这意味着,在同时做其它事时,无需退出IPython,就可以像Windows或Unix使用命令行操作,包括shell命令、更改目录、用Python对象(列表或字符串)存储结果。它还有简单的命令别名和目录书签功能。
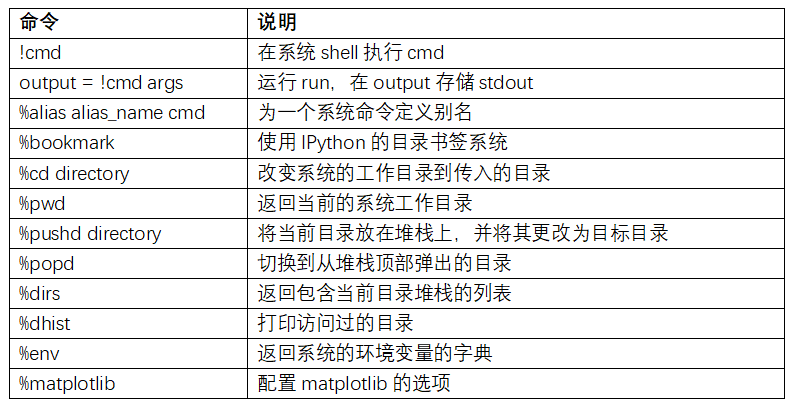
表B-1总结了调用shell命令的魔术函数和语法。我会在下面几节介绍这些功能。
Shell命令和别名
用叹号开始一行,是告诉IPython执行叹号后面的所有内容。这意味着你可以删除文件(取决于操作系统,用rm或del)、改变目录或执行任何其他命令。
通过给变量加上叹号,你可以在一个变量中存储命令的控制台输出。例如,在我联网的基于Linux的主机上,我可以获得IP地址为Python变量:
In [1]: ip_info = !ifconfig wlan0 | grep "inet "
In [2]: ip_info[0].strip()
Out[2]: 'inet addr:10.0.0.11 Bcast:10.0.0.255 Mask:255.255.255.0'
返回的Python对象ip_info实际上是一个自定义的列表类型,它包含着多种版本的控制台输出。
当使用!,IPython还可以替换定义在当前环境的Python值。要这么做,可以在变量名前面加上$符号:
In [3]: foo = 'test*'
In [4]: !ls $foo
test4.py test.py test.xml
%alias魔术函数可以自定义shell命令的快捷方式。看一个简单的例子:
In [1]: %alias ll ls -l
In [2]: ll /usr
total 332
drwxr-xr-x 2 root root 69632 2012-01-29 20:36 bin/
drwxr-xr-x 2 root root 4096 2010-08-23 12:05 games/
drwxr-xr-x 123 root root 20480 2011-12-26 18:08 include/
drwxr-xr-x 265 root root 126976 2012-01-29 20:36 lib/
drwxr-xr-x 44 root root 69632 2011-12-26 18:08 lib32/
lrwxrwxrwx 1 root root 3 2010-08-23 16:02 lib64 -> lib/
drwxr-xr-x 15 root root 4096 2011-10-13 19:03 local/
drwxr-xr-x 2 root root 12288 2012-01-12 09:32 sbin/
drwxr-xr-x 387 root root 12288 2011-11-04 22:53 share/
drwxrwsr-x 24 root src 4096 2011-07-17 18:38 src/
你可以执行多个命令,就像在命令行中一样,只需用分号隔开:
In [558]: %alias test_alias (cd examples; ls; cd ..)
In [559]: test_alias
macrodata.csv spx.csv tips.csv
当session结束,你定义的别名就会失效。要创建恒久的别名,需要使用配置。
目录书签系统
IPython有一个简单的目录书签系统,可以让你保存常用目录的别名,这样在跳来跳去的时候会非常方便。例如,假设你想创建一个书签,指向本书的补充内容:
In [6]: %bookmark py4da /home/wesm/code/pydata-book
这么做之后,当使用%cd魔术命令,就可以使用定义的书签:
In [7]: cd py4da
(bookmark:py4da) -> /home/wesm/code/pydata-book
/home/wesm/code/pydata-book
如果书签的名字,与当前工作目录的一个目录重名,你可以使用-b标志来覆写,使用书签的位置。使用%bookmark的-l选项,可以列出所有的书签:
In [8]: %bookmark -l
Current bookmarks:
py4da -> /home/wesm/code/pydata-book-source
书签,和别名不同,在session之间是保持的。
B.3 软件开发工具
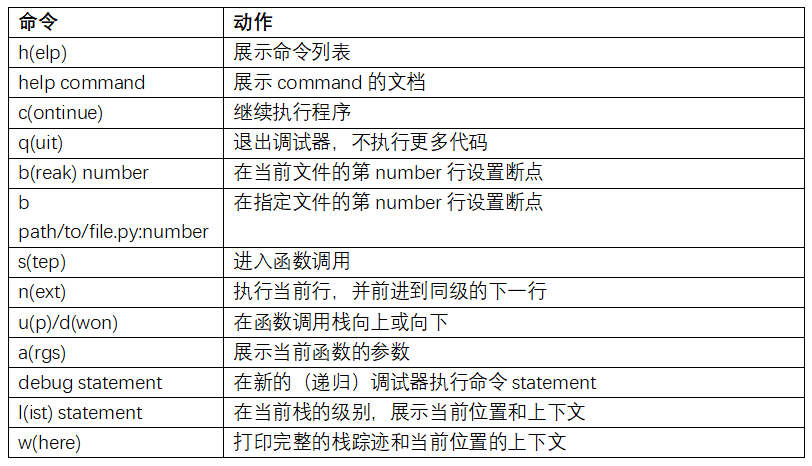
除了作为优秀的交互式计算和数据探索环境,IPython也是有效的Python软件开发工具。在数据分析中,最重要的是要有正确的代码。幸运的是,IPython紧密集成了和加强了Python内置的pdb调试器。第二,需要快速的代码。对于这点,IPython有易于使用的代码计时和分析工具。我会详细介绍这些工具。
交互调试器
IPython的调试器用tab补全、语法增强、逐行异常追踪增强了pdb。调试代码的最佳时间就是刚刚发生错误。异常发生之后就输入%debug,就启动了调试器,进入抛出异常的堆栈框架:
In [2]: run examples/ipython_bug.py
---------------------------------------------------------------------------
AssertionError Traceback (most recent call last)
/home/wesm/code/pydata-book/examples/ipython_bug.py in ()
13 throws_an_exception()
14
---> 15 calling_things()
/home/wesm/code/pydata-book/examples/ipython_bug.py in calling_things()
11 def calling_things():
12 works_fine()
---> 13 throws_an_exception()
14
15 calling_things()
/home/wesm/code/pydata-book/examples/ipython_bug.py in throws_an_exception()
7 a = 5
8 b = 6
----> 9 assert(a + b == 10)
10
11 def calling_things():
AssertionError:
In [3]: %debug
> /home/wesm/code/pydata-book/examples/ipython_bug.py(9)throws_an_exception()
8 b = 6
----> 9 assert(a + b == 10)
10
ipdb>
一旦进入调试器,你就可以执行任意的Python代码,在每个堆栈框架中检查所有的对象和数据(解释器会保持它们活跃)。默认是从错误发生的最低级开始。通过u(up)和d(down),你可以在不同等级的堆栈踪迹切换:
ipdb> u
> /home/wesm/code/pydata-book/examples/ipython_bug.py(13)calling_things()
12 works_fine()
---> 13 throws_an_exception()
14
执行%pdb命令,可以在发生任何异常时让IPython自动启动调试器,许多用户会发现这个功能非常好用。
用调试器帮助开发代码也很容易,特别是当你希望设置断点或在函数和脚本间移动,以检查每个阶段的状态。有多种方法可以实现。第一种是使用%run和-d,它会在执行传入脚本的任何代码之前调用调试器。你必须马上按s(step)以进入脚本:
In [5]: run -d examples/ipython_bug.py
Breakpoint 1 at /home/wesm/code/pydata-book/examples/ipython_bug.py:1
NOTE: Enter 'c' at the ipdb> prompt to start your script.
> (1)()
ipdb> s
--Call--
> /home/wesm/code/pydata-book/examples/ipython_bug.py(1)()
1---> 1 def works_fine():
2 a = 5
3 b = 6
然后,你就可以决定如何工作。例如,在前面的异常,我们可以设置一个断点,就在调用works_fine之前,然后运行脚本,在遇到断点时按c(continue):
ipdb> b 12
ipdb> c
> /home/wesm/code/pydata-book/examples/ipython_bug.py(12)calling_things()
11 def calling_things():
2--> 12 works_fine()
13 throws_an_exception()
这时,你可以step进入works_fine(),或通过按n(next)执行works_fine(),进入下一行:
ipdb> n
> /home/wesm/code/pydata-book/examples/ipython_bug.py(13)calling_things()
2 12 works_fine()
---> 13 throws_an_exception()
14
然后,我们可以进入throws_an_exception,到达发生错误的一行,查看变量。注意,调试器的命令是在变量名之前,在变量名前面加叹号!可以查看内容:
ipdb> s
--Call--
> /home/wesm/code/pydata-book/examples/ipython_bug.py(6)throws_an_exception()
5
----> 6 def throws_an_exception():
7 a = 5
ipdb> n
> /home/wesm/code/pydata-book/examples/ipython_bug.py(7)throws_an_exception()
6 def throws_an_exception():
----> 7 a = 5
8 b = 6
ipdb> n
> /home/wesm/code/pydata-book/examples/ipython_bug.py(8)throws_an_exception()
7 a = 5
----> 8 b = 6
9 assert(a + b == 10)
ipdb> n
> /home/wesm/code/pydata-book/examples/ipython_bug.py(9)throws_an_exception()
8 b = 6
----> 9 assert(a + b == 10)
10
ipdb> !a
5
ipdb> !b
6
提高使用交互式调试器的熟练度需要练习和经验。表B-2,列出了所有调试器命令。如果你习惯了IDE,你可能觉得终端的调试器在一开始会不顺手,但会觉得越来越好用。一些Python的IDEs有很好的GUI调试器,选择顺手的就好。
使用调试器的其它方式
还有一些其它工作可以用到调试器。第一个是使用特殊的set_trace函数(根据pdb.set_trace命名的),这是一个简装的断点。还有两种方法是你可能想用的(像我一样,将其添加到IPython的配置):
from IPython.core.debugger import Pdb
def set_trace():
Pdb(color_scheme='Linux').set_trace(sys._getframe().f_back)
def debug(f, *args, **kwargs):
pdb = Pdb(color_scheme='Linux')
return pdb.runcall(f, *args, **kwargs)
第一个函数set_trace非常简单。如果你想暂时停下来进行仔细检查(比如发生异常之前),可以在代码的任何位置使用set_trace:
In [7]: run examples/ipython_bug.py
> /home/wesm/code/pydata-book/examples/ipython_bug.py(16)calling_things()
15 set_trace()
---> 16 throws_an_exception()
17
按c(continue)可以让代码继续正常行进。
我们刚看的debug函数,可以让你方便的在调用任何函数时使用调试器。假设我们写了一个下面的函数,想逐步分析它的逻辑:
def f(x, y, z=1):
tmp = x + y
return tmp / z
普通地使用f,就会像f(1, 2, z=3)。而要想进入f,将f作为第一个参数传递给debug,再将位置和关键词参数传递给f:
In [6]: debug(f, 1, 2, z=3)
> (2)f()
1 def f(x, y, z):
----> 2 tmp = x + y
3 return tmp / z
ipdb>
这两个简单方法节省了我平时的大量时间。
最后,调试器可以和%run一起使用。脚本通过运行%run -d,就可以直接进入调试器,随意设置断点并启动脚本:
In [1]: %run -d examples/ipython_bug.py
Breakpoint 1 at /home/wesm/code/pydata-book/examples/ipython_bug.py:1
NOTE: Enter 'c' at the ipdb> prompt to start your script.
> (1)()
ipdb>
加上-b和行号,可以预设一个断点:
In [2]: %run -d -b2 examples/ipython_bug.py
Breakpoint 1 at /home/wesm/code/pydata-book/examples/ipython_bug.py:2
NOTE: Enter 'c' at the ipdb> prompt to start your script.
> (1)()
ipdb> c
> /home/wesm/code/pydata-book/examples/ipython_bug.py(2)works_fine()
1 def works_fine():
1---> 2 a = 5
3 b = 6
ipdb>
代码计时:%time 和 %timeit
对于大型和长时间运行的数据分析应用,你可能希望测量不同组件或单独函数调用语句的执行时间。你可能想知道哪个函数占用的时间最长。幸运的是,IPython可以让你开发和测试代码时,很容易地获得这些信息。
手动用time模块和它的函数time.clock和time.time给代码计时,既单调又重复,因为必须要写一些无趣的模板化代码:
import time
start = time.time()
for i in range(iterations):
# some code to run here
elapsed_per = (time.time() - start) / iterations
因为这是一个很普通的操作,IPython有两个魔术函数,%time和%timeit,可以自动化这个过程。
%time会运行一次语句,报告总共的执行时间。假设我们有一个大的字符串列表,我们想比较不同的可以挑选出特定开头字符串的方法。这里有一个含有600000字符串的列表,和两个方法,用以选出foo开头的字符串:
# a very large list of strings
strings = ['foo', 'foobar', 'baz', 'qux',
'python', 'Guido Van Rossum'] * 100000
method1 = [x for x in strings if x.startswith('foo')]
method2 = [x for x in strings if x[:3] == 'foo']
看起来它们的性能应该是同级别的,但事实呢?用%time进行一下测量:
In [561]: %time method1 = [x for x in strings if x.startswith('foo')]
CPU times: user 0.19 s, sys: 0.00 s, total: 0.19 s
Wall time: 0.19 s
In [562]: %time method2 = [x for x in strings if x[:3] == 'foo']
CPU times: user 0.09 s, sys: 0.00 s, total: 0.09 s
Wall time: 0.09 s
Wall time(wall-clock time的简写)是主要关注的。第一个方法是第二个方法的两倍多,但是这种测量方法并不准确。如果用%time多次测量,你就会发现结果是变化的。要想更准确,可以使用%timeit魔术函数。给出任意一条语句,它能多次运行这条语句以得到一个更为准确的时间:
In [563]: %timeit [x for x in strings if x.startswith('foo')]
10 loops, best of 3: 159 ms per loop
In [564]: %timeit [x for x in strings if x[:3] == 'foo']
10 loops, best of 3: 59.3 ms per loop
这个例子说明了解Python标准库、NumPy、pandas和其它库的性能是很有价值的。在大型数据分析中,这些毫秒的时间就会累积起来!
%timeit特别适合分析执行时间短的语句和函数,即使是微秒或纳秒。这些时间可能看起来毫不重要,但是一个20微秒的函数执行1百万次就比一个5微秒的函数长15秒。在上一个例子中,我们可以直接比较两个字符串操作,以了解它们的性能特点:
In [565]: x = 'foobar'
In [566]: y = 'foo'
In [567]: %timeit x.startswith(y)
1000000 loops, best of 3: 267 ns per loop
In [568]: %timeit x[:3] == y
10000000 loops, best of 3: 147 ns per loop
基础分析:%prun和%run -p
分析代码与代码计时关系很紧密,除了它关注的是“时间花在了哪里”。Python主要的分析工具是cProfile模块,它并不局限于IPython。cProfile会执行一个程序或任意的代码块,并会跟踪每个函数执行的时间。
使用cProfile的通常方式是在命令行中运行一整段程序,输出每个函数的累积时间。假设我们有一个简单的在循环中进行线型代数运算的脚本(计算一系列的100×100矩阵的最大绝对特征值):
import numpy as np
from numpy.linalg import eigvals
def run_experiment(niter=100):
K = 100
results = []
for _ in xrange(niter):
mat = np.random.randn(K, K)
max_eigenvalue = np.abs(eigvals(mat)).max()
results.append(max_eigenvalue)
return results
some_results = run_experiment()
print 'Largest one we saw: %s' % np.max(some_results)
你可以用cProfile运行这个脚本,使用下面的命令行:
python -m cProfile cprof_example.py
运行之后,你会发现输出是按函数名排序的。这样要看出谁耗费的时间多有点困难,最好用-s指定排序:
$ python -m cProfile -s cumulative cprof_example.py
Largest one we saw: 11.923204422
15116 function calls (14927 primitive calls) in 0.720 seconds
Ordered by: cumulative time
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.001 0.001 0.721 0.721 cprof_example.py:1()
100 0.003 0.000 0.586 0.006 linalg.py:702(eigvals)
200 0.572 0.003 0.572 0.003 {numpy.linalg.lapack_lite.dgeev}
1 0.002 0.002 0.075 0.075 __init__.py:106()
100 0.059 0.001 0.059 0.001 {method 'randn')
1 0.000 0.000 0.044 0.044 add_newdocs.py:9()
2 0.001 0.001 0.037 0.019 __init__.py:1()
2 0.003 0.002 0.030 0.015 __init__.py:2()
1 0.000 0.000 0.030 0.030 type_check.py:3()
1 0.001 0.001 0.021 0.021 __init__.py:15()
1 0.013 0.013 0.013 0.013 numeric.py:1()
1 0.000 0.000 0.009 0.009 __init__.py:6()
1 0.001 0.001 0.008 0.008 __init__.py:45()
262 0.005 0.000 0.007 0.000 function_base.py:3178(add_newdoc)
100 0.003 0.000 0.005 0.000 linalg.py:162(_assertFinite)
只显示出前15行。扫描cumtime列,可以容易地看出每个函数用了多少时间。如果一个函数调用了其它函数,计时并不会停止。cProfile会记录每个函数的起始和结束时间,使用它们进行计时。
除了在命令行中使用,cProfile也可以在程序中使用,分析任意代码块,而不必运行新进程。Ipython的%prun和%run -p,有便捷的接口实现这个功能。%prun使用类似cProfile的命令行选项,但是可以分析任意Python语句,而不用整个py文件:
In [4]: %prun -l 7 -s cumulative run_experiment()
4203 function calls in 0.643 seconds
Ordered by: cumulative time
List reduced from 32 to 7 due to restriction <7>
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.000 0.000 0.643 0.643 :1()
1 0.001 0.001 0.643 0.643 cprof_example.py:4(run_experiment)
100 0.003 0.000 0.583 0.006 linalg.py:702(eigvals)
200 0.569 0.003 0.569 0.003 {numpy.linalg.lapack_lite.dgeev}
100 0.058 0.001 0.058 0.001 {method 'randn'}
100 0.003 0.000 0.005 0.000 linalg.py:162(_assertFinite)
200 0.002 0.000 0.002 0.000 {method 'all' of 'numpy.ndarray'}
相似的,调用%run -p -s cumulative cprof_example.py有和命令行相似的作用,只是你不用离开Ipython。
在Jupyter notebook中,你可以使用%%prun魔术方法(两个%)来分析一整段代码。这会弹出一个带有分析输出的独立窗口。便于快速回答一些问题,比如“为什么这段代码用了这么长时间”?
使用IPython或Jupyter,还有一些其它工具可以让分析工作更便于理解。其中之一是SnakeViz(https://github.com/jiffyclub/snakeviz/),它会使用d3.js产生一个分析结果的交互可视化界面。
逐行分析函数
有些情况下,用%prun(或其它基于cProfile的分析方法)得到的信息,不能获得函数执行时间的整个过程,或者结果过于复杂,加上函数名,很难进行解读。对于这种情况,有一个小库叫做line_profiler(可以通过PyPI或包管理工具获得)。它包含IPython插件,可以启用一个新的魔术函数%lprun,可以对一个函数或多个函数进行逐行分析。你可以通过修改IPython配置(查看IPython文档或本章后面的配置小节)加入下面这行,启用这个插件:
# A list of dotted module names of IPython extensions to load.
c.TerminalIPythonApp.extensions = ['line_profiler']
你还可以运行命令:
%load_ext line_profiler
line_profiler也可以在程序中使用(查看完整文档),但是在IPython中使用是最为强大的。假设你有一个带有下面代码的模块prof_mod,做一些NumPy数组操作:
from numpy.random import randn
def add_and_sum(x, y):
added = x + y
summed = added.sum(axis=1)
return summed
def call_function():
x = randn(1000, 1000)
y = randn(1000, 1000)
return add_and_sum(x, y)
如果想了解add_and_sum函数的性能,%prun可以给出下面内容:
In [569]: %run prof_mod
In [570]: x = randn(3000, 3000)
In [571]: y = randn(3000, 3000)
In [572]: %prun add_and_sum(x, y)
4 function calls in 0.049 seconds
Ordered by: internal time
ncalls tottime percall cumtime percall filename:lineno(function)
1 0.036 0.036 0.046 0.046 prof_mod.py:3(add_and_sum)
1 0.009 0.009 0.009 0.009 {method 'sum' of 'numpy.ndarray'}
1 0.003 0.003 0.049 0.049 :1()
上面的做法启发性不大。激活了IPython插件line_profiler,新的命令%lprun就能用了。使用中的不同点是,我们必须告诉%lprun要分析的函数是哪个。语法是:
%lprun -f func1 -f func2 statement_to_profile
我们想分析add_and_sum,运行:
In [573]: %lprun -f add_and_sum add_and_sum(x, y)
Timer unit: 1e-06 s
File: prof_mod.py
Function: add_and_sum at line 3
Total time: 0.045936 s
Line # Hits Time Per Hit % Time Line Contents
==============================================================
3 def add_and_sum(x, y):
4 1 36510 36510.0 79.5 added = x + y
5 1 9425 9425.0 20.5 summed = added.sum(axis=1)
6 1 1 1.0 0.0 return summed
这样就容易诠释了。我们分析了和代码语句中一样的函数。看之前的模块代码,我们可以调用call_function并对它和add_and_sum进行分析,得到一个完整的代码性能概括:
In [574]: %lprun -f add_and_sum -f call_function call_function()
Timer unit: 1e-06 s
File: prof_mod.py
Function: add_and_sum at line 3
Total time: 0.005526 s
Line # Hits Time Per Hit % Time Line Contents
==============================================================
3 def add_and_sum(x, y):
4 1 4375 4375.0 79.2 added = x + y
5 1 1149 1149.0 20.8 summed = added.sum(axis=1)
6 1 2 2.0 0.0 return summed
File: prof_mod.py
Function: call_function at line 8
Total time: 0.121016 s
Line # Hits Time Per Hit % Time Line Contents
==============================================================
8 def call_function():
9 1 57169 57169.0 47.2 x = randn(1000, 1000)
10 1 58304 58304.0 48.2 y = randn(1000, 1000)
11 1 5543 5543.0 4.6 return add_and_sum(x, y)
我的经验是用%prun (cProfile)进行宏观分析,%lprun (line_profiler)做微观分析。最好对这两个工具都了解清楚。
笔记:使用%lprun必须要指明函数名的原因是追踪每行的执行时间的损耗过多。追踪无用的函数会显著地改变结果。
B.4 使用IPython高效开发的技巧
方便快捷地写代码、调试和使用是每个人的目标。除了代码风格,流程细节(比如代码重载)也需要一些调整。
因此,这一节的内容更像是门艺术而不是科学,还需要你不断的试验,以达成高效。最终,你要能结构优化代码,并且能省时省力地检查程序或函数的结果。我发现用IPython设计的软件比起命令行,要更适合工作。尤其是当发生错误时,你需要检查自己或别人写的数月或数年前写的代码的错误。
重载模块依赖
在Python中,当你输入import some_lib,some_lib中的代码就会被执行,所有的变量、函数和定义的引入,就会被存入到新创建的some_lib模块命名空间。当下一次输入some_lib,就会得到一个已存在的模块命名空间的引用。潜在的问题是当你%run一个脚本,它依赖于另一个模块,而这个模块做过修改,就会产生问题。假设我在test_script.py中有如下代码:
import some_lib
x = 5
y = [1, 2, 3, 4]
result = some_lib.get_answer(x, y)
如果你运行过了%run test_script.py,然后修改了some_lib.py,下一次再执行%run test_script.py,还会得到旧版本的some_lib.py,这是因为Python模块系统的“一次加载”机制。这一点区分了Python和其它数据分析环境,比如MATLAB,它会自动传播代码修改。解决这个问题,有多种方法。第一种是在标准库importlib模块中使用reload函数:
import some_lib
import importlib
importlib.reload(some_lib)
这可以保证每次运行test_script.py时可以加载最新的some_lib.py。很明显,如果依赖更深,在各处都使用reload是非常麻烦的。对于这个问题,IPython有一个特殊的dreload函数(它不是魔术函数)重载深层的模块。如果我运行过some_lib.py,然后输入dreload(some_lib),就会尝试重载some_lib和它的依赖。不过,这个方法不适用于所有场景,但比重启IPython强多了。
代码设计技巧
对于这单,没有简单的对策,但是有一些原则,是我在工作中发现很好用的。
保持相关对象和数据活跃
为命令行写一个下面示例中的程序是很少见的:
from my_functions import g
def f(x, y):
return g(x + y)
def main():
x = 6
y = 7.5
result = x + y
if __name__ == '__main__':
main()
在IPython中运行这个程序会发生问题,你发现是什么了吗?运行之后,任何定义在main函数中的结果和对象都不能在IPython中被访问到。更好的方法是将main中的代码直接在模块的命名空间中执行(或者在__name__ == '__main__':中,如果你想让这个模块可以被引用)。这样,当你%rundiamante,就可以查看所有定义在main中的变量。这等价于在Jupyter notebook的代码格中定义一个顶级变量。
扁平优于嵌套
深层嵌套的代码总让我联想到洋葱皮。当测试或调试一个函数时,你需要剥多少层洋葱皮才能到达目标代码呢?“扁平优于嵌套”是Python之禅的一部分,它也适用于交互式代码开发。尽量将函数和类去耦合和模块化,有利于测试(如果你是在写单元测试)、调试和交互式使用。
克服对大文件的恐惧
如果你之前是写JAVA(或者其它类似的语言),你可能被告知要让文件简短。在多数语言中,这都是合理的建议:太长会让人感觉是坏代码,意味着重构和重组是必要的。但是,在用IPython开发时,运行10个相关联的小文件(小于100行),比起两个或三个长文件,会让你更头疼。更少的文件意味着重载更少的模块和更少的编辑时在文件中跳转。我发现维护大模块,每个模块都是紧密组织的,会更实用和Pythonic。经过方案迭代,有时会将大文件分解成小文件。
我不建议极端化这条建议,那样会形成一个单独的超大文件。找到一个合理和直观的大型代码模块库和封装结构往往需要一点工作,但这在团队工作中非常重要。每个模块都应该结构紧密,并且应该能直观地找到负责每个功能领域功能和类。
B.5 IPython高级功能
要全面地使用IPython系统需要用另一种稍微不同的方式写代码,或深入IPython的配置。
让类是对IPython友好的
IPython会尽可能地在控制台美化展示每个字符串。对于许多对象,比如字典、列表和元组,内置的pprint模块可以用来美化格式。但是,在用户定义的类中,你必自己生成字符串。假设有一个下面的简单的类:
class Message:
def __init__(self, msg):
self.msg = msg
如果这么写,就会发现默认的输出不够美观:
In [576]: x = Message('I have a secret')
In [577]: x
Out[577]: <__main__.Message instance at 0x60ebbd8>
IPython会接收repr魔术方法返回的字符串(通过output = repr(obj)),并在控制台打印出来。因此,我们可以添加一个简单的repr方法到前面的类中,以得到一个更有用的输出:
class Message:
def __init__(self, msg):
self.msg = msg
def __repr__(self):
return 'Message: %s' % self.msg
In [579]: x = Message('I have a secret')
In [580]: x
Out[580]: Message: I have a secret
文件和配置
通过扩展配置系统,大多数IPython和Jupyter notebook的外观(颜色、提示符、行间距等等)和动作都是可以配置的。通过配置,你可以做到:
- 改变颜色主题
- 改变输入和输出提示符,或删除输出之后、输入之前的空行
- 执行任意Python语句(例如,引入总是要使用的代码或者每次加载IPython都要运行的内容)
- 启用IPython总是要运行的插件,比如line_profiler中的%lprun魔术函数
- 启用Jupyter插件
- 定义自己的魔术函数或系统别名
IPython的配置存储在特殊的ipython_config.py文件中,它通常是在用户home目录的.ipython/文件夹中。配置是通过一个特殊文件。当你启动IPython,就会默认加载这个存储在profile_default文件夹中的默认文件。因此,在我的Linux系统,完整的IPython配置文件路径是:
/home/wesm/.ipython/profile_default/ipython_config.py
要启动这个文件,运行下面的命令:
ipython profile create
这个文件中的内容留给读者自己探索。这个文件有注释,解释了每个配置选项的作用。另一点,可以有多个配置文件。假设你想要另一个IPython配置文件,专门是为另一个应用或项目的。创建一个新的配置文件很简单,如下所示:
ipython profile create secret_project
做完之后,在新创建的profile_secret_project目录便捷配置文件,然后如下启动IPython:
$ ipython --profile=secret_project
Python 3.5.1 | packaged by conda-forge | (default, May 20 2016, 05:22:56)
Type "copyright", "credits" or "license" for more information.
IPython 5.1.0 -- An enhanced Interactive Python.
? -> Introduction and overview of IPython's features.
%quickref -> Quick reference.
help -> Python's own help system.
object? -> Details about 'object', use 'object??' for extra details.
IPython profile: secret_project
和之前一样,IPython的文档是一个极好的学习配置文件的资源。
配置Jupyter有些不同,因为你可以使用除了Python的其它语言。要创建一个类似的Jupyter配置文件,运行:
jupyter notebook --generate-config
这样会在home目录的.jupyter/jupyter_notebook_config.py创建配置文件。编辑完之后,可以将它重命名:
$ mv ~/.jupyter/jupyter_notebook_config.py ~/.jupyter/my_custom_config.py
打开Jupyter之后,你可以添加--config参数:
jupyter notebook --config=~/.jupyter/my_custom_config.py
B.6 总结
学习过本书中的代码案例,你的Python技能得到了一定的提升,我建议你持续学习IPython和Jupyter。因为这两个项目的设计初衷就是提高生产率的,你可能还会发现一些工具,可以让你更便捷地使用Python和计算库。
你可以在nbviewer(https://nbviewer.jupyter.org/)上找到更多有趣的Jupyter notebooks。
第1章 准备工作
第2章 Python语法基础,IPython和Jupyter
第3章 Python的数据结构、函数和文件
第4章 NumPy基础:数组和矢量计算
第5章 pandas入门
第6章 数据加载、存储与文件格式
第7章 数据清洗和准备
第8章 数据规整:聚合、合并和重塑
第9章 绘图和可视化
第10章 数据聚合与分组运算
第11章 时间序列
第12章 pandas高级应用
第13章 Python建模库介绍
第14章 数据分析案例
附录A NumPy高级应用
附录B 更多关于IPython的内容(完)
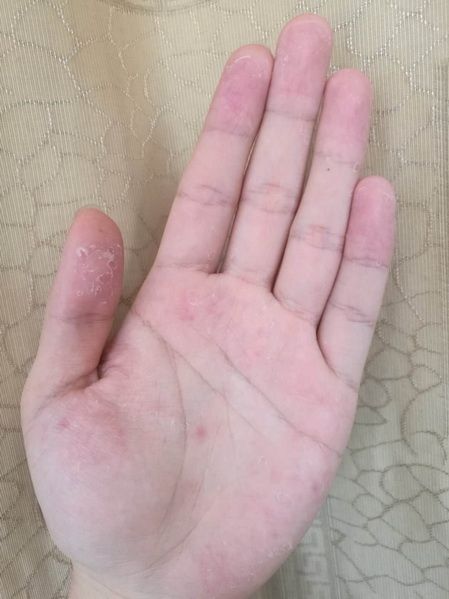
后记:经过三个月,总算翻译完成了这本书。工作砌码,回家码字。最大的改变是,十个手指头,除了两个大拇指和右手的小拇指,其它指尖竟然掉皮、磨出了茧。好长时间,只要手一沾水,就会起皱。读者们持续的阅读、点赞、留言、指出错误,让我感觉是和很多人一起完成一项有意义的事情。Thanks all!
后记2:2018年8月5日,做完了第一次校阅。