使用ggstatsplot简化数据探索:一步完成数据可视化和统计建模 | 简说基因 Recommend...
【简说基因】ggstatsplot:画图并自动为图形加上丰富的统计信息。
在典型的探索性数据分析工作流中,数据可视化和统计建模是两个不同的阶段:可视化有助于建模,而建模反过来又可以建议不同的可视化方法,依此类推。ggstatsplot 的核心思想很简单:将这两个阶段合并为一个,以图形的形式呈现统计细节,使数据探索变得更简单、更快速。
ggstatsplot 是 ggplot2 的一个扩展包,它提供 9 个函数用于常见的统计作图,根据功能可以将它们分成 4 类:
分布
数据的分布(gghistostats(),直方图)
带标签的数据的分布(ggdotplotstats(), 点图)
比较
数值数据的组间比较(ggbetweenstats(), 小提琴图)
数值数据的组内比较(ggwithinstats(), 小提琴图)
分类数据的组间比较(ggbarstats(), 柱状图)
分类数据的组间比较(ggpiestats(), 饼图)
相关性
两个变量之间的相关性(ggscatterstats(), 散点图)
多个变量之间的相关性(ggcorrmat(), 相关性矩阵图)
回归
回归模型和 meta 分析(ggcoefstats(), 点须图)
我们先来看一下统计报告的格式。ggstatsplot 默认的模板遵循统计报告的黄金标准,既报告了传统的频率学派分析结果,也包含贝叶斯分析结果,详情看下图:

分布
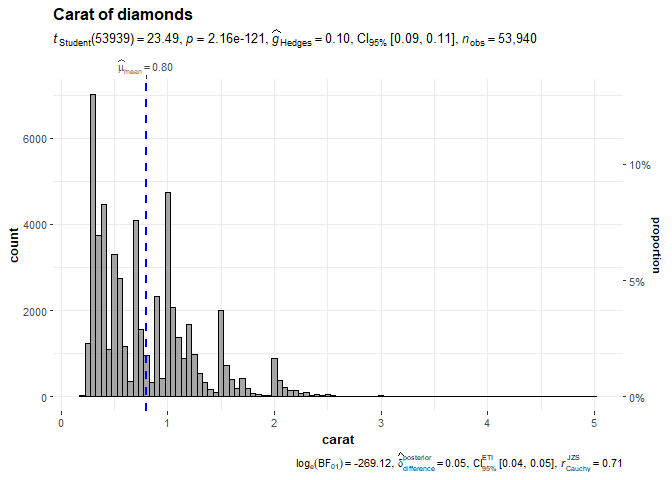
1. 数据的分布(gghistostats(),直方图)
可视化单个变量的分布,并进行单样本检验,看样本均值与指定值是否有显著不同。
# install.packages("ggstatsplot")
library(ggstatsplot)
library(ggplot2)
set.seed(123)
gghistostats(
data = diamonds,
x = carat,
title = "Carat of diamonds",
test.value = 0.75,
binwidth = 0.05
)
2. 带标签的数据的分布(ggdotplotstats(), 点图)
当数字变量带有标签时,用点图进行可视化,同时进行单样本检验。
set.seed(123)
ggdotplotstats(
data = dplyr::filter(gapminder::gapminder, continent == "Asia"),
x = lifeExp,
y = country,
test.value = 55,
type = "robust",
title = "Distribution of life expectancy in Asian continent",
xlab = "Life expectancy"
)
比较
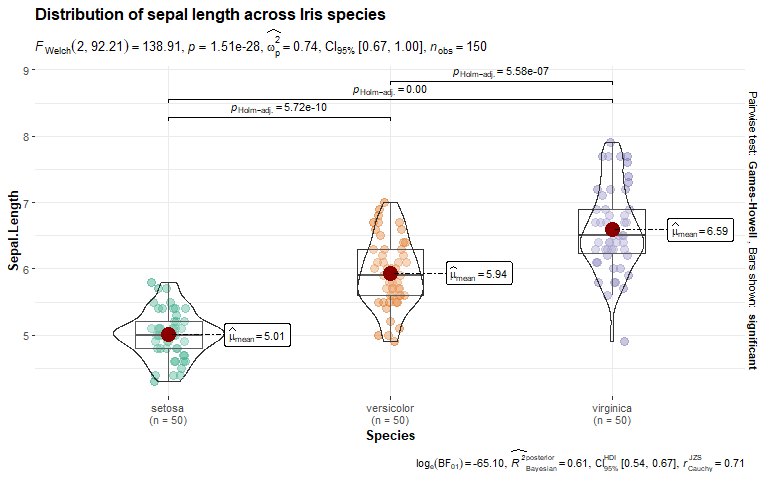
3. 数值数据的组间比较(ggbetweenstats(), 小提琴图)
组合箱线图、小提琴图和抖动散点图,并将统计信息展示在副标题和图注中。
set.seed(123)
ggbetweenstats(
data = iris,
x = Species,
y = Sepal.Length,
title = "Distribution of sepal length across Iris species"
)
4. 数值数据的组内比较(ggwithinstats(), 小提琴图)
对于重复测量数据,可以使用 ggwithinstats()绘图并进行配对样本检验。
set.seed(123)
library(WRS2) ## for data
library(afex) ## to run ANOVA
ggwithinstats(
data = WineTasting,
x = Wine,
y = Taste,
title = "Wine tasting"
)
5. 分类数据的组间比较(ggbarstats(), 柱状图)
可以通过百分比柱状图展示分类变量,注意 x 参数将作为列联表的行,y 参数将作为列联表的列。
set.seed(123)
library(ggplot2)
ggbarstats(
data = mtcars,
x = am,
y = cyl,
title = "cyl by am",
legend.title = "Transmission",
ggplot.component = list(ggplot2::scale_x_discrete(guide = ggplot2::guide_axis(n.dodge = 2))),
palette = "Set2"
)
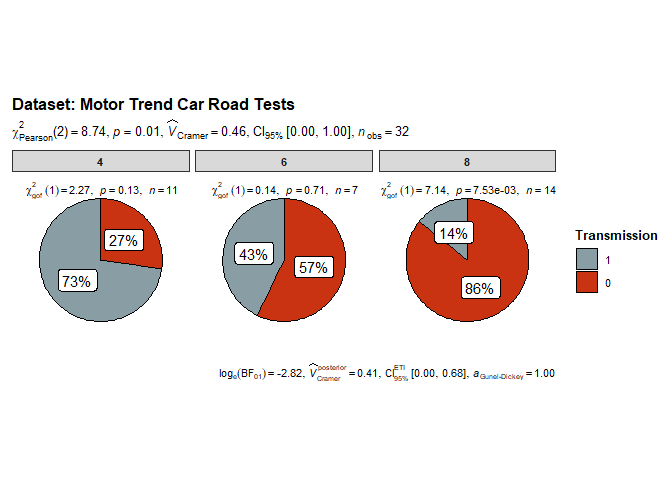
6. 分类数据的组间比较(ggpiestats(), 饼图)
也可以通过饼图研究分类变量之间的相互作用:
set.seed(123)
ggpiestats(
data = mtcars,
x = am,
y = cyl,
package = "wesanderson",
palette = "Royal1",
title = "Dataset: Motor Trend Car Road Tests",
legend.title = "Transmission"
)
相关性
7. 两个变量之间的相关性(ggscatterstats(), 散点图)
散点图探索两个变量之间的相关性,其边缘添加直方图用于显示数据的分布。
ggscatterstats(
data = mtcars,
x = disp,
y = mpg,
title = "The relationship between disp and mpg"
)
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.
## `stat_bin()` using `bins = 30`. Pick better value with `binwidth`.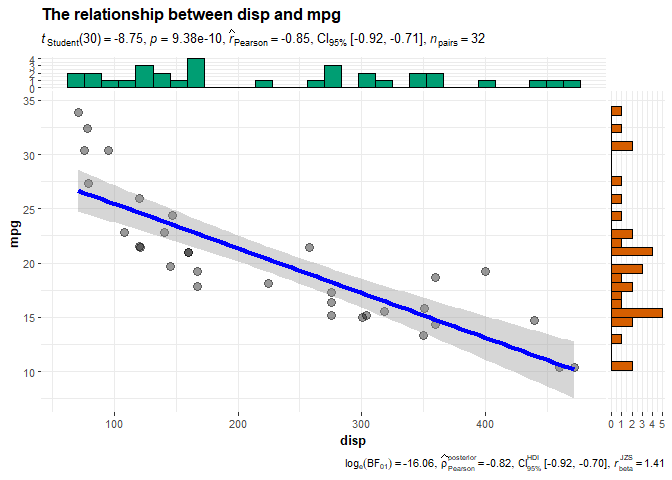
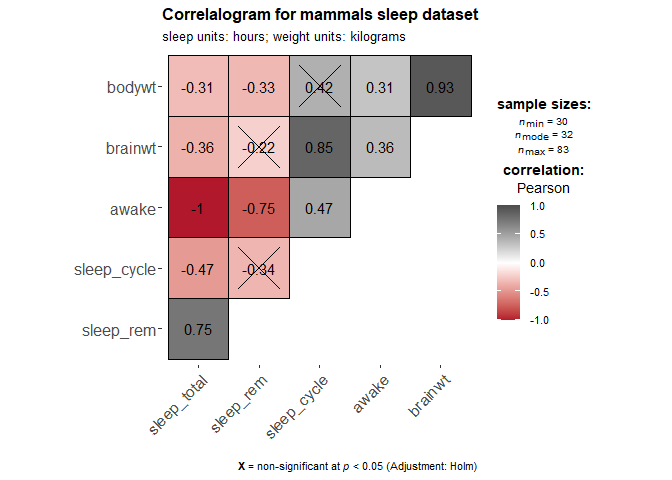
8. 多个变量之间的相关性(ggcorrmat(), 相关性矩阵图)
对于多个变量,可以用相关性矩阵图展示它们之间的关系。
set.seed(123)
## as a default this function outputs a correlation matrix plot
ggcorrmat(
data = ggplot2::msleep,
colors = c("#B2182B", "white", "#4D4D4D"),
title = "Correlalogram for mammals sleep dataset",
subtitle = "sleep units: hours; weight units: kilograms"
)
回归
9. 回归模型和 meta 分析(ggcoefstats(), 点须图)
点须图:回归系数的点估计显示为点,置信区间显示为须,其他统计信息则显示为标签。
set.seed(123)
## model
mod <- stats::lm(formula = mpg ~ am * cyl, data = mtcars)
ggcoefstats(mod)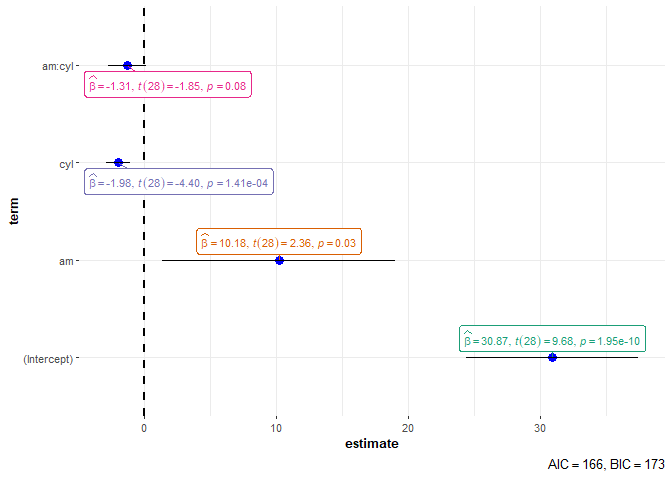
提取统计信息
ggstatsplot 图形中的统计信息可以通过一些方便的函数提取出来,比如:
set.seed(123)
p <- ggbetweenstats(mtcars, cyl, mpg)
extract_subtitle(p)
## list(italic("F")["Welch"](2, 18.03) == "31.62", italic(p) ==
## "1.27e-06", widehat(omega["p"]^2) == "0.74", CI["95%"] ~
## "[" * "0.53", "1.00" * "]", italic("n")["obs"] == "32")
extract_caption(p)
## list(log[e] * (BF["01"]) == "-14.92", widehat(italic(R^"2"))["Bayesian"]^"posterior" ==
## "0.71", CI["95%"]^HDI ~ "[" * "0.57", "0.79" * "]", italic("r")["Cauchy"]^"JZS" ==
## "0.71")
extract_stats(p)
## $subtitle_data
## # A tibble: 1 x 14
## statistic df df.error p.value
##
## 1 31.6 2 18.0 0.00000127
## method effectsize estimate
##
## 1 One-way analysis of means (not assuming equal variances) Omega2 0.744
## conf.level conf.low conf.high conf.method conf.distribution n.obs expression
##
## 1 0.95 0.531 1 ncp F 32
##
## $caption_data
## # A tibble: 6 x 17
## term pd prior.distribution prior.location prior.scale bf10
##
## 1 mu 1 cauchy 0 0.707 3008850.
## 2 cyl-4 1 cauchy 0 0.707 3008850.
## 3 cyl-6 0.780 cauchy 0 0.707 3008850.
## 4 cyl-8 1 cauchy 0 0.707 3008850.
## 5 sig2 1 cauchy 0 0.707 3008850.
## 6 g_cyl 1 cauchy 0 0.707 3008850.
## method log_e_bf10 effectsize estimate std.dev
##
## 1 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## 2 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## 3 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## 4 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## 5 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## 6 Bayes factors for linear models 14.9 Bayesian R-squared 0.714 0.0503
## conf.level conf.low conf.high conf.method n.obs expression
##
## 1 0.95 0.574 0.788 HDI 32
## 2 0.95 0.574 0.788 HDI 32
## 3 0.95 0.574 0.788 HDI 32
## 4 0.95 0.574 0.788 HDI 32
## 5 0.95 0.574 0.788 HDI 32
## 6 0.95 0.574 0.788 HDI 32
##
## $pairwise_comparisons_data
## # A tibble: 3 x 9
## group1 group2 statistic p.value alternative distribution p.adjust.method
##
## 1 4 6 -6.67 0.00110 two.sided q Holm
## 2 4 8 -10.7 0.0000140 two.sided q Holm
## 3 6 8 -7.48 0.000257 two.sided q Holm
## test expression
##
## 1 Games-Howell
## 2 Games-Howell
## 3 Games-Howell
##
## $descriptive_data
## NULL
##
## $one_sample_data
## NULL
##
## $tidy_data
## NULL
##
## $glance_data
## NULL
将统计信息应用于个性化图形
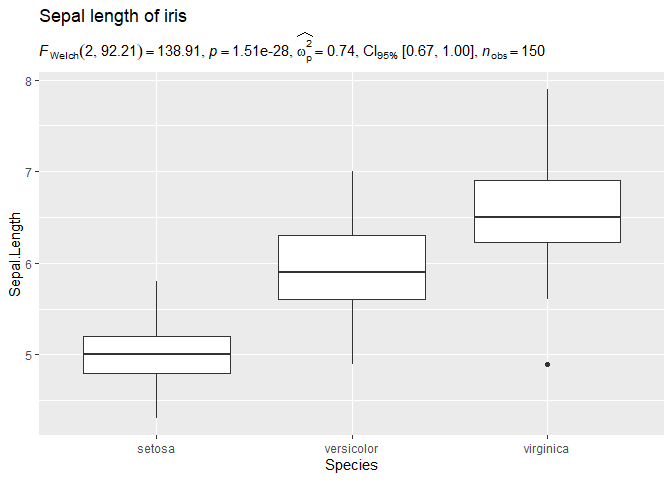
提取出来的统计信息,可以绘制到其他作图系统创建的图形上,例如:
## loading the needed libraries
set.seed(123)
library(ggplot2)
## using `{ggstatsplot}` to get expression with statistical results
stats_results <- ggbetweenstats(iris, Species, Sepal.Length) %>% extract_subtitle()
## creating a custom plot of our choosing
ggplot(iris, aes(x = Species, y = Sepal.Length)) +
geom_boxplot() +
labs(
title = "Sepal length of iris",
subtitle = stats_results,
)
总结
ggstatsplot 确实为探索性数据分析带来了极大的便利,其优点有:
统计作图一个包搞定,无需使用其他大量的软件包。
易于使用,所有函数都只需要少量代码(通常只需要指定 data, x 和 y 即可),这可以极大地减少错误。
各种统计方法可以任选。
独立的图形就可以让人看懂,无需上下文信息。
本文首发于公众号:简说基因,欢迎关注。
