Python科学计算包MNE——头模型和前向计算
目录
- 前言
- 一. Freesurfer安装及配置
-
- 1.1 Freesurfer下载安装
- 1.2 Freesurfer功能测试
- 二. 计算和可视化BEM表面
-
- 2.1 创建BEM的surfer
- 三. 可视化配准
- 四. 计算源空间
-
- 4.1 源空间定义
- 4.2 设置源空间
- 五. 计算正向解
前言
- mne是一款用于处理神经信号的Python 科学计算包,其中所有的示例数据集都是来自同一个机构中来自 60 通道电极帽的 EEG 数据与 MEG 同时获取的,因此实际脑电帽采集的数据由于电极方案和通道数与示例数据集不同,需要在示例代码的基础上做适配。
- 其中示例数据集的采集实验为以下设置:
在这个实验中,棋盘图案呈现给受试者的左右视野,左耳或右耳穿插着音调。刺激之间的间隔为 750 毫秒。视野中央时不时出现一张笑脸。受试者被要求在面部出现后尽快用右手食指按下一个键。 - 数据集主要包括两个目录:
MEG/sample(MEG/EEG 数据)和subjects/sample(MRI 重建)。
数据格式及其说明:
| 文件 | 内容 |
|---|---|
| sample/audvis_raw.fif | 原始 MEG/EEG 数据 |
| audvis.ave | 离线平均的模板脚本 |
| auvis.cov | 用于计算噪声协方差矩阵的模板脚本 |
| 文件 | 内容 |
|---|---|
| bem | 正演建模数据目录 |
| bem/watershed | 使用分水岭算法计算的 BEM 表面分割数据 |
| bem/inner_skull.surf | BEM 的颅骨内表面 |
| bem/outer_skull.surf | BEM 的外颅骨表面 |
| bem/outer_skin.surf | BEM 的皮肤表面 |
| sample-head.fif | 用于 mne_analyze 可视化的 fif 格式的皮肤表面 |
| surf | 表面重建 |
| mri/T1 | 可视化中使用的 T1 加权 MRI 数据 |
示例数据集中已经完成了以下预处理步骤:
- 使用 FreeSurfer 软件计算了 MRI 表面重建。
- BEM 表面已使用分水岭算法创建,请参阅使用分水岭算法。
- fsaverage是基于 40 个真实大脑 MRI 扫描组合的模板大脑。主题fsaverage 文件夹包含正常主题重建会产生的所有文件。最常见的用途之一fsaverage是作为皮质变形/源估计转换的目标空间。换句话说,通常将每个个体受试者的大脑活动估计值变形到fsaverage大脑上,以便可以进行组级统计比较。
环境:
Ubuntu 20.04
AMD5800 8core 16Thread
NVIDIA RTX 3090 24GB
RAM 64GB
一. Freesurfer安装及配置
Freesurfer 是1999年在美国麻省总医院开发的大脑分析与可视化软件。
开发初衷是重建大脑皮层表面,主要用于结构像,功能像和弥散像等数据的分析和可视化。其只能运行在Linux和MAC os上,Windows系统中需要使用虚拟机。
1.1 Freesurfer下载安装
- ubuntu用户首先到Freesurfer官网下载deb安装包:
https://surfer.nmr.mgh.harvard.edu/pub/dist/freesurfer/7.3.2/freesurfer_ubuntu20-7.3.2_amd64.deb
执行以下命令进行安装:
sudo apt update
#首先安装所需要的依赖
sudo apt-get -f install
sudo dpkg -i freesurfer_ubuntu20-7.3.2_amd64.deb
如果产生如下图依赖错误,需要首先卸载之前安装的包,然后重新安装。

sudo apt remove ./free*
安装成功后:
deb模式安装将默认安装在/usr/local/freesurfer 目录下。
- 也可以下载压缩包直接离线解压安装:
https://surfer.nmr.mgh.harvard.edu/pub/dist/freesurfer/7.3.2/freesurfer-linux-ubuntu20_amd64-7.3.2.tar.gz
tar -zxv -f freesurfer-linux-ubuntu20_amd64-7.3.2.tar.gz
sudo apt-get install tcsh
#根据解压的路径设置文件夹权限
sudo chmod -R 777 ./freesurfer
3.通过以上两种的任意一种安装方式安装freesurfer后,需要添加环境变量,这里需要修改为自己电脑freesurfer解压的路径:
使用 gedit ~/.bashrc 打开系统环境文件,在~/.bashrc文件中添加如下两句:
export FREESURFER_HOME=/home/geek/freesurfer #freesurfer解压路径
source $FREESURFER_HOME/SetUpFreeSurfer.sh
export SUBJECTS_DIR=/home/geek/Brain/MRI #处理结果保存路径
source ~/.bashrc #环境修改立即生效
1.2 Freesurfer功能测试
安装完毕以后要想正常使用,还需要到官网获取license证书,具体方式如下:
到官网注册邮箱:
https://surfer.nmr.mgh.harvard.edu/registration.html
注册完毕后查看邮箱下载license.txt:

然后将下载下来的license.txt文件复制到usr/local/freesurfer中:
sudo cp ./license.txt usr/local/freesurfer
执行一个demo查看重建结果:
my_subject=sample
my_NIfTI=/home/geek/Brain/MRI/NIfTI.nii.gz
recon-all -i $my_NIfTI -s $my_subject -all
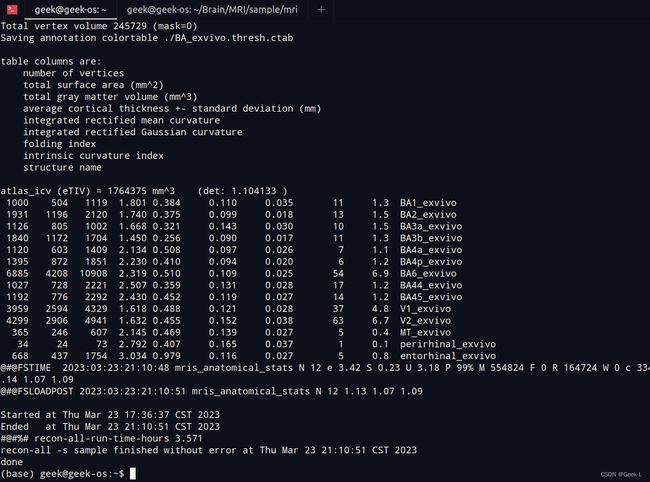
如果运行完毕后没有任何报错,正常输出重建信息,则表明重建成功:
ps: 解剖重建可能需要几个小时,即使在快速计算机上也是如此。重建成功后的终端信息如下:
二. 计算和可视化BEM表面
BEM全称是Boundary Element Model。BEM由定义头部组织隔室的表面组成,如内颅骨,外颅骨和外头皮。
2.1 创建BEM的surfer
安装mne后,可以直接使用mne自带的分水岭算法创建BEM表面:
mne watershed_bem -s sample
创建结束后的输出如下:

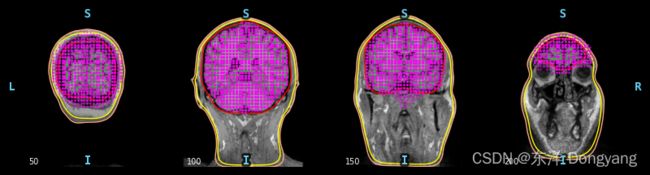
接下来对创建后的BEM表面进行测试:
import mne
subject = 'sample'
plot_bem_kwargs = dict(
subject=subject, subjects_dir="/home/geek/Brain/MRI",
brain_surfaces='white', orientation='coronal',
slices=[50, 100, 150, 200])
mne.viz.plot_bem(**plot_bem_kwargs)
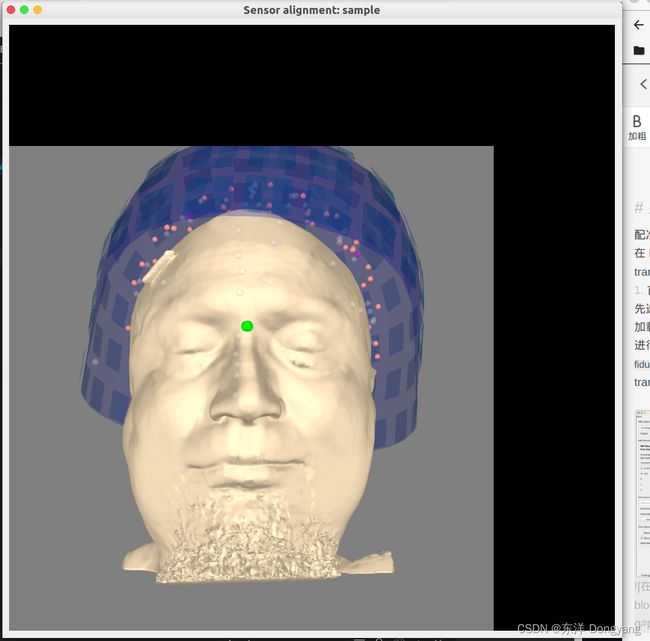
三. 可视化配准
配准是允许将头部和传感器定位在公共坐标系中的操作。在 MNE 软件中,对齐头部和传感器的转换存储在所谓的trans 文件中。
- 首先使用mne中的coregister的GUI工具进行手工配准,先选择MRI的subject目录,然后进行选择信息源文件进行加载,我这里以
sample_audvis_filt-0-40_raw.fif文件为例,进行加载,得到头模型。然后在右侧工具栏进行FIt fiducials和Fit ICP的拟合。最后点击save选择保存的trans.fif文件路径即可。
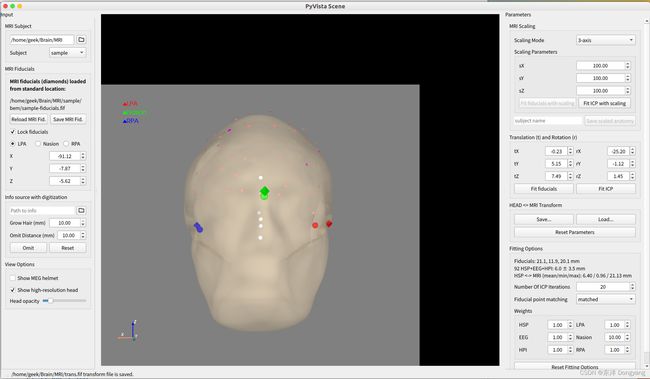
- 然后加载生成的trans.fif文件,绘制配准后的结果,绘制代码为
trans = '/home/geek/Brain/MRI/trans.fif'
info = mne.io.read_info(raw_fname)
# Here we look at the dense head, which isn't used for BEM computations but
# is useful for coregistration.
mne.viz.plot_alignment(info, trans, subject=subject, dig=True,
meg=['helmet', 'sensors'], subjects_dir=subjects_dir,
surfaces='head-dense')
Read a total of 3 projection items:
PCA-v1 (1 x 102) idle
PCA-v2 (1 x 102) idle
PCA-v3 (1 x 102) idle
Using lh.seghead for head surface.
Getting helmet for system 306m
Channel types:: grad: 203, mag: 102, eeg: 59
.viz.backends._pyvista.PyVistaFigure at 0x1460e0333670
四. 计算源空间
4.1 源空间定义
源空间定义候选源位置的位置和方向。有两种类型的源空间:
- 当候选者被限制在一个表面时,基于表面的源空间。
- 体积或离散源空间,当候选者是离散的、任意位于表面边界的源点时。
基于表面的源空间src包含两部分,一部分用于左半球(258 个位置),另一部分用于右半球(258 个位置)。源可以在紫色的 BEM 表面上可视化。

计算以 (0.0, 0.0, 40.0) mm 为中心的半径为 90mm 的球体内的候选偶极子网格定义的基于体积的源空间:
sphere = (0.0, 0.0, 0.04, 0.09)
vol_src = mne.setup_volume_source_space(
subject, subjects_dir=subjects_dir, sphere=sphere, sphere_units='m',
add_interpolator=False) # just for speed!
print(vol_src)
mne.viz.plot_bem(src=vol_src, **plot_bem_kwargs)
4.2 设置源空间
这个阶段包括以下内容:
- 在白质表面创建合适的抽取偶极子网格。
- 创建 fif 格式的源空间文件。
计算基于大脑内部候选偶极子网格定义的基于体积的源空间(需要BEM表面):
surface = "/home/geek/Brain/MRI/sample/bem/inner_skull.surf"
vol_src = mne.setup_volume_source_space(
subject, subjects_dir=subjects_dir, surface=surface,
add_interpolator=False) # Just for speed!
print(vol_src)
mne.viz.plot_bem(src=vol_src, **plot_bem_kwargs)
3D 方式查看所有源:

请注意,BEM不涉及对 trans 文件的任何使用。BEM 仅取决于头部几何形状和电导率。因此它独立于 MEG 数据和头部位置。
五. 计算正向解
现在让我们计算正向运算符,通常称为增益或前场矩阵。
在设置 MRI-MEG/EEG 对齐后,正向解,即由于位于皮层上的偶极源,测量传感器和电极处的磁场和电势,可以通过以下方式计算
fwd = mne.make_forward_solution(raw_fname, trans=trans, src=src, bem=bem,
meg=True, eeg=False, mindist=5.0, n_jobs=None,
verbose=True)
mne.write_forward_solution("/home/geek/Brain/MRI/fwd.fif",fwd)
print(fwd)