稳健回归 : RANSACRegressor随机采样一致性算法和TheilSenRegressor泰尔森回归
最小二乘法的弊端
之前文章里的关于线性回归的模型,都是基于最小二乘法来实现的。但是,当数据样本点出现很多的异常点(outliers),这些异常点对回归模型的影响会非常的大,传统的基于最小二乘的回归方法将不适用。
当然,可以考虑在做回归分析之前,对数据做预处理,剔除掉那些异常点。但是,在实际的数据中,存在两个问题:
- 异常点并不能很好的确定,并没有一个很好的标准用于确定哪些点是异常点
- 即便确定了异常点,但这些被确定为异常的点,真的是错误的数据吗?很有可能这看似异常的点,就是原始模型的数据,如果是这样的话,那么这些异常的点就会带有大量的原始模型的信息,剔除之后就会丢失大量的信息。
稳健回归
稳健回归(Robust regression),就是当最小二乘法遇到上述的,数据样本点存在异常点的时候,用于代替最小二乘法的一个算法。当然,稳健回归还可以用于异常点检测,或者是找出那些对模型影响最大的样本点。
Breakdown point
关于稳健回归,有一个名词需要做解释:Breakdown point,这个名词我并不想翻译,我也没找到一个很好的中文翻译。对于一个估计器而言,原始数据中混入了脏数据,那么,Breakdown point 指的就是在这个估计器给出错误的模型估计之前,脏数据最大的比例 α,Breakdown point 代表的是一个估计器对脏数据的最大容忍度。
举个简单的例子:有 n 个随机变量,(X1,X2,…,Xn), 其对应的数据为(x1,x2,…,xn),那么,我么可以求出这 n 个随机变量的均值:
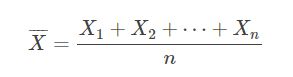
这个均值估计器的Breakdown point 为0,因为使任意一个xi变成足够大的脏数据之后,上面估计出来的均值,就不再正确了。
毫无疑问,Breakdown point越大,估计器就越稳健。
Breakdown point 是不可能达到 50% 的,因为如果总体样本中超过一半的数据是脏数据了,那么从统计上来说,就无法将样本中的隐藏分布和脏数据的分布给区分开来。
本文主要介绍两种稳健回归模型:RANSAC(RANdom SAmple Consensus 随机采样一致性)和Theil-Sen estimator。
RANSAC随机采样一致性算法
RANSAC算法的输入是一组观测数据(往往含有较大的噪声或无效点),它是一种重采样技术(resampling technique),通过估计模型参数所需的最小的样本点数,来得到备选模型集合,然后在不断的对集合进行扩充,其算法步骤为:
- 随机的选择估计模型参数所需的最少的样本点。
- 估计出模型的参数。
- 找出在误差 ϵ 内,有多少点适合当前这个模型,并将这些点标记为模型内点
- 如果内点的数目占总样本点的比例达到了事先设定的阈值 τ,那么基于这些内点重新估计模型的参数,并以此为最终模型, 终止程序。
- 否则重复执行1到4步。
RANSAC算法是从输入样本集合的内点的随机子集中学习模型。
RANSAC算法是一个非确定性算法(non-deterministic algorithm),这个算法只能得以一定的概率得到一个还不错的结果,在基本模型已定的情况下,结果的好坏程度主要取决于算法最大的迭代次数。
RANSAC算法在线性和非线性回归中都得到了广泛的应用,而其最典型也是最成功的应用,莫过于在图像处理中处理图像拼接问题,这部分在Opencv中有相关的实现。
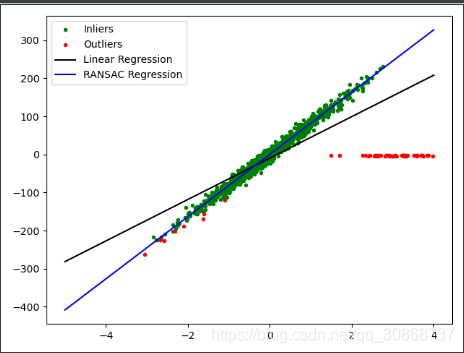
从总体上来讲,RANSAC算法将输入样本分成了两个大的子集:内点(inliers)和外点(outliers)。其中内点的数据分布会受到噪声的影响;而外点主要来自于错误的测量手段或者是对数据错误的假设。而RANSAC算法最终的结果是基于算法所确定的内点集合得到的。
下面这份代码是RANSAC的适用实例:
# -*- coding: utf-8 -*-
import numpy as np
import time
from sklearn import linear_model,datasets
import matplotlib.pyplot as plt
# 产生数据样本点集合
# 样本点的特征X维度为1维,输出y的维度也为1维
# 输出是在输入的基础上加入了高斯噪声N(0,10)
# 产生的样本点数目为1000个
n_samples = 1000
X, y, coef = datasets.make_regression(n_samples=n_samples,
n_features=1,
n_informative=1,
noise=10,
coef=True,
random_state=0)
# 将上面产生的样本点中的前50个设为异常点(外点)
# 即:让前50个点偏离原来的位置,模拟错误的测量带来的误差
n_outliers = 50
np.random.seed(int(time.time()) % 100)
X[:n_outliers] = 3 + 0.5 * np.random.normal(size=(n_outliers, 1))
y[:n_outliers] = -3 + 0.5 * np.random.normal(size=n_outliers)
# 用普通线性模型拟合X,y
model = linear_model.LinearRegression()
model.fit(X, y)
# 使用RANSAC算法拟合X,y
model_ransac = linear_model.RANSACRegressor(linear_model.LinearRegression())
model_ransac.fit(X, y)
inlier_mask = model_ransac.inlier_mask_
outlier_mask = np.logical_not(inlier_mask)
# 使用一般回归模型和RANSAC算法分别对测试数据做预测
line_X = np.arange(-5, 5)
line_y = model.predict(line_X[:, np.newaxis])
line_y_ransac = model_ransac.predict(line_X[:, np.newaxis])
print ("真实数据参数:", coef)
print ("线性回归模型参数:", model.coef_)
print ("RANSAC算法参数: ", model_ransac.estimator_.coef_)
plt.plot(X[inlier_mask], y[inlier_mask], '.g', label='Inliers')
plt.plot(X[outlier_mask], y[outlier_mask], '.r', label='Outliers')
plt.plot(line_X, line_y, '-k', label='Linear Regression')
plt.plot(line_X, line_y_ransac, '-b', label="RANSAC Regression")
plt.legend(loc='upper left')
plt.show()
运行结果为:
真实数据参数: 82.1903908408
线性回归模型参数: [ 55.19291974]
RANSAC算法参数: [ 82.08533159]
Theil-Sen Regression 泰尔森回归
Theil-Sen回归是一个参数中值估计器,它适用泛化中值,对多维数据进行估计,因此其对多维的异常点(outliers 外点)有很强的稳健性。
一般的回归模型为:y=α+βx+ϵ
其中,α,β 模型的参数,而 ϵ 为模型的随机误差。
Theil-Sen回归则是这么处理的:

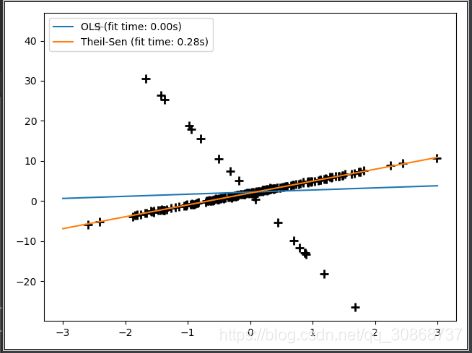
在实践中发现,随着数据特征维度的提升,Theil-Sen回归的效果不断的下降,在高维数据中,Theil-Sen回归的效果有时甚至还不如OLS(最小二乘)。
在之间的文章《线性回归》中讨论过,OLS方法是渐进无偏的,Theil-Sen方法在渐进无偏方面和OLS性能相似。和OLS方法不同的是,Theil-Sen方法是一种非参数方法,其对数据的潜在分布不做任何的假设。Theil-Sen方法是一种基于中值的估计其,所以其对异常点有更强的稳健性。
在单变量回归问题中,Theil-Sen方法的Breakdown point为29.3%,也就是说,Theil-Sen方法可以容忍29.3%的数据是outliers。
# -*- coding: utf-8 -*-
"""
Theil-Sen 回归
本例生成一个数据集,然后在该数据集上测试Theil-Sen回归
"""
import time
import numpy as np
import matplotlib.pyplot as plt
from sklearn.linear_model import LinearRegression, TheilSenRegressor,RANSACRegressor
estimators = [('OLS', LinearRegression()),
('Theil-Sen', TheilSenRegressor())]
# 异常值仅仅出现在y轴
np.random.seed((int)(time.time() % 100))
n_samples = 200
# 线性模型的函数形式为: y = 3 * x + N(2, .1 ** 2)
x = np.random.randn(n_samples)
w = 3.
c = 2.
noise = c + 0.1 * np.random.randn(n_samples)
y = w * x + noise
# 加入10%的异常值,最后20个值称为异常值
y[-20:] += -20 * x[-20:]
X = x[:, np.newaxis]
plt.plot(X, y, 'k+', mew=2, ms=8)
line_x = np.array([-3, 3])
for name, estimator in estimators:
t0 = time.time()
estimator.fit(X, y)
elapsed_time = time.time() - t0
y_pred = estimator.predict(line_x.reshape(2, 1))
plt.plot(line_x, y_pred, label='%s (fit time: %.2fs)'
%(name, elapsed_time))
plt.axis('tight')
plt.legend(loc='upper left')
plt.show()