医疗图像论文学习
医学图像分割
DoDNet:Learning to segment multi-organ and tumors from multiple partially labeled datasets(2021)
DoDNet,一个具有动态头的单一编码器-解码器网络,用来解决腹部 CT 扫描中多器官和肿瘤分割的部分标记问题。还创建一个大规模部分标记数据集MOTS,并对它进行了广泛的实验。
结果表明,受益于任务编码和动态滤波学习,DoDNet 不仅在七个器官和肿瘤分割任务上取得了最佳的整体性能,而且推理速度也高于其他竞争对手。另外,还证明了 DoDNet 和 MOTS 数据集的价值,并成功地将在 MOTS 上预训练的权重迁移到只有有限标注的下游任务中。也表明这项工作的副产品(即预训练的三维网络)有利于其他小样本的三维医学图像分割任务。
问题提出
部分标记医学图像分割多器官和肿瘤分割是医学图像分析中普遍存在的困难,特别是在没有大规模全标记数据集的情况下。虽然有几个部分标记的数据集可用,但每个数据集都专门用于一个特定器官和/或肿瘤的分割。
因此,分割模型通常在一个部分标记的数据集上进行训练,因此只能分割一个特定的器官和肿瘤,如肝脏和肝脏肿,肾脏和肾脏肿瘤。然而,训练多个网络会导致计算资源的浪费和可扩展性较差。
前人研究
为了解决这个问题,人们已经多次尝试以一种更有效的方式来探索多个部分标记的数据集。
Chen等人从不同的医疗领域收集了多个部分标记的数据集,并在它们上共同训练了一个异构的3D网络,该网络是专门设计的任务共享编码器和8个分割任务的任务特定解码器。
Huang等人提出在模型上共同训练一对权重平均模型,用于少器官数据集的统一多器官分割。
Zhou等人首先在一个完全标记的数据集上近似出腹部器官大小的解剖先验,然后在几个部分标记的数据集上规范化器官大小分布。
Fang等人将以未知标签的体素作为背景,提出了在多个部分标记数据集上训练的分割网络的目标自适应损失(TAL)。
Shi等人将未标记的器官与背景合并,并对每个体素(即每个体素属于一个体素中的一个器官或背景)施加独家约束,在一个完全标记的数据集和几个部分标记的数据集上联合学习分割模型。
为了从单类数据集中学习多类分割,Dmitriev等人将分割任务作为先验,并将其纳入中间激活信号中。
创新点
-
将部分标签(一部分是打了标签的数据集)的问题作为一个多类分割任务,并将未标记的器官作为背景,这可能会产生误导,因为在该数据集中未标记的器官确实是另一项任务的前景。为了解决这个问题,我们将部分标记的问题制定为一个单类的分割任务,目的是分别分割每个器官;
-
之前那些方法大多采用多头结构,由共享的骨干网络和针对不同任务的多个分割头组成。每个头要么是一个解码器[3],要么是最后一个分割层[9,30]。相比之下,所提出的DoDNet是一个单头网络,其中的头是灵活的和动态的;
-
我们的DoDNet使用动态分割头来解决部分标记的问题,而不是将任务之前嵌入编码器和解码器;
-
现有的方法大多侧重于多器官分割,而我们的DoDNet将器官和肿瘤分割,这更具挑战性。
网络设计
有三种方法来执行m的部分标记分割任务。
-
(a)多网络:分别在m个部分标记子集上训练m个网络;
-
(b)多头网络:训练一个由共享编码器和m个任务特定解码器(头)组成的网络,每个网络执行部分标记的分割任务;
-
(c )提出的DoDNet:它有一个编码器、一个任务编码模块、一个动态过滤器生成模块和一个动态分割头。动态头部中的内核以输入图像和分配的任务为条件。
代码实现
Learning Calibrated Medical Image Segmentation via Multi-rater Agreement Modeling(2021)
在医学图像分析中,通常会收集多个注释,每个注释都来自不同的临床专家或评分者,以期望能够减轻可能的诊断错误。
同时,从计算机视觉从业者的角度来看,采用通过多数投票或简单的首选评分者获得的真相标签是一种常见的做法。
然而,这个过程往往忽略了原始多评估者注释中根深蒂固的协议或不一致的丰富信息。为了解决这个问题,我们建议明确地建模多评分者(dis-)协议,称为MRNet,它有两个主要贡献。
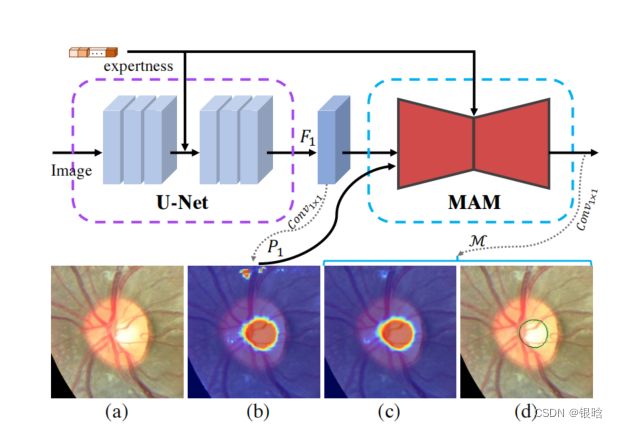
- 首先,设计了一个专家感知推理模块或EIM,将个体评分者的专业水平作为先验知识嵌入,形成高级语义特征。
- 其次,我们的方法能够从粗糙的预测中重建多评分者分级,并进一步利用多评分者(非)协议线索来提高分割性能。据我们所知,我们的工作是第一次在不同的专业知识水平下对医学i产生经过校准的预测
- 说白了就是,先大致的检测出来目标区域在哪,进行一个粗略的判断,然后针对性的放大这个区域再进行检测
模型描述
MRNet框架:
-
(a)处理管道的概述,并继续放大图的各个模块
-
(b)专业意识推断模块(EIM)
-
(c )多评分者协议建模(MAM)由多评分者重建模块(MRM)和多评分者感知模块(MPM)。
MR Image Super-Resolution with Squeeze and Excitation Reasoning Attention Network(2021)
-
MR图像通常具有一些共同的视觉特征:重复的模式、相对简单的结构和信息量较少的背景。Mr图像通常包含较大的背景区域,其信息量远少于目标结构区域(冗余信息)
-
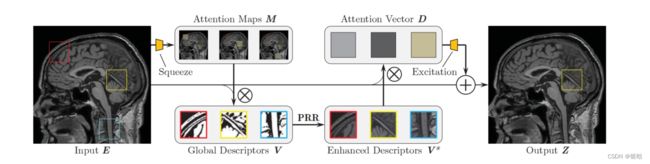
为了解决这些问题,我们提出挤压激励(squeeze and excitation)推理注意力网络(SERAN)用于精确的MR图像SR。
- 我们从全局空间信息输入挤压注意力,获取全局描述符。这种全局描述符增强了网络关注MR图像中更多信息区域和结构的能力。
- 它允许模型聚焦于Mr图像中信息更丰富的区域和结构。我们进一步建立了全局描述符之间的关系,即建立原语之间的关系,并应用图卷积网络(GCN)进行推理,获得原语关系的推理注意。
- 全局描述符通过学习的注意力进一步细化。为了充分利用聚合信息,我们使用学习的自适应注意力向量自适应地重校准特征响应。这些注意力向量选择一个全局描述子子集来补充每个空间位置,以实现精确的细节和纹理重建。
简要说明了我们的挤压和兴奋注意机制。
与[3]类似,首先通过双线性池化收集全局特征,然后通过考虑相应的局部特征分布到每个空间位置。然而,我们通过原始关系推理(PRR)来增强全局特征
首先,这里直接使用残差学习将使训练过程在数值上不稳定。
其次,残差连接允许我们将SEAB插入任何预训练网络,而不会过多地影响其初始行为.
随着SEAB的使用,即使有限的感受野大小,随后的卷积层也可以感知整个空间。
SEAB允许网络专注于更多信息的视觉特征,并实现更好的MR图像SR重建质量。
GCN卷积提取注意力的过程
自适应注意力重构图像
收集全局特征描述符后,我们希望将它们分发到原始特征的每个位置。这将有助于我们更好地利用与计算的二阶统计量的复杂关系,并补偿丢失的信息以获得更好的MR图像重建。

我们可以看到原始特征的每个位置都有其对全局描述符的特定需求。我们根据学习的注意向量 d i d_i di , 在每个位置自适应地分配全局描述符V 。这意味着每个位置都可以自适应地选择互补的视觉原语义。
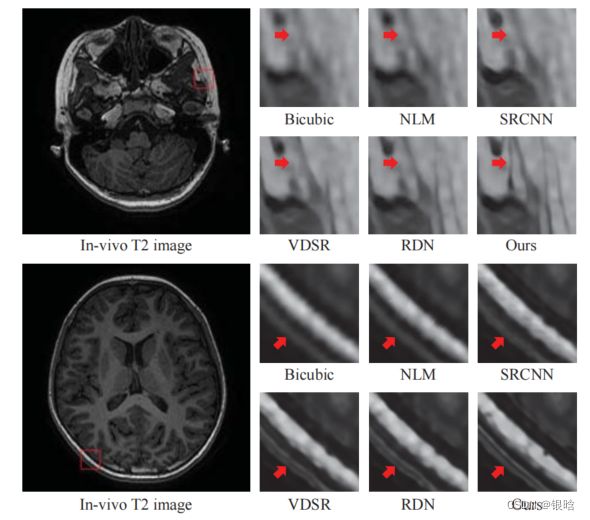
- 最终效果
Preservational Learning Improves Self-supervised Medical Image Models by Reconstructing Diverse Contexts
保留最大的信息是设计自监督学习方法的原则之一。
为了达到这一目标,对比学习采用了内隐式的对比图像对。对比学习的目标是通过对比医学图像对来学习不变表示,这可以看作是一种保持最大信息的隐式方法。
然而,我们认为简单地使用对比估计来保存并不是完全最优的。我们认为除了对比损失之外,明确地保留更多的信息仍然是有益的和互补的。
从这个角度来看,我们引入保存学习来重建不同的图像环境,以便在学习的表示中保留更多的信息。
一个直观的解决方案是使用学习到的表示来重建原始输入,以便这些表示可以保存与输入密切相关的信息。然而,我们发现直接添加一个普通的重建分支来恢复原始输入并不会显著改善学习到的表示。为了解决这个问题,我们引入了保留性对比表示学习,利用从对比损失中学习到的表示来重建不同的上下文。
结合对比损失,我们提出了保守性对比表示学习(PCRL),用于学习自我监督的医学表征。
PCRL在预训练-微调协议下提供了非常有竞争力的结果,在5个分类/分割任务中大大优于自我监督和有监督的对应的结果。
本文的贡献可以概括为三个方面:
- 引入保守性对比性表示学习,通过重构不同的上下文,将更多的信息编码到从对比性损失中学习到的表示中。
- 为了恢复不同的图像,我们提出了两个模块:转换条件注意和交叉模型混合,以建立一个三重编码器,单解码器的自监督学习架构。
- 广泛的实验和分析表明,所提出的PCRL在5个分类/分割任务中具有明显的优势,在显著方面优于自监督和监督任务。
前人研究
-
通过图像旋转度,对象颜色,对象数[25]和应用的变换函数[30]。基于对比估计的方法也利用借口任务,通过对比图像对来学习不变表示。最近,有一些研究试图去除对比学习中的负对。相比之下,我们的方法遵循了一个不同的原则,即使表示法能够完全描述它们的来源(即相应的输入图像)。
-
医学图像分析中的自监督学习。在对比学习之前,解决拼图问题[54,53,35]和重建损坏的图像[9,52]是医学图像中基于借口的方法的两个主要课题。除此之外,Xie等人[44]还在核图像中引入了一种用于自监督学习的三联体损失。Haghigi等[19]通过附加一个分类分支将高级特征分类为不同的解剖模式,改进了[52]。对于对比学习,Zhou等人[51]将对比损失应用于二维x线片。类似的想法也出现在少镜头[49]和半监督学习[50]中。Taleb等人[34]提出了利用三维医学图像的三维对比预测编码。有两个[16,8]与我们最相关的作品。Feng等人的[16]研究表明,部分图像的重建过程与使用对比损失具有相似的效果。[8]等人引入了一种去噪自动编码器来捕获一个潜在的空间表示。然而,这两种方法都未能通过上下文重建来改善对比学习,而我们的方法在这个aspect上取得了成功

我们尝试将不同的图像重建作为借口任务,纳入对比学习中。主要的动机是将更多的信息编码到学习到的表示中。
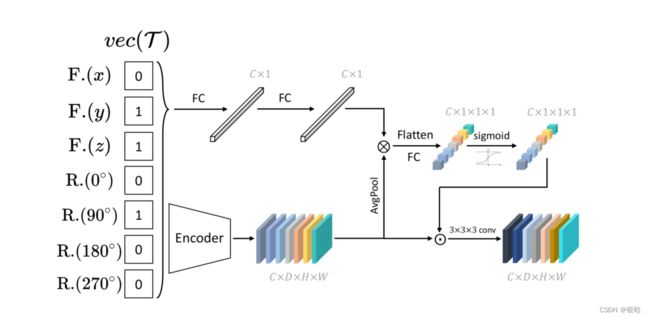
具体地说,我们引入了转换条件注意和交叉模型混合来丰富表示所携带的信息。第一个模块将一个转换指示器向量(vec (T)嵌入到高级特征图中。
基于嵌入式向量,网络需要在输入固定的情况下动态重建不同的图像目标。
- 通过混合普通编码器和动量编码器的特征映射来生成混合编码器,并要求混合编码器重建混合图像目标。
我们表明,与仅使用对比学习相比,这两个模块都可以帮助编码更多的信息,并产生更强的表示。
GO表示将特征映射转换为特征向量的全局操作。蓝色的特征向量来自于动量编码器。vec (T)表示T的指标向量,其中包含一组变换函数。vec (T)中的每个分量均为1或0,表示是应用了相应的变换还是n
模型框架

PCRL采用了一种类似于U-Net的架构来学习表示法。对于编码器和解码器,我们绘制它们的特征图,以更好地演示。
混合编码器不接受输入图像,因为它由来自普通编码器和动量编码器的混合特征映射组成。{C., F., R., I., O., B.} 分别是随机裁剪、随机翻转、随机旋转、内画、外画和高斯模糊的缩写。
NCE是噪声对比估计的缩写。GO表示全局操作,其中包括全局平均池化层和完全连接层。vec(·)表示指标向量。T{o、m、h}(·)表示针对不同编码器的一组转换函数。⊙表示通道级乘法。为简单起见,我们不绘制跳过连接。
- 我们的转换条件下的注意模块。
F. 和R.分别代表翻转和旋转。{x、y、z}表示坐标轴。{0、90°、180°、270°}表示旋转度。vec (T)表示T的指示向量,为简单起见,省略其下标。⊗表示外部产物。⊙表示通道级乘法。请注意,上面的图演示了当每个输入都是3D时的实现。对于二维输入,指示向量中没有F(z)。对于二维和三维输入,旋转只应用于xy平面。
- 通过重建不同的上下文,使用对比损失的学习表示可以大大改善在医学图像分析。我们的方法在各种医疗任务和数据集中显示了自我监督学习的积极结果。
说的nb的不行,我看看你代码行不行
import segmentation_models_pytorch as smp
import torch.nn as nn
import torch.nn.functional as F
import torch
from segmentation_models_pytorch.base import modules as md
import numpy as np
from torchvision.models.resnet import ResNet
from torchvision.models.resnet import BasicBlock
from torchvision.models.resnet import Bottleneck
from pretrainedmodels.models.torchvision_models import pretrained_settings
from segmentation_models_pytorch.base.initialization import initialize_decoder, initialize_head
from segmentation_models_pytorch.base import SegmentationHead
from segmentation_models_pytorch.encoders._base import EncoderMixin
import copy
import random
def initialize_decoder(module):
for m in module.modules():
if isinstance(m, nn.Conv2d):
nn.init.kaiming_uniform_(m.weight, mode="fan_in", nonlinearity="relu")
if m.bias is not None:
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.BatchNorm2d):
nn.init.constant_(m.weight, 1)
nn.init.constant_(m.bias, 0)
elif isinstance(m, nn.Linear):
nn.init.xavier_uniform_(m.weight)
if m.bias is not None:
nn.init.constant_(m.bias, 0)
def initialize_head(module):
for m in module.modules():
if isinstance(m, (nn.Linear, nn.Conv2d)):
nn.init.xavier_uniform_(m.weight)
if m.bias is not None:
nn.init.constant_(m.bias, 0)
class CenterBlock(nn.Sequential):
def __init__(self, in_channels, out_channels, use_batchnorm=True):
conv1 = md.Conv2dReLU(
in_channels,
out_channels,
kernel_size=3,
padding=1,
use_batchnorm=use_batchnorm,
)
conv2 = md.Conv2dReLU(
out_channels,
out_channels,
kernel_size=3,
padding=1,
use_batchnorm=use_batchnorm,
)
super().__init__(conv1, conv2)
class DecoderBlock(nn.Module):
def __init__(
self,
in_channels,
skip_channels,
out_channels,
use_batchnorm=True,
attention_type=None,
):
super().__init__()
self.conv1 = md.Conv2dReLU(
in_channels + skip_channels,
out_channels,
kernel_size=3,
padding=1,
use_batchnorm=use_batchnorm,
)
self.attention1 = md.Attention(attention_type, in_channels=in_channels + skip_channels)
self.conv2 = md.Conv2dReLU(
out_channels,
out_channels,
kernel_size=3,
padding=1,
use_batchnorm=use_batchnorm,
)
self.attention2 = md.Attention(attention_type, in_channels=out_channels)
def forward(self, x, skip=None):
x = F.interpolate(x, scale_factor=2, mode="nearest")
if skip is not None:
x = torch.cat([x, skip], dim=1)
x = self.attention1(x)
x = self.conv1(x)
x = self.conv2(x)
x = self.attention2(x)
return x
class ShuffleUnetDecoder(nn.Module):
def __init__(
self,
# decoder,
encoder_channels=512,
n_class=3,
decoder_channels=(256, 128, 64, 32, 16),
n_blocks=5,
use_batchnorm=True,
center=False,
attention_type=None
):
super().__init__()
# self.decoder = decoder
# self.segmentation_head = segmentation_head
if n_blocks != len(decoder_channels):
raise ValueError(
"Model depth is {}, but you provide `decoder_channels` for {} blocks.".format(
n_blocks, len(decoder_channels)
)
)
encoder_channels = encoder_channels[1:] # remove first skip with same spatial resolution
encoder_channels = encoder_channels[::-1] # reverse channels to start from head of encoder
# computing blocks input and output channels
head_channels = encoder_channels[0]
in_channels = [head_channels] + list(decoder_channels[:-1])
skip_channels = list(encoder_channels[1:]) + [0]
out_channels = decoder_channels
# self.conv = nn.Conv2d(1024, 512, kernel_size=3, padding=1, stride=1)
if center:
self.center = CenterBlock(
head_channels, head_channels, use_batchnorm=use_batchnorm
)
else:
self.center = nn.Identity()
kwargs = dict(use_batchnorm=use_batchnorm, attention_type=attention_type)
blocks = [
DecoderBlock(in_ch, skip_ch, out_ch, **kwargs)
for in_ch, skip_ch, out_ch in zip(in_channels, skip_channels, out_channels)
]
self.blocks = nn.ModuleList(blocks)
initialize_decoder(self.blocks)
# self.segmentation_head = SegmentationHead(16, 3)
# initialize_head(self.segmentation_head)
# self.segmentation_head = segmentation_head
#
# # combine decoder keyword arguments
def forward(self, features1, features2, alpha, aug_tensor1, aug_tensor2, mixup=False):
# x = self.decoder(*features)
# return self.segmentation_head(x)
# def forward(self, features1, features2):
#
features1 = features1[1:] # remove first skip with same spatial resolution
features1 = features1[::-1] # reverse channels to start from head of encoder
features2 = features2[1:]
features2 = features2[::-1]
head1 = features1[0]
skips1 = features1[1:]
head2 = features2[0]
skips2 = features2[1:]
x1 = self.center(head1)
x2 = self.center(head2)
if not mixup:
x1 = x1 * aug_tensor1
x2 = x2 * aug_tensor2
x3 = x1.clone()
x1 = alpha * x1 + (1 - alpha) * x2
for i, decoder_block in enumerate(self.blocks):
# print(i, x1.shape, skips1[i].shape, x2.shape, skips2[i].shape)
skip1 = skips1[i] if i < len(skips1) else None
#skip1_shuffle = self.decoder_shuffle(skip1, shuffle_num + i + 1) if i < len(skips1) else None
x3 = decoder_block(x3, skip1)
# x1 = decoder_block(x1, skip1)
skip2 = skips2[i] if i < len(skips2) else None
skip = alpha * skip1 + (1 - alpha) * skip2 if i < len(skips2) else None
# skip = self.decoder_shuffle(skip, shuffle_num + i + 1) if i < len(skips2) else None
# x2 = decoder_block(x2, skip2)
x1 = decoder_block(x1, skip)
# x1 = self.segmentation_head(x1)
return x1, x3
def decoder_shuffle(self, x, shuffle_num):
w = x.shape[2]
h = x.shape[3]
shuffle_col_index = torch.randperm(w)[:shuffle_num].cuda()
shuffle_row_index = torch.randperm(h)[:shuffle_num].cuda()
col_index = shuffle_col_index[torch.randperm(shuffle_col_index.shape[0])]
row_index = shuffle_row_index[torch.randperm(shuffle_row_index.shape[0])]
# print(col_index, row_index, shuffle_row_index, shuffle_col_index)
# print(shuffle_row_index, x.shape, x[:, :, shuffle_row_index].shape)
x = x.index_copy(2, col_index, x.index_select(2, shuffle_col_index))
x = x.index_copy(3, row_index, x.index_select(3, shuffle_row_index))
return x
class PCRLModel(nn.Module):
def __init__(self, n_class=3, low_dim=128, student=False):
super(PCRLModel, self).__init__()
self.model = smp.Unet('resnet18', in_channels=3, classes=n_class, encoder_weights=None)
self.model.decoder = ShuffleUnetDecoder(self.model.encoder.out_channels)
# self.segmentation_head = self.unet.segmentation_head
# self.model = net
self.avg_pool = nn.AdaptiveAvgPool2d((1, 1))
self.fc1 = nn.Linear(512, low_dim)
self.relu = nn.ReLU(inplace=True)
self.student = student
self.fc2 = nn.Linear(low_dim, low_dim)
self.aug_fc1 = nn.Linear(6, 256)
self.aug_fc2 = nn.Linear(256, 512)
self.sigmoid = nn.Sigmoid()
def forward(self, x, features_ema=None, alpha=None, aug_tensor1=None, aug_tensor2=None, mixup=False):
b = x.shape[0]
features = self.model.encoder(x)
feature = self.avg_pool(features[-1])
feature = feature.view(b, -1)
feature = self.fc1(feature)
feature = self.relu(feature)
feature = self.fc2(feature)
if self.student:
if not mixup:
aug_tensor1 = self.aug_fc1(aug_tensor1)
aug_tensor1 = self.relu(aug_tensor1)
aug_tensor1 = self.aug_fc2(aug_tensor1)
aug_tensor2 = self.aug_fc1(aug_tensor2)
aug_tensor2 = self.relu(aug_tensor2)
aug_tensor2 = self.aug_fc2(aug_tensor2)
aug_tensor1 = self.sigmoid(aug_tensor1)
aug_tensor2 = self.sigmoid(aug_tensor2)
aug_tensor1 = aug_tensor1.view(b, 512, 1, 1)
aug_tensor2 = aug_tensor2.view(b, 512, 1, 1)
# print(aug_tensor2.shape)
decoder_output_alpha, decoder_output = self.model.decoder(features, features_ema, alpha, aug_tensor1,
aug_tensor2, mixup)
masks_alpha = self.model.segmentation_head(decoder_output_alpha)
masks = self.model.segmentation_head(decoder_output)
return feature, masks_alpha, masks
return feature, features
来源:合集