K-均值聚类算法
概述
聚类是一种无监督的学习,他将相似的对象归到同一个簇中,簇内的对象 越相似,聚类的效果越好。聚类与分类的最大不同在于,分类的目标事先巳知,而聚类则不一样。因为其产生的结果与分类相同,而只是类别没有预先定义,聚类有时也被称为无监督分类
K均值聚类的算法:发现k个不同的簇,且每个簇的中心采用簇中所含值计算而成。
K-均值聚类的一般流程
(1)收集数据:使用任意方法。
⑵准备数据:需要数值型数据来计算距离,也可以将标称型数据映射为二值型数据再用
于距离计算。
(3)分析数据:使用任意方法。
(4)训练算法:不适用于无监督学习,即无监督学习没有训练过程。
(5)测试算法:应用聚类算法、观察结果。可以使用量化的误差指标如误差平方和(后面
会介绍)来评价算法的结果。
(6)使用算法:可以用于所希望的任何应用。通常情况下,簇质心可以代表整个簇的数据
来做出决策。
K-均值聚类算法
K-均值算法的工作流程是这样的。1、随机确定&个初始点作为质心。2、将数据集中的每个点分配到一个簇中,具体来讲,为每个点找距其最近的质心,并将其分配给该质心所对应的簇。3、每个簇的质心更新为该簇所有点的平均值。以下代码实现创建K均值聚类:
from numpy import *
def loadDataSet(fileName):
dataMat =[]
fr = open(fileName).readlines()
for line in fr:
curLine = line.strip().split('\t')
# map 两个参数,第一个为函数,第二个为序列
# 即rangeJ = float(max(dataSet[:, j]) - minJ)相减的是两个map类型的数据,经过查找,发现fltLine = map(float, curLine)
# 在python2中返回的是一个list类型数据,而在python3中该语句返回的是一个map类型的数据。
# fltLine = map(float,curLine)
fltLine = list(map(float, curLine))
dataMat.append(fltLine)
return dataMat
# 计算欧式距离
def disEclud(vecA,vecB):
return sqrt(sum(power(vecA - vecB,2)))
# 随机构造k个簇质心
def randCent(dataSet,k):
n = shape(dataSet)[1] # 列数
centroids = mat(zeros((k,n))) # 构建k行n列为0的矩阵,存放k个质心
# 构建簇质心
for j in range(n):
minJ = min(dataSet[:,j]) #一列中最小数据
rangeJ = float(max(dataSet[:,j])-minJ) # 一列中最大值-最小值的差值
centroids[:,j] = minJ + rangeJ * random.rand(k,1)
return centroids
# dataMat = mat(loadDataSet('testSet.txt'))
# print(randCent(dataMat,2))
# K均值聚类
def KMeans(dataSet,k,distMeans=disEclud,createCent=randCent):
m = shape(dataMat)[0] # 数据行数
clusterAssment = mat(zeros((m,2))) # 创建m行2列0矩阵,一列记录簇索引值,一列存储误差
centroids = createCent(dataSet, k) #随机构建k质心
clusterChanged = True # 簇分配改变标志,改变则继续迭代,否则退出while循环
while clusterChanged:
clusterChanged = False
for i in range(m): # 遍历数据所有行
minDist = inf; minIndex = -1 # 初始化最小值
for j in range(k): # 遍历所有质心
distJI = distMeans(centroids[j,:],dataSet[i,:]) # 计算样本与质心
if distJI < minDist:
minDist = distJI; minIndex = j # 找出样本距离最近的质心的索引
if clusterAssment[i,0] != minIndex: # 更新当前所属质心
clusterChanged = True # 如果距离所属质心的距离不是最小,设置clusterChanged = True
clusterAssment[i,:] = minIndex,minDist**2
print(centroids)
for cent in range(k):
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]] # 过滤来获得所给定簇的所有点
centroids[cent,:] = mean(ptsInClust,axis=0) #沿矩阵列方向进行均值计算
return centroids,clusterAssment # 返回所有类质心和点分配结果
dataMat = mat(loadDataSet('testSet.txt'))
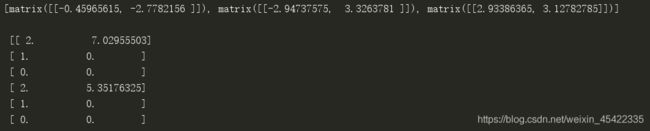
centroids,clusterAssment = KMeans(dataMat,4)
print(centroids,'\n\n',clusterAssment)
返回K个质心坐标和簇分配结果,可以看到结果给出4个质心,经过4次迭代之后K-均值算法收敛。

二分K均值聚类算法
在^K均值聚类中簇的数目是一个用户预先定义的参数,那么用户如何才能知道乂的选择是否正确?如何才能知道生成的簇比较好呢?在包含簇分配结果的矩阵中保存着每个点的误差,即该点到簇质心的距离平方值。。K-均值算法收敛但聚类效果较差的原因是,K-均值算法收敛到了局部最小值,而非全局最小值。一种用于度量聚类效果的指标是SSE(SumofSquaredError,误差平方和)。SSE值越小表示数据点越接近于它们的质心,聚类效果也越好。因为对误差取了平方,因此更加重视那些远离中心的点。一种肯定可以降低SSE值的方法是增加簇的个数,但这违背了聚类的目标。聚类的目标是在保持族数目不变的情况下提高簇的质量。
可以对生成的簇进行后处理,一种方法是将具有最大SSE值的簇划分成两个簇。具体实现时可以将最大簇包含的点过滤出来并在这些点上运行尺-均值算法,其中的K为2。为克服K-均值算法收敛于局部最小值的问题,有人提出了另一个称为二分K均值(bisecting K-means)的算法K 该算法首先将所有点作为一个簇,然后将该簇一分为二。之后选择其中一个簇继续进行划分,选择哪一个簇进行划分取决于对"其划分是否可以最大程度降低SSE的值。上述基于SSE的划分过程不断重复,直到得到用户指定的簇数目为止。
二分K-均值算法的代码如下:
from numpy import *
def loadDataSet(fileName):
dataMat =[]
fr = open(fileName).readlines()
for line in fr:
curLine = line.strip().split('\t')
# map 两个参数,第一个为函数,第二个为序列
# 即rangeJ = float(max(dataSet[:, j]) - minJ)相减的是两个map类型的数据,经过查找,发现fltLine = map(float, curLine)
# 在python2中返回的是一个list类型数据,而在python3中该语句返回的是一个map类型的数据。
# fltLine = map(float,curLine)
fltLine = list(map(float, curLine))
dataMat.append(fltLine)
return dataMat
# 计算欧式距离
def disEclud(vecA,vecB):
return sqrt(sum(power(vecA - vecB,2)))
# 随机构造k个簇质心
def randCent(dataSet,k):
n = shape(dataSet)[1] # 列数
centroids = mat(zeros((k,n))) # 构建k行n列为0的矩阵,存放k个质心
# 构建簇质心
for j in range(n):
minJ = min(dataSet[:,j]) #一列中最小数据
rangeJ = float(max(dataSet[:,j])-minJ) # 一列中最大值-最小值的差值
centroids[:,j] = minJ + rangeJ * random.rand(k,1)
return centroids
# dataMat = mat(loadDataSet('testSet.txt'))
# print(randCent(dataMat,2))
# K均值聚类
def KMeans(dataSet,k,distMeans=disEclud,createCent=randCent):
m = shape(dataSet)[0] # 数据行数
clusterAssment = mat(zeros((m,2))) # 创建m行2列0矩阵,一列记录簇索引值,一列存储误差
centroids = createCent(dataSet, k) #随机构建k质心
clusterChanged = True # 簇分配改变标志,改变则继续迭代,否则退出while循环
while clusterChanged:
clusterChanged = False
for i in range(m): # 遍历数据所有行
minDist = inf; minIndex = -1 # 初始化最小值
for j in range(k): # 遍历所有质心
distJI = distMeans(centroids[j,:],dataSet[i,:]) # 计算样本与质心
if distJI < minDist:
minDist = distJI; minIndex = j # 找出样本距离最近的质心的索引
if clusterAssment[i,0] != minIndex: # 更新当前所属质心
clusterChanged = True # 如果距离所属质心的距离不是最小,设置clusterChanged = True
clusterAssment[i,:] = minIndex,minDist**2
print(centroids)
for cent in range(k):
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]] # 过滤来获得所给定簇的所有点
centroids[cent,:] = mean(ptsInClust,axis=0) #沿矩阵列方向进行均值计算
return centroids,clusterAssment # 返回所有类质心和点分配结果
# 二分K-均值聚类算法
def biKmeans(dataSet,K,distMeans=disEclud):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2)))
centroid0 = mean(dataSet,axis=0).tolist()[0]
centList = [centroid0]
for j in range(m):
clusterAssment[j,1] = distMeans(mat(centroid0),dataSet[j,:])**2
while (len(centList) < K):
lowestSSE = inf # 初始化最小SSE为正无穷大
for i in range(len(centList)):
pstInCurrCluster = dataSet[nonzero(clusterAssment[:, 0].A == i)[0], :] # 取出每个簇的数据集
centroidMat,splitClustAss = KMeans(pstInCurrCluster,2,distMeans) # 将一个簇划分2个簇,返回各个簇质心和簇分配结果
sseSplit = sum(splitClustAss[:, 1]) # 簇的误差值
sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:,0].A !=i)[0],:]) # 剩余数据集的误差
#print("sseSplit,and notSplit:",sseSplit,sseNotSplit)
if (sseSplit + sseNotSplit) < lowestSSE:
bestCentToSplit = i # 要分割的簇质心索引
bestNewCents = centroidMat # 确定新的簇质心
bestClustAss = splitClustAss.copy()
lowestSSE = sseSplit + sseNotSplit # 划分误差和剩余数据集的误差之和
# 更新簇的分配结果
# 将需要分割的聚类中心下的点进行1划分
# 新增的聚类中心编号为len(centList)
bestClustAss[nonzero(bestClustAss[:,0].A==1)[0],0] = len(centList)
bestClustAss[nonzero(bestClustAss[:,0].A==0)[0],0] = bestCentToSplit
#print("the bestCentTosplit is:" ,bestNewCents)
#print("the len of bestClustAss is:",len(bestClustAss))
# 更新被分割的聚类中心的坐标
centList[bestCentToSplit] = bestNewCents[0,:]
# 增加聚类中心
centList.append(bestNewCents[1,:])
clusterAssment[nonzero(clusterAssment[:,0].A == bestCentToSplit)[0],:] = bestClustAss
return centList,clusterAssment
dataMat3 = mat(loadDataSet('testSet2.txt'))
centList,clusterAssment = biKmeans(dataMat3,3)
print(centList,'\n\n',clusterAssment)
对地理坐标进行聚类
现在我们有一个包含格式化地理坐标的列表,接下来可以对这些俱乐部进行聚类。这个例子中要聚类的俱乐部给出的信息为经度和维度,但这些信息对于距离计算还不够。在北极附近每走几米的经度变化可能达到数10度;而在赤道附近走相同的距离,带来的经度变化可能只是零点几。可以使用球面余弦定理来计算两个经纬度之间的距离<
from numpy import *
def loadDataSet(fileName):
dataMat =[]
fr = open(fileName).readlines()
for line in fr:
curLine = line.strip().split('\t')
# map 两个参数,第一个为函数,第二个为序列
# 即rangeJ = float(max(dataSet[:, j]) - minJ)相减的是两个map类型的数据,经过查找,发现fltLine = map(float, curLine)
# 在python2中返回的是一个list类型数据,而在python3中该语句返回的是一个map类型的数据。
# fltLine = map(float,curLine)
fltLine = list(map(float, curLine))
dataMat.append(fltLine)
return dataMat
# 计算欧式距离
def disEclud(vecA,vecB):
return sqrt(sum(power(vecA - vecB,2)))
# 随机构造k个簇质心
def randCent(dataSet,k):
n = shape(dataSet)[1] # 列数
centroids = mat(zeros((k,n))) # 构建k行n列为0的矩阵,存放k个质心
# 构建簇质心
for j in range(n):
minJ = min(dataSet[:,j]) #一列中最小数据
rangeJ = float(max(dataSet[:,j])-minJ) # 一列中最大值-最小值的差值
centroids[:,j] = minJ + rangeJ * random.rand(k,1)
return centroids
# dataMat = mat(loadDataSet('testSet.txt'))
# print(randCent(dataMat,2))
# K均值聚类
def KMeans(dataSet,k,distMeans=disEclud,createCent=randCent):
m = shape(dataSet)[0] # 数据行数
clusterAssment = mat(zeros((m,2))) # 创建m行2列0矩阵,一列记录簇索引值,一列存储误差
centroids = createCent(dataSet, k) #随机构建k质心
clusterChanged = True # 簇分配改变标志,改变则继续迭代,否则退出while循环
while clusterChanged:
clusterChanged = False
for i in range(m): # 遍历数据所有行
minDist = inf; minIndex = -1 # 初始化最小值
for j in range(k): # 遍历所有质心
distJI = distMeans(centroids[j,:],dataSet[i,:]) # 计算样本与质心
if distJI < minDist:
minDist = distJI; minIndex = j # 找出样本距离最近的质心的索引
if clusterAssment[i,0] != minIndex: # 更新当前所属质心
clusterChanged = True # 如果距离所属质心的距离不是最小,设置clusterChanged = True
clusterAssment[i,:] = minIndex,minDist**2
# print(centroids)
for cent in range(k):
ptsInClust = dataSet[nonzero(clusterAssment[:,0].A==cent)[0]] # 过滤来获得所给定簇的所有点
centroids[cent,:] = mean(ptsInClust,axis=0) #沿矩阵列方向进行均值计算
return centroids,clusterAssment # 返回所有类质心和点分配结果
# 二分K-均值聚类算法
def biKmeans(dataSet,K,distMeans=disEclud):
m = shape(dataSet)[0]
clusterAssment = mat(zeros((m,2)))
centroid0 = mean(dataSet,axis=0).tolist()[0]
centList = [centroid0]
for j in range(m):
clusterAssment[j,1] = distMeans(mat(centroid0),dataSet[j,:])**2
while (len(centList) < K):
lowestSSE = inf # 初始化最小SSE为正无穷大
for i in range(len(centList)):
pstInCurrCluster = dataSet[nonzero(clusterAssment[:, 0].A == i)[0], :] # 取出每个簇的数据集
centroidMat,splitClustAss = KMeans(pstInCurrCluster,2,distMeans) # 将一个簇划分2个簇,返回各个簇质心和簇分配结果
sseSplit = sum(splitClustAss[:, 1]) # 簇的误差值
sseNotSplit = sum(clusterAssment[nonzero(clusterAssment[:,0].A !=i)[0],:]) # 剩余数据集的误差
#print("sseSplit,and notSplit:",sseSplit,sseNotSplit)
if (sseSplit + sseNotSplit) < lowestSSE:
bestCentToSplit = i # 要分割的簇质心索引
bestNewCents = centroidMat # 确定新的簇质心
bestClustAss = splitClustAss.copy()
lowestSSE = sseSplit + sseNotSplit # 划分误差和剩余数据集的误差之和
# 更新簇的分配结果
# 将需要分割的聚类中心下的点进行1划分
# 新增的聚类中心编号为len(centList)
bestClustAss[nonzero(bestClustAss[:,0].A==1)[0],0] = len(centList)
bestClustAss[nonzero(bestClustAss[:,0].A==0)[0],0] = bestCentToSplit
#print("the bestCentTosplit is:" ,bestNewCents)
#print("the len of bestClustAss is:",len(bestClustAss))
# 更新被分割的聚类中心的坐标
centList[bestCentToSplit] = bestNewCents[0,:].tolist()[0]
# 增加聚类中心
centList.append(bestNewCents[1,:].tolist()[0] )
clusterAssment[nonzero(clusterAssment[:,0].A == bestCentToSplit)[0],:] = bestClustAss
return mat(centList),clusterAssment
# dataMat3 = mat(loadDataSet('testSet2.txt'))
# centList,clusterAssment = biKmeans(dataMat3,3)
# print(centList,'\n\n',clusterAssment)
"""
函数distSLC()返回地球表面两点间的距离,单位是英里。给定两个点的经纬度,可以使用
球面余弦定理来计算两点的距离。这里的纬度和经度用角度作为单位,但是sin()以及cos()以
弧度为输入。可以将角度除以180然后再乘以圆周率pi转换为弧度。导入NumPy的时候就会导
入pi
"""
def distSLC(vecA, vecB):#Spherical Law of Cosines
a = sin(vecA[0,1]*pi/180) * sin(vecB[0,1]*pi/180)
b = cos(vecA[0,1]*pi/180) * cos(vecB[0,1]*pi/180) * \
cos(pi * (vecB[0,0]-vecA[0,0]) /180)
return arccos(a + b)*6371.0 #pi is imported with numpy
import matplotlib
import matplotlib.pyplot as plt
def clusterClubs(numClust=5):#参数:希望得到的簇数目
datList = []
for line in open('places.txt').readlines():#获取地图数据
lineArr = line.split('\t')
datList.append([float(lineArr[4]), float(lineArr[3])])#逐个获取第四列和第五列的经纬度信息
datMat = mat(datList)
myCentroids, clustAssing = biKmeans(datMat, numClust, distMeans=distSLC)
#draw
fig = plt.figure()
rect=[0.1,0.1,0.8,0.8]#创建矩形
#创建不同标记图案
scatterMarkers=['s', 'o', '^', '8', 'p', \
'd', 'v', 'h', '>', '<']
axprops = dict(xticks=[], yticks=[])
ax0=fig.add_axes(rect, label='ax0', **axprops)
imgP = plt.imread('Portland.png')#导入地图
ax0.imshow(imgP)
ax1=fig.add_axes(rect, label='ax1', frameon=False)
for i in range(numClust):
ptsInCurrCluster = datMat[nonzero(clustAssing[:,0].A==i)[0],:]
markerStyle = scatterMarkers[i % len(scatterMarkers)]
ax1.scatter(ptsInCurrCluster[:,0].flatten().A[0], ptsInCurrCluster[:,1].flatten().A[0], marker=markerStyle, s=90)
ax1.scatter(myCentroids[:,0].flatten().A[0], myCentroids[:,1].flatten().A[0], marker='+', s=300)
plt.show()
clusterClubs()