论文解读:GBPNet:蛋白质结构的通用几何表示学习
GBPNet: Universal Geometric Representation Learning on Protein Structures
DOI:https://doi.org/10.1145/3534678.3539441
Github:GBPNet/gbpnet/datamodules at main · sarpaykent/GBPNet · GitHub
摘要:
蛋白质3D结构的表示学习对于例如计算蛋白质设计或蛋白质工程的应用是具有挑战性的并且是必不可少的。近年来,几何深度学习在非欧几里得领域取得了巨大成功。尽管蛋白质可以自然地表示为图形,但主要由于在建模复杂的表示和捕捉3D结构建模中的固有相关性方面存在重大挑战,因此它仍然没有得到充分的探索。几个挑战包括:1)在学习过程中提取和保存多级旋转和翻译等变信息是一项挑战。2) 难以开发适当的工具来有效地利用输入的空间表示来捕捉空间维度上的复杂几何图形。3) 难以结合各种几何特征并保留固有的结构关系。在这项工作中,我们引入了几何瓶颈感知器,以及一个一般的SO(3)-等变消息p
数据集
蛋白质结构分类数据集:CPD中使用的数据集是基于蛋白质结构分级分类(CATH)构建的[29]。具有相同CATH拓扑分类的测试集中的所有链都从训练和验证拆分中删除,以避免不平衡的数据集偏差。在实验中,我们在之前的工作[10]之后使用了80%、10%、10%的分割来测试我们的模型。过滤后,训练集中有18024个链,验证集中有609个链,测试集中有1120个链。
蛋白质结构排名数据集:PSR数据集是提交给CASP[18]竞赛的预测3D模型的集合。
预测的模型通过全局距离检验(GDT_TS)相对于实验观察到的天然蛋白质结构进行评估。
该数据集包含在九场CASP竞赛中提交的预测和目标。我们遵循与先前工作相同的数据集分割[30]。
配体结合亲和力预测数据集:LBA数据集是属
Benchmarks The proposed method is compared with the state-of-art methods for all three tasks.
• To validate the significance of the proposed GBP for PSR task, the proposed model is compared with 3DCNN [11], ProQ3D [31], VoroMQA [24], RWplus [36], SBROD [14], Ornate [26], DimeNet [17], GraphQA [3], and GVP [11].
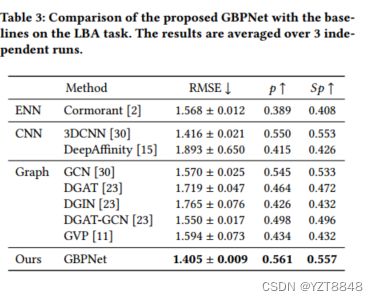
• For the LBA task, we compared the proposed model with Cormorant [2], 3DCNN [11], DeepAffinity [15], DGIN [23], DGAT [23] , DGAT-GCN [23] and GVP [11].
• To validate the superiority of the proposed GBP for the classification task CPD, the proposed method is compared with STran [10], SGNN [10], and GVP [12].
实验
在本节中,我们评估了我们提出的GBPNet在蛋白质3D结构的几何表示学习中的三个核心任务:CPD、PSR和LBA。除了在所需任务输出和真实世界用例中的多样性外,这三个任务还跨越了我们提出的几何消息传递框架的不同用例:CPD是一个分类任务,PSR和LBA是回归任务。
实验是在具有24GB内存的4x Nvidia 3090 GPU上进行的。在补充材料中进一步讨论了设计选择和超参数
模型结构
学习复杂蛋白质结构的几何性质是一个具有挑战性的问题。我们的研究旨在学习表象 嵌入几何结构信息的图,并满足分析和执行下游任务的几个方面,包括强判别力和等变性质。要实现这一目标,需要解决几个挑战,包括:1)难以利用节点/边和图谱中不同类型和级别的几何信息。2) 难以在空间维度上捕捉复杂的几何图形。3) 为广泛的几何表示学习任务设计通用框架的困难。
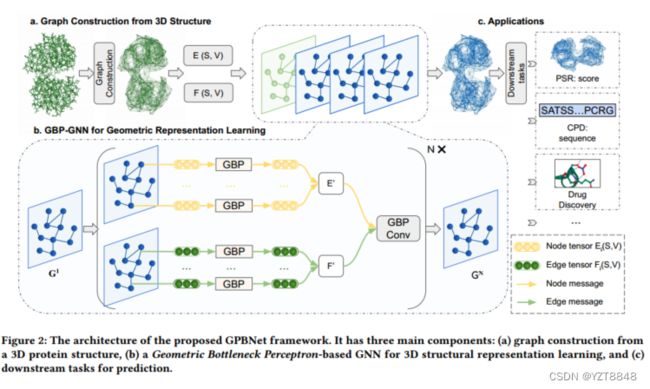
为了纠正上述挑战,我们提出了一种新的基于几何瓶颈感知器的图神经网络(GBPNet)来对蛋白质的3D结构进行建模。如图2所示,整个体系结构由三个主要组件组成。我们首先构造t中的几何图
结果
7.1.1计算蛋白质设计。表1显示了GBPNet与CPD任务基线的比较。我们提出的方法在困惑和恢复分数方面都优于基线方法。此外,所提出的模型将Short和Single子集的困惑增加了15%以上。所有结构的回收率提高了8%以上。平均而言,我们的模型将性能提高了8%。
7.1.2蛋白质结构分级。表2显示了GBPNet在PSR任务上的性能比较。我们将结果分为两部分,即局部和全局。局部表示每个目标计算评估度量,并对结果进行平均以获得最终值。全局表示应用于测试集中所有样本的评估度量。与所有基线方法相比,我们的模型在每个指标上都取得了最佳性能。平均而言,与最佳基线方法相比,局部指标的改进超过5%。
消融实验
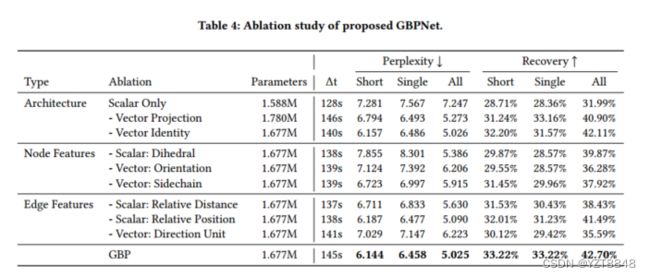
在本节中,我们对GBP的两种变化进行了消融实验,以研究影响模型性能的因素。表4显示了我们的评估结果。Δt表示以秒为单位完成一个训练时期的平均时间。
7.2.1 GBP表达路径的影响。我们比较了GBP表达式路径对 和 如表4的第一行所示。仅标量变体删除( ) 节点和边的路径。模型性能在困惑和恢复分数上都严重下降。因此,与标量相互作用的向量表达式路径对于几何图表示学习至关重要。矢量投影变化删除上的瓶颈缩小和放大操作 路径(如果可能)。结果表明,具有瓶颈的载体表达路径成功地帮助模型学习了蛋白质的几何结构。Vector Identity变量删除了用于计算的逐元素乘法 ′ 英镑板块。尽管这种变化在困惑得分方面与我们提出的模型表现相似,但恢复得分平均下降了3%。
结论
本文的重点是学习蛋白质结构的几何表示。我们提出了GBPNet,一种新的SO(3)-等变信息传递神经网络,用于学习蛋白质结构的几何表示。此外,我们还提出了一个名为“几何瓶颈感知器”的插件模块,以集成几何特征并捕捉三维结构中复杂的几何关系。我们提出的GBP是一个功能强大且通用的模块,用于学习和表示几何特征。我们展示了GBPNet在三个任务上的性能,实验结果验证了所提出架构的有效性。