python数据分析-聚类分析
大家好,聚类分析是一类将数据所对应的研究对象进行分类的统计方法。这一类方法的共同特点是,事先不知道类别的个数与结构;进行分析的数据是表明对象之间的相似性或相异性的数据,将这些数据看成对对象“距离”远近的一种度量,将距离近的对象归入一类,不同类对象之间的距离较远。
聚类分析根据对象的不同分为Q型聚类分析和R型聚类分析,其中,Q型聚类是指对样本的聚类,R型聚类是指对变量的聚类,今天我们主要介绍Q型聚类。
一、距离和相似系数
1.1、距离
在聚类过程中,相距较近的样本点倾向于归为一类,相距较远的样本点应归属于不同的类。最常用的是Minkowski距离。
当各变量的单位不同或变异性相差很大时,不应直接采用Minkowski距离,而应先对各变量的数据做标准化处理,然后用标准化后的数据计算距离。
使用SciPy库spatial模块下的distance子模块可以计算距离,使用该子模块下的pdist函数可以计算n维空间中观测值之间的距离,其语法格式如下:
scipy.spatial.distance.pdist(X,metric='euclidean',p=None,w=None,V=None,VI=None)
# X:接受ndarray,表示需要计算距离的数据。无默认值
# metric:接受特定str或function,表示进行计算距离的方法。
取值为euclidean时,表示欧式距离;
取值为minkowski时,表示Minkowski距离;
取值为cityblock时,表示绝对值距离;
取值为cosine时,表示夹角余弦。默认为euclidean实例:设有5个样本(x1,x2,…,x5),每个样本只测量了一个指标,分别是1、2、5、9、13,请采用绝对值距离求这5个样本的距离矩阵。
import numpy as np
from scipy import spatial
data = np.array([[1,2,5,9,13]])
dist1 = spatial.distance.pdist(data.T,'cityblock')
print('距离矩阵为:\n', dist1)输出结果:
距离矩阵为:
[ 1. 4. 8. 12. 3. 7. 11. 4. 8. 4.]1.2、相似系数
在对变量进行聚类时,常常采用相似系数进行变量之间相似性的度量,相似系数(或其绝对值)越大,认为变量之间的相似性程度就越高,反之越低。聚类时,相似系数高的变量聚类倾向于归为一类,相似系数低的变量归属不同的类。
在Python中采用夹角余弦度量变量之间的相似度。
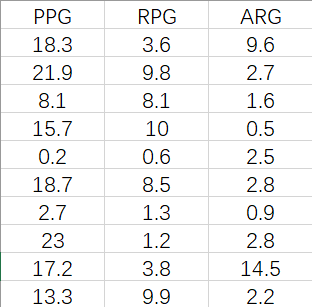
实例:某篮球联赛共计257名篮球运动员,下表展示了他们的赛季场均得分(PPG)、场均篮板(RPG)和场均助攻(ARG)的前10条记录,试采用夹角余弦度量每个球员之间的相似度。
import scipy.cluster.hierarchy as sch
ball = np.loadtxt(r'D:/data/basketball.csv ', delimiter= ',')
dist2 = sch.distance.pdist(ball,'cosine')
print('每个变量之间的相似度为:\n', dist2)结果输出:
每个变量之间的相似度为:
[0.08784095 0.20554827 0.15998659 ... 0.0228912 0.27088262 0.22936317]二、系统聚类法
系统聚类法也称为层次聚类法,其基本思想为:开始时将n个样本各自为一类,并规定样本之间的距离和类与类之间的距离,然后将距离最近的两类合并成一个新类,再计算新类与其他类的距离;重复进行两个最近类的合并,每次减少一类,直至所有样本合并成一类。
2.1、最短距离法
定义类与类之间的距离为两类最近样本间的距离,这种系统聚类法称为最短距离法或单连接法(Single Linkage Method)。
使用SciPy库cluster模块的hierarchy子模块可以实现系统聚类,使用该子模块下的linkage函数可以实现最短距离法、最长距离法、类平均法和重心法等,其语法格式如下:
scipy.cluster.hierarchy.linkage(y,method='single',metric='euclidean')
# y:接受ndarray,表示需要聚类的数据。无默认值
# method:接受str,表示计算聚类的方法。
取值为single时,表示最短距离法;
取值为complete时,表示最长距离法;
取值为average时,表示类平均法;
取值为ward时,表示离差平方和法(ward法)等。默认为single
# metric:接受特定str或function,表示在y是观测向量集合的情况下使用的计算距离的方法。
取值为euclidean时,表示欧式距离;
取值为minkowski时,表示Minkowski距离;
取值为cityblock时,表示绝对值距离;
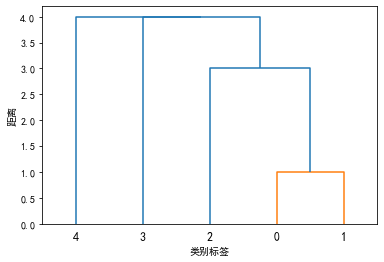
取值为cosine时,表示夹角余弦。默认为euclidean实例:对1.1的例子的5个样本(x1,x2,…,x5)采用最短距离法进行聚类。
import matplotlib.pylab as plt
import numpy as np
import scipy.cluster.hierarchy as sch
plt.rcParams['font.sans-serif']=['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False # 用来正常显示负号
data = np.array([[1,2,5,9,13]])
#Min = sch.linkage(dist1, method='single') # 方法一
Min = sch.linkage(data.T, method='single', metric='cityblock') # 方法二
P = sch.dendrogram(Min)
plt.xlabel('类别标签')
plt.ylabel('距离')
plt.savefig('D:/data/最短距离法聚类结果.png')
plt.show()输出结果:
2.2、最长距离法
定义类与类之间的距离为两类最远样本间的距离,这种系统聚类法称为最长距离法或完全连接法(Complete Linkage Method),最短距离法和最长距离法的并类步骤的区别在于类间距离的递推公式不同。
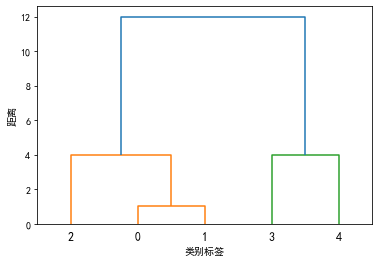
实例:对1.1的例子的5个样本(x1,x2,…,x5)采用最长距离法进行聚类。
import matplotlib.pylab as plt
import numpy as np
import scipy.cluster.hierarchy as sch
plt.rcParams['font.sans-serif']=['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False # 用来正常显示负号
data = np.array([[1,2,5,9,13]])
#Max1 = sch.linkage(dist1, method='complete') # 方法一
Max1 = sch.linkage(data.T, method='complete', metric='cityblock') # 方法二
P = sch.dendrogram(Max1)
plt.xlabel('类别标签')
plt.ylabel('距离')
plt.savefig('D:/data/最长距离法聚类结果1.png')
plt.show()输出结果:
2.3、类平均法
平均法或平均连接法(Average Linkage Method)有两种定义,一种定义方法是把类与类之间的距离定义为所有样本对之间的平均距离。
另一种定义方法是定义类与类之间的平方距离为样本对之间的平方距离的平均值。
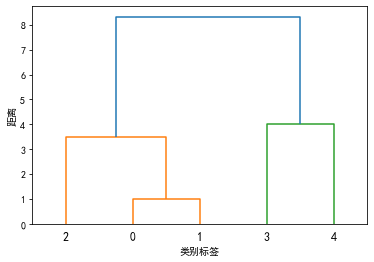
实例:在Python中,对1.1的例子的5个样本(x1,x2,…,x5)的数据采用类平均法对球员进行聚类。
import matplotlib.pylab as plt
import numpy as np
import scipy.cluster.hierarchy as sch
plt.rcParams['font.sans-serif']=['SimHei'] # 用来正常显示中文标签
plt.rcParams['axes.unicode_minus']=False # 用来正常显示负号
data = np.array([[1,2,5,9,13]])
#Ave1 = sch.linkage(dist1, method='average') # 方法一
Ave1 = sch.linkage(data.T, method='average', metric='cityblock') # 方法二
P = sch.dendrogram(Ave1)
plt.xlabel('类别标签')
plt.ylabel('距离')
plt.savefig('D:/data/类平均法聚类结果1.png')
plt.show()输出结果:
三、 K-Means聚类法
基本步骤:
第一步:从n个样本数据中随机选取k个对象作为初始的聚类中心。
第二步:分别计算每个样本到各个聚类质心的距离,将样本分配到距离最近的那个聚类中心类别中。
第三步:所有样本分配完成后,重新计算k个聚类的中心。
第四步:与前一次计算得到的k个聚类中心比较,如果聚类中心发生变化,转第二步,否则转第五步。
第五步:当中心不再发生变化时,停止并输出聚类结果。
使用SciPy库cluster模块的vq子模块可以实现K-Means聚类。使用该子模块下的kmeans函数或kmeans2函数可以返回最终的聚类中心,语法格式如下:
scipy.cluster.vq.kmeans(obs,k_or_guess,iter=20,thresh=1e-05,check_finite=Ture)
# obs:接收ndarray,表示需要进行的聚类的数据。无默认值
# k_or_guess:接收 int或ndarray,表示聚类的个数。无默认值
# iter:接收int,表示迭代的次数。默认为20
scipy.cluster.vq.kmeans2(data,k,iter=10,thresh=1e-05,minit='random',missing='warn',check_finite=Ture)
# data:接收ndarray,表示需要进行的聚类的数据。无默认值
# k:接收 int或ndarray,表示聚类的个数。无默认值
# iter:接收int,表示迭代的次数。默认为10在使用kmeans函数或kmeans2函数聚类前,都需用whiten函数对数据的每个变量的观测值进行单位化,语法格式如下:
scipy.cluster.vq.whiten(obs,check_finite=Ture)
# obs:接收ndarray,表示需要进行单位化的数据。无默认值实例:在Python中,对例1.2中的球员场均得分、篮板助攻的数据采用K-Means聚类法对球员进行聚类,指定聚类的个数k=2
import numpy as np
import scipy.cluster.vq as vq
import matplotlib.pylab as plt
ball = np.loadtxt(r'D:/data/basketball.csv ', delimiter= ',')
data1 = vq.whiten(ball) # 对数据进行单位化
# 方法一
kmeans_cent1 = vq.kmeans2(data1, 2)
print('聚类中心为:\n', kmeans_cent1[0])
# 方法二
kmeans_cent2 = vq.kmeans(data1, 2)
print('聚类中心为:\n', kmeans_cent2[0])
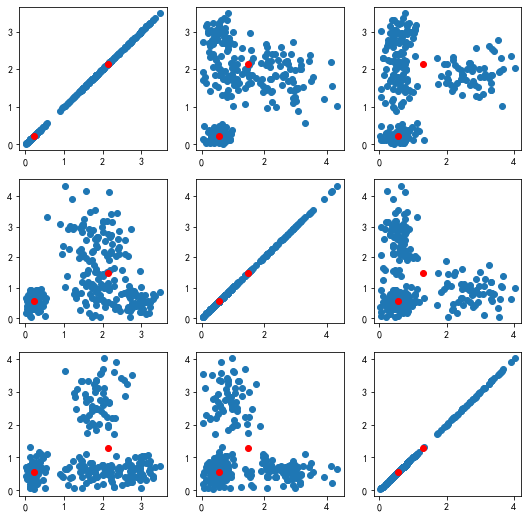
# 画出单位化后和聚类中心的散点图
p = plt.figure(figsize=(9,9))
for i in range(3):
for j in range(3):
ax = p.add_subplot(3,3,i*3+1+j)
plt.scatter(data1[:, j], data1[:, i])
plt.scatter(kmeans_cent2[0][:, j], kmeans_cent2[0][:, i], c='r')
plt.savefig('D:/data/K-Means聚类法聚类结果.png')
plt.show()输出结果:
聚类中心为:
[[0.23038297 0.55161329 0.5646255 ]
[2.14466124 1.49294345 1.29623859]]