Opencv医学图片分割-以血管、胼胝体MR等分割为例
用到环境
1、pycharm community edition 2022.3.2
2、Python 3.10
后面应该会传代码到资源,比较需要的可以私信我。
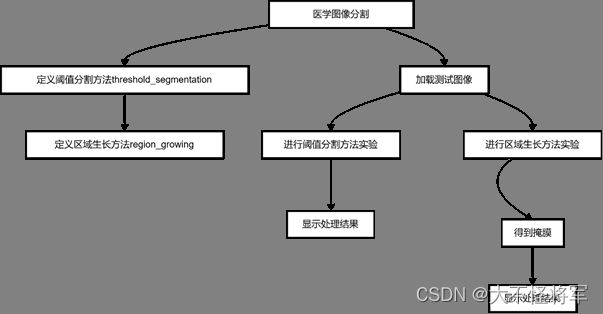
总体设计
结果展示
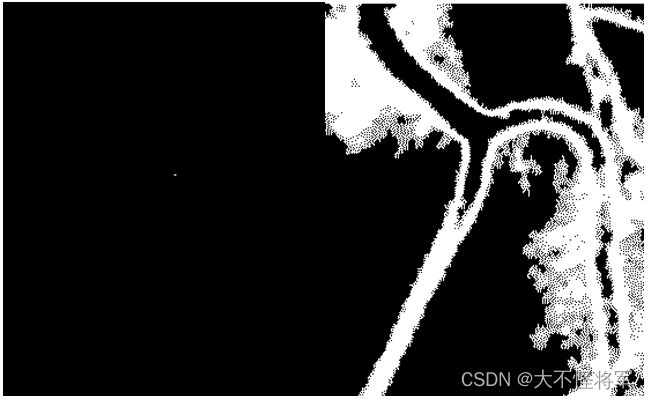
血管
分析:都可以有效实现分割,但是左边的阈值分割去噪能力更强,右边的区域生长法分割的更全面。
分析:左边为阈值选取不合理,右边为阈值选取合理,可以看出阈值选取得当对区域生长法很重要。
分析:左边为区域生长法种子点坐标选取不得当,右边为区域生长法种子点坐标选取得当,可以看出区域生长法种子点坐标选取对结果有致命的影响。
胼胝体MR图像
代码:
import cv2
import numpy as np
# 阈值分割方法
def threshold_segmentation(image, threshold):
# 将图像转为灰度图
gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)
# 进行阈值分割
ret, binary = cv2.threshold(gray, threshold, 255, cv2.THRESH_BINARY)
return binary
# 区域生长方法
def region_growing(image, seed_point, threshold):
# 将图像转为灰度图
gray = cv2.cvtColor(image, cv2.COLOR_BGR2GRAY)
# 创建一个和图像大小相同的掩膜,用于记录已经生长的区域
mask = np.zeros_like(gray)
# 将种子点加入到生长队列中
queue = []
queue.append(seed_point)
# 进行区域生长
while len(queue) > 0:
# 取出队首像素点
current_point = queue.pop(0)
# 如果当前像素点未被访问过,且符合生长条件,则加入到生长区域中
if mask[current_point[1], current_point[0]] == 0 and abs(gray[current_point[1], current_point[0]] - gray[seed_point[1], seed_point[0]]) < threshold:
mask[current_point[1], current_point[0]] = 255
# 将当前像素点的相邻像素加入到生长队列中
if current_point[1] > 0:
queue.append([current_point[0], current_point[1] - 1])
if current_point[1] < image.shape[0] - 1:
queue.append([current_point[0], current_point[1] + 1])
if current_point[0] > 0:
queue.append([current_point[0] - 1, current_point[1]])
if current_point[0] < image.shape[1] - 1:
queue.append([current_point[0] + 1, current_point[1]])
return mask
30
# 加载测试图像
image = cv2.imread('1.png')
# 阈值分割方法实验
threshold = 80
binary = threshold_segmentation(image, threshold)
cv2.imshow('Threshold Segmentation', binary)
# 区域生长方法实验
seed_point = [100, 100] # 种子点坐标
threshold =30 # 生长阈值
mask = region_growing(image, seed_point, threshold)
cv2.imshow('Region Growing', mask)
cv2.waitKey(0)
cv2.destroyAllWindows()
编写不易,求个点赞!!!!!!!
“你是谁?”
“一个看帖子的人。”
“看帖子不点赞啊?”
“你点赞吗?”
“当然点了。”
“我也会点。”
“谁会把经验写在帖子里。”
“写在帖子里的那能叫经验贴?”
“上流!”
cheer!!!
![]()