深入理解Local Outlier Factor(LOF)局部异常因子算法
目录
- 一、概述
- 二、LOF算法
-
- 1. 直观理解
- 2. 核心思想
- 3.深入理解LOF
-
- 3.1. k邻近距离
- 3.2. k距离邻域
- 3.3. 可达距离
- 3.4. 局部可达密度
- 3.5. 局部异常因子
- 4.LOF算法流程
- 5.LOF算法优缺点
- 三、Python代码实现
- 四、参考文档
一、概述
首先,写这篇文章的初衷是为了记录自身对LOF的理解,另一个原因是个人在学习该算法的时候,也查阅过不少的文章或者视频,有一些知识点(如可达距离、局部可达密度等概念)可能并没有清晰的表达出来,因此该文章本着个人对该算法的理解记录学习该算法的过程,如有错误,请直接私信tinstone,希望对刚接触该算法的同学有所帮助,让知识传播下去。
Local Outlier Factor(LOF)是基于密度的经典算法,文章发表于 SIGMOD 2000。在LOF之前的异常检测算法大多是基于统计的方法(3α原则、箱型图等),或者是借用一些聚类算法的异常识别(如DBSCAN),而这些方法都有一些不完美的地方:
- 基于统计的方法:通常需要假设数据服从某些特定的概率分布
- 聚类方法:通常只能给出0/1的判断(即是否为异常点),不能量化每个数据点异常程度,而且DBSCAN算法是一个全局性指标算法,对于所有数据使用相同的模型参数。
相比较而言,基于密度的LOF算法要更简单、直观。它不需要对数据的分布做太多的假设,还能量化的给出每个数据点的异常程度。
二、LOF算法
1. 直观理解
首先,基于密度的离群点检测方法有一个假设:非离群点对象周围的密度与其相邻点的周围的密度类似,而离群点对象周围的密度显著不同于其邻域周围点的密度。
通过下面的图片,可以直观的感受下:

聚集簇C1的点的分布间距比较大,即密度较小,但该簇内的点的密度都类似,而聚集簇C2 的点的分布间距更紧密,即密度更大,但该簇内的点的密度也都是类似的,再来观察O1、O2两个数据点,其周边的点数量较少或者密度较低,因此这两个点判定为异常点,因为基于假设得到:这两个点周围的密度显著不同于周围点的密度。
2. 核心思想
LOF,又称局部异常因子算法,从这个算法名称中我们先剧透一些信息:
- 局部:如何定义局部??联想KNN中的k近邻
- 异常因子:如何定义异常?如何计算异常因子?LOF使用密度的概念定义了点与周围点的相对密度来确定点是否异常
有了上面的理解,这里一句话总结LOF的核心思想:通过计算一个score来反映一个样本的异常程度(该score是一个相对值,注意区别使用KNN进行异常检测的平均距离),如何理解score:一个样本点周围的样本的所处位置的平均密度比上该样本点所在位置的密度,比值越大于1,则该点所在位置的密度越小于其周围样本所在位置的密度, 这个点就越有可能是异常点。
3.深入理解LOF
在了解LOF之前,必须知道几个基本的概念,因为LOF是基于这几个概念来定义“局部”、“密度”、“异常因子”等相关概念的。
3.1. k邻近距离
在距离数据点P最近的几个点中,第 k 个最近的点跟点 P 之间的距离称为点 P的 k邻近距离,记为k-distance(p),定义了局部异常因子算法中“局部”的概念,公式如下:
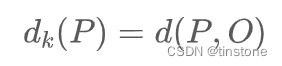
注:点 O 为距离点 P 最近的第 k 个点(k 为正整数,与KNN中的 k 的概念相同)。

比如上图中,距离点 P 最近的第 4 个点是 点4、5、6(即可能存在距离相等的点)。
这里的距离计算可以采用欧氏距离、汉明距离、马氏距离等等,通常采用欧氏距离(公式省略。。。)。
3.2. k距离邻域
以点 P 为圆心,以 k邻近距离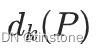 为半径画圆,这个圆以内的范围就是 k 距离邻域,公式如下:
为半径画圆,这个圆以内的范围就是 k 距离邻域,公式如下:
还是以上图所示,假设 k=4,则 1-6均是邻域范围内的点。
3.3. 可达距离
点 P 到点 O 的第 k 可达距离(注意:该距离公式通常容易理解错误),公式如下:
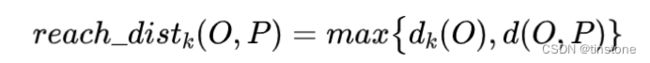
这个可达距离需要特别注意:
- 此处的点 O 为点 P 的 k邻域内的点
- 可达距离是p的邻域点 Nk(p)到p的可达距离,不是p到Nk(p)的可达距离,一定要弄清楚关系,因为该距离不具有对称性(点 P 的 k 近邻与点 O的 k近邻可能不同(密度不同))
通俗理解:点P到点O的可达距离实际上包含两层含义:
- (1) 点O是点P的K邻域内的点
- (2) 可达距离是以点O为对象,对比O的K邻近距离与OP实际距离的大小,然后选择较大的距离作为点P到点O的可达距离
而可达距离代表的数学含义为:
- (1) 此时存在点O的k邻近距离可能小于P的k邻近距离,即点O的密度大导致其k距离范围小,点O的k邻近距离并不包含点P,此时的点P到点O的第k可达距离为OP的距离
- (2) 点P到点O的第k可达距离是以O为对象,选择O的k邻近距离或者OP的实际距离表示了对点P周边点的密度的衡量:P点周边的密度要小于O点周边的密度,然后通过求得点P的K邻域内所有点的可达距离,可以大致判断点P周边的点平均密度(通过理解局部可达密度的概念再来看可达距离更容易理解)。
3.4. 局部可达密度
点 P 的局部可达密度就是借助点 P到其 k邻域(“局部”)内的点(如点O,或者同心圆图例中的1-6的点)的可达距离的平均距离的倒数,具体公式如下:

取倒数的意义:使得距离与密度能达成认知上的一致性:距离越大,密度越小
3.5. 局部异常因子
根据局部可达密度的定义,如果一个数据点与周围数据点的平均密度相似(现在就只考虑密度,暂时忘记距离的概念,不然容易混),则其比值应该趋近于1,因此LOF算法衡量一个数据点的异常程度,并不是看它的绝对局部密度,而是看它跟周围邻近点的相对密度,即比值的概念,因此局部异常因子即是用局部密度来定义的,数据点 P 的局部异常因子为点 P 的 k邻域内的点的平均局部可达密度与点 P 的局部可达密度的比值,公式如下: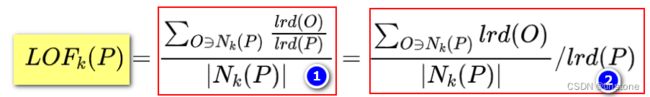
公式理解:
- 点 O 是点 P的 k邻域内的点,lrd§,即为点 P的局部可达密度,而 lrd(O) 表示的则是点 P的 k邻域内的点,基于这些点再去求得点的局部可达密度,即lrd(O)其实就是点P周围点的局部可达密度
- lrd(O) / lrd§ 不就是点P 的 k邻域内某个点的密度与点 P的密度的比值:点周边的密度与点的密度的比值
- 对于标号1变换为标号2,其实就是项的拆分,因为 lrd§是个固定值,直接提出来即可,而且通过这种变形,更容易立即LOF§的含义:点周边的密度的平均值与点的密度的比值
注:取相对密度的好处是可以允许数据分布不均匀、密度不同的情况存在,比如直观理解中C1、C2的点的分布
4.LOF算法流程
了解了LOF的相关概念之后,整个算法也就清晰明了了:
5.LOF算法优缺点
优点:
- LOF 的一个优点是它同时考虑了数据集的局部和全局属性。异常值不是按绝对值确定的,而是相对于它们的邻域点密度确定的。当数据集中存在不同密度的不同集群时,LOF表现良好,比较适用于中等高维的数据集。
缺点:
- LOF中 k参数的选择及调优
- LOF算法中关于局部可达密度的定义其实暗含了一个假设,即:不存在大于等于 k 个重复的点
当这样的重复点存在的时候,这些点的平均可达距离为零,局部可达密度就变为无穷大,会给计算带来一些麻烦。在实际应用时,为了避免这样的情况出现,可以把 k-distance 改为 k-distinct-distance,不考虑重复的情况。或者,还可以考虑给可达距离都加一个很小的值,避免可达距离等于零。
另外,LOF 算法需要计算数据点两两之间的距离,造成整个算法时间复杂度为 。为了提高算法效率,后续有算法尝试改进。FastLOF (Goldstein,2012)先将整个数据随机的分成多个子集,然后在每个子集里计算 LOF 值。对于那些 LOF 异常得分小于等于 1 的,从数据集里剔除,剩下的在下一轮寻找更合适的 nearest-neighbor,并更新 LOF 值。
三、Python代码实现
Python实现LOF代码主要基于sklearn库,主要的代码参数如下:
from sklearn.neighbors import LocalOutlierFactor
主要参数:
n_neighbors:设置k, default=20
contamination: 设置样本中异常点的比例, default=0.1
主要属性:
negative_outlier_factor_: numpy array, shape(n_samples)
和LOF相反的值,值越小,越有可能是异常点,(注:上面提到的LOF的值越接近1, 越可能是正样本, LOF的值越大于1, 则越可能是异常样本)
主要方法:
fit_predict(x): 返回一个数组, -1,表示异常点, 1表示正常点
完整代码
# !/usr/bin/python
# -*- coding:utf-8 -*-
import numpy as np
import matplotlib
import matplotlib.pyplot as plt
from sklearn.neighbors import LocalOutlierFactor
print(__doc__)
np.random.seed(42)
xx, yy = np.meshgrid(np.linspace(-5, 5, 500), np.linspace(-5, 5, 500))
# Generate normal (not abnormal) training observations
X = 0.3 * np.random.randn(100, 2)
X_train = np.r_[X + 2, X - 2]
# Generate new normal (not abnormal) observations
X = 0.3 * np.random.randn(20, 2)
X_test = np.r_[X + 2, X - 2]
# Generate some abnormal novel observations
X_outliers = np.random.uniform(low=-4, high=4, size=(20, 2))
# fit the model for novelty detection (novelty=True)
clf = LocalOutlierFactor(n_neighbors=20, novelty=True, contamination=0.1)
clf.fit(X_train)
# DO NOT use predict, decision_function and score_samples on X_train as this
# would give wrong results but only on new unseen data (not used in X_train),
# e.g. X_test, X_outliers or the meshgrid
y_pred_test = clf.predict(X_test)
y_pred_outliers = clf.predict(X_outliers)
n_error_test = y_pred_test[y_pred_test == -1].size
n_error_outliers = y_pred_outliers[y_pred_outliers == 1].size
# plot the learned frontier, the points, and the nearest vectors to the plane
Z = clf.decision_function(np.c_[xx.ravel(), yy.ravel()])
Z = Z.reshape(xx.shape)
plt.title("Novelty Detection with LOF")
plt.contourf(xx, yy, Z, levels=np.linspace(Z.min(), 0, 7), cmap=plt.cm.PuBu)
a = plt.contour(xx, yy, Z, levels=[0], linewidths=2, colors='darkred')
plt.contourf(xx, yy, Z, levels=[0, Z.max()], colors='palevioletred')
s = 40
b1 = plt.scatter(X_train[:, 0], X_train[:, 1], c='white', s=s, edgecolors='k')
b2 = plt.scatter(X_test[:, 0], X_test[:, 1], c='blueviolet', s=s,
edgecolors='k')
c = plt.scatter(X_outliers[:, 0], X_outliers[:, 1], c='gold', s=s,
edgecolors='k')
plt.axis('tight')
plt.xlim((-5, 5))
plt.ylim((-5, 5))
plt.legend([a.collections[0], b1, b2, c],
["learned frontier", "training observations",
"new regular observations", "new abnormal observations"],
loc="upper left",
prop=matplotlib.font_manager.FontProperties(size=11))
plt.xlabel(
"errors novel regular: %d/40 ; errors novel abnormal: %d/40"
% (n_error_test, n_error_outliers))
plt.show()
四、参考文档
- 一文读懂异常检测 LOF 算法(Python代码)
- 异常点/离群点检测算法——LOF
- 基于LOF的异常检测算法

