二十五章:TransUNet:Transformer为医学图像分割提供强大的编码器
0.摘要
医学图像分割是发展医疗系统的重要先决条件,特别是对于疾病诊断和治疗计划。在各种医学图像分割任务中,U型架构,也称为U-Net,已成为事实上的标准,并取得了巨大的成功。然而,由于卷积操作的固有局部性,U-Net在明确建模长距离依赖性方面通常存在局限性。Transformer是为序列到序列预测而设计的,具有固有的全局自注意机制,但由于缺乏低级细节,可能导致定位能力有限。在本文中,我们提出了TransUNet,它将Transformer和U-Net结合起来,作为医学图像分割的强大替代方案。一方面,Transformer将经过令牌化的图像块从卷积神经网络(CNN)特征图中编码为输入序列,以提取全局上下文。另一方面,解码器将上采样的编码特征与高分辨率的CNN特征图相结合,实现精确的定位。我们认为,Transformer可以作为医学图像分割任务的强大编码器,通过结合U-Net来恢复局部空间信息,从而增强细节。TransUNet在多器官分割和心脏分割等不同医学应用中,取得了优于各种竞争方法的性能。代码和模型可在https://github.com/Beckschen/TransUNet获得。
1.引言
卷积神经网络(CNNs),特别是全卷积网络(FCNs)[8],已经在医学图像分割中占据主导地位。在不同的变体中,U-Net [12]由对称的编码器-解码器网络和跳跃连接组成,以增强细节保留,已成为事实上的选择。基于这一方法,已在广泛的医学应用中取得了巨大的成功,例如基于磁共振(MR)的心脏分割[16],基于计算机断层扫描(CT)的器官分割[7,17,19]以及结肠镜视频中的息肉分割[20]。
尽管基于CNN的方法具有出色的表征能力,但由于卷积操作的固有局部性,这些方法往往在建模明确的长程关系方面存在局限性。因此,这些架构通常对纹理、形状和大小在不同患者之间呈现大的变化的目标结构表现出较弱的性能。为了克服这一限制,现有研究提出了基于CNN特征的自注意机制[13,15]。另一方面,为了序列到序列预测而设计的Transformer已成为替代架构,它完全摒弃了卷积操作符,完全依赖于注意机制[14]。与之前基于CNN的方法不同,Transformer不仅在建模全局上下文方面具有强大的能力,而且在大规模预训练下游任务的可转移性方面表现出优越性。这一成功在机器翻译和自然语言处理(NLP)领域得到了广泛的验证[3,14]。最近,一些尝试也在各种图像识别任务中达到或甚至超过了最先进的性能[4,18]。
在本文中,我们首次研究了在医学图像分割领域中应用Transformer的潜力。然而,有趣的是,我们发现简单地使用Transformer(即使用Transformer对令牌化的图像块进行编码,然后直接将隐藏特征表示上采样为完整分辨率的密集输出)无法产生令人满意的结果。
这是因为Transformer将输入视为一维序列,并且在所有阶段都专注于建模全局上下文,因此导致低分辨率特征缺乏详细的定位信息。这些信息无法通过直接上采样到完整分辨率来有效恢复,因此导致分割结果较粗糙。另一方面,CNN架构(例如U-Net [12])提供了提取低级视觉线索的途径,可以很好地补充这些细致的空间细节。
为此,我们提出了TransUNet,这是第一个医学图像分割框架,从序列到序列预测的角度建立了自注意机制。为了弥补Transformer带来的特征分辨率损失,TransUNet采用混合CNN-Transformer架构,利用CNN特征提取详细的高分辨率空间信息和由Transformer编码的全局上下文。受到U型架构设计的启发,由Transformer编码的自注意特征经过上采样后与来自编码路径的不同高分辨率CNN特征相结合,以实现精确的定位。我们展示了这样的设计使得我们的框架既能保持Transformer的优势,又能受益于医学图像分割。实证结果表明,与以前基于CNN的自注意方法相比,我们基于Transformer的架构提供了更好的利用自注意力的方式。此外,我们观察到更加密集地结合低级特征通常会导致更好的分割准确性。大量实验证明了我们的方法在各种医学图像分割任务上的优越性。
2.相关工作
将CNN与自注意机制相结合。许多研究尝试通过基于特征图对所有像素的全局交互进行建模,将自注意机制集成到CNN中。例如,Wang等人设计了一个非局部操作符,可以插入多个中间卷积层[15]。基于编码器-解码器U型架构,Schlemper等人[13]提出了将加性注意门模块集成到跳跃连接中。与这些方法不同的是,我们在我们的方法中使用Transformer来嵌入全局自注意力。
Transformers. Transformer最初由[14]提出,用于机器翻译,并在许多自然语言处理任务中取得了最先进的成果。为了使Transformer也适用于计算机视觉任务,进行了一些修改。例如,Parmar等人[11]将自注意力仅应用于每个查询像素的局部邻域,而不是全局。Child等人[1]提出了Sparse Transformers,它使用可扩展的近似方法来处理全局自注意力。最近,Vision Transformer (ViT)[4]通过将具有全局自注意力的Transformer直接应用于全尺寸图像,在ImageNet分类任务上取得了最先进的结果。据我们所知,提出的TransUNet是第一个基于Transformer的医学图像分割框架,它基于非常成功的ViT模型构建。
3.方法
给定一个空间分辨率为H×W,通道数为C的图像x ∈ RH×W×C。我们的目标是预测相应的像素级标签地图,大小为H×W。最常见的方法是直接训练一个CNN(例如U-Net),将图像编码为高级特征表示,然后将其解码回完整的空间分辨率。与现有方法不同的是,我们的方法通过使用Transformer将自注意机制引入到编码器设计中。在第3.1节中,我们将首先介绍如何直接应用Transformer来从分解的图像补丁中编码特征表示。然后,在第3.2节中详细介绍TransUNet的整体框架。
3.1.Transformer作为编码器
图像序列化。在[4]的基础上,我们首先通过将输入x重新调整为扁平化的2D补丁序列f(x_i)^p \in R^{P \times P \times C},其中每个补丁的大小为P×P,并且N=HW/P^2是图像补丁的数量(即输入序列的长度)。
补丁嵌入。我们使用可训练的线性投影将向量化的补丁xp映射到一个D维的潜在嵌入空间中。为了编码补丁的空间信息,我们学习特定的位置嵌入,将其添加到补丁嵌入中以保留位置信息,具体如下: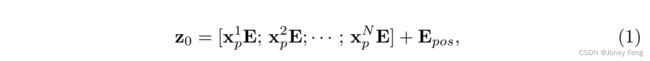 其中E ∈ R^{(P^2 \cdot C) \times D}是补丁嵌入投影,E_{\text{pos}} ∈ R^{N \times D}表示位置嵌入。Transformer编码器由L层多头自注意力(MSA)和多层感知机(MLP)块(Eq.(2)(3))组成。因此,第L层的输出可以写成如下形式:
其中E ∈ R^{(P^2 \cdot C) \times D}是补丁嵌入投影,E_{\text{pos}} ∈ R^{N \times D}表示位置嵌入。Transformer编码器由L层多头自注意力(MSA)和多层感知机(MLP)块(Eq.(2)(3))组成。因此,第L层的输出可以写成如下形式: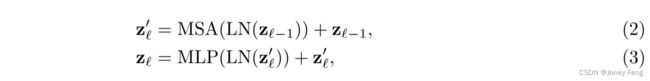
其中LN(·)表示层归一化操作符,z^L是编码的图像表示。Transformer层的结构如图1(a)所示。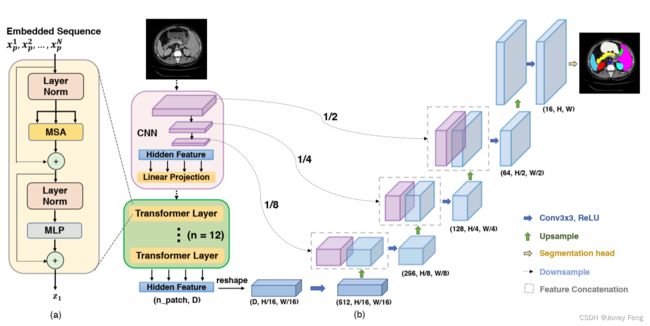
图1:框架概述。(a) Transformer层的示意图;(b) 提出的TransUNet的架构。
3.2.TransUNet
为了进行分割,一种直观的解决方案是将编码的特征表示z^L ∈ R^{H \times W \times P^2 \times D}上采样到完整的分辨率,以预测密集输出。为了恢复空间顺序,编码特征的大小首先应该从HWP^2调整为HP \times WP。我们使用一个1×1的卷积来将调整后的特征通道大小减少到类别数,并且特征图直接进行双线性上采样到完整的分辨率H×W,以预测最终的分割结果。在后面第4.3节中的比较中,我们将这种简单的上采样基准线称为“None”作为解码器设计中的基准。
尽管将Transformer与简单的上采样相结合已经产生了可观的性能,但正如上面提到的,这种策略并不是在分割中最优的Transformer使用方法,因为HP×WP通常远小于原始图像分辨率H×W,因此必然会导致低层次细节(例如器官的形状和边界)的丢失。因此,为了补偿这种信息损失,TransUNet采用了混合的CNN-Transformer架构作为编码器,并使用级联的上采样器实现精确的定位。所提出的TransUNet的概览如图1所示。
CNN-Transformer混合作为编码器。与使用纯Transformer作为编码器(第3.1节)不同,TransUNet采用了CNN-Transformer混合模型,其中CNN首先被用作特征提取器,生成输入的特征图。补丁嵌入不是从原始图像中提取,而是从CNN特征图中提取1×1的补丁。我们选择这种设计的原因是:
1)它允许我们在解码路径中利用中间的高分辨率CNN特征图;
2)我们发现混合的CNN-Transformer编码器比仅使用纯Transformer作为编码器效果更好。
级联上采样器。我们引入了级联上采样器(CUP),它由多个上采样步骤组成,用于解码隐藏特征以输出最终的分割掩码。将隐藏特征z^L ∈ R^{H \times W \times P^2 \times D}的序列重新调整为HP \times WP \times D的形状后,我们通过级联多个上采样块来实例化CUP,以从HP \times WP达到完整的分辨率H \times W,其中每个块依次包含一个2×上采样操作符,一个3×3的卷积层和一个ReLU层。我们可以看到,CUP和混合编码器一起形成了一个U形结构,通过跳跃连接在不同分辨率级别上进行特征聚合。CUP的详细架构以及中间的跳跃连接可以在图1(b)中找到。
4.实验和讨论
4.1.数据集和评估
Synapse多器官分割数据集。我们使用MICCAI 2015多图谱腹部标签挑战中的30个腹部CT扫描,总共包含3779个轴向增强腹部临床CT图像。每个CT体积由85∼198个512×512像素的切片组成,体素空间分辨率为([0:54∼0:54]×[0:98∼0:98]×[2:5∼5:0])mm3。按照[5]的方法,我们报告了8个腹部器官(主动脉、胆囊、脾脏、左肾、右肾、肝脏、胰腺、胃)的平均Dice相似系数(DSC)和平均Hausdorff距离(HD),其中18个训练样本(2212个轴向切片)进行随机划分,12个样本用于验证。
自动心脏诊断挑战赛。ACDC挑战赛收集了来自不同患者的MRI扫描。心脏MR图像是在屏息状态下获取的,一系列的短轴切片从左心室的底部到尖端覆盖整个心脏,切片厚度为5到8毫米。短轴平面的空间分辨率从0.83到1.75 mm2/像素不等。每个患者的扫描结果都进行了手动标注,包括左心室(LV)、右心室(RV)和心肌(MYO)的真实标签。我们报告了平均Dice相似系数(DSC),使用了70个训练样本(1930个轴向切片)进行随机划分,10个样本用于验证,20个样本用于测试。
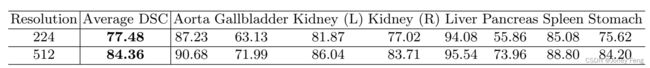
表1:Synapse多器官CT数据集的比较(平均Dice分数%和平均Hausdorff距离(以毫米为单位),以及每个器官的Dice分数%)。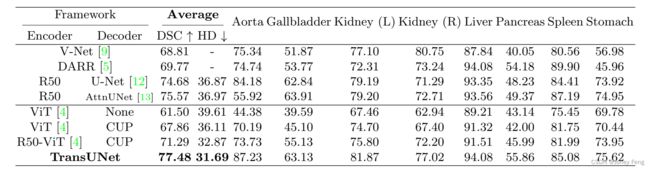
4.2.实现细节
在所有实验中,我们采用简单的数据增强方法,例如随机旋转和翻转。
对于纯Transformer-based编码器,我们简单地采用了具有12个Transformer层的ViT [4]。对于混合编码器设计,我们将ResNet-50 [6]和ViT结合起来,称为“R50-ViT”,在本文中一直采用这种组合。所有的Transformer主干(即ViT)和ResNet-50(表示为“R-50”)都在ImageNet [2]上进行了预训练。输入分辨率和补丁大小P设置为224×224和16,除非另有说明。因此,我们需要在CUP中连续级联四个2×上采样块以达到完整的分辨率。模型使用学习率为0.01、动量为0.9和权重衰减为1e-4的SGD优化器进行训练。默认的批量大小为24,ACDC数据集的默认训练迭代次数为20k,Synapse数据集的默认训练迭代次数为14k。所有实验都是在一块单独的Nvidia RTX2080Ti GPU上进行的。
根据[17,19]的方法,所有的3D体积都以逐层切片的方式进行推断,预测的2D切片被堆叠在一起以重建用于评估的3D预测结果。
4.3.与当前主流方法的比较
我们在Synapse多器官分割数据集上进行了主要实验,将我们的TransUNet与四种先前的最先进方法进行了比较:1) V-Net [9]; 2) DARR [5]; 3) U-Net [12]; 4) AttnUNet [13]。
为了展示我们的CUP解码器的有效性,我们使用ViT [4]作为编码器,并分别比较使用简单的上采样("None")和CUP作为解码器的结果;为了展示我们的混合编码器设计的有效性,我们使用CUP作为解码器,并分别比较使用ViT和R50-ViT作为编码器的结果。为了使ViT混合基线(R50-ViT-CUP)和我们的TransUNet之间的比较公平,我们还将U-Net [12]和AttnUNet [10]的原始编码器替换为预训练于ImageNet的ResNet-50。通过DSC和平均Hausdorff距离(以毫米为单位)的结果在表1中报告。
首先,我们可以看到,与ViT-None相比,ViT-CUP在平均DSC和Hausdorff距离方面分别提高了6.36%和3.50 mm。这种改进表明我们的CUP设计比直接上采样具有更好的解码策略。类似地,与ViT-CUP相比,R50-ViT-CUP在DSC上还额外提高了3.43%,在Hausdorff距离上提高了3.24 mm,这证明了我们的混合编码器的有效性。在R50-ViT-CUP的基础上,我们的TransUNet还配备了跳跃连接,在不同变种的基于Transformer的模型中取得了最好的结果。
其次,表1还显示,所提出的TransUNet在先前的方法上有显著的改进,例如,在平均DSC方面的性能提升范围从1.91%到8.67%不等。特别是,直接应用Transformer进行多器官分割可以得到合理的结果(ViT-CUP的DSC为67.86%),但无法与U-Net或AttnUNet的性能相匹配。这是因为Transformer可以很好地捕捉有利于分类任务的高级语义,但缺乏用于细分医学图像的低级提示。另一方面,将Transformer与CNN结合,即R50-ViT-CUP,优于V-Net和DARR,但仍然产生比纯CNN-based R50-U-Net和R50-AttnUNet差的结果。最后,当与U-Net结构通过跳跃连接相结合时,提出的TransUNet创造了一个新的最先进水平,比R50-ViT-CUP和先前最佳的R50-AttnUNet分别提高了6.19%和1.91%,显示了TransUNet在学习高级语义特征和低级细节方面的强大能力,这在医学图像分割中至关重要。平均Hausdorff距离也呈现出类似的趋势,进一步证明了我们的TransUNet相对于这些基于CNN的方法的优势。
4.4.分析性研究
为了全面评估所提出的TransUNet框架,并验证其在不同设置下的性能,进行了多种消融研究,包括:
1)跳跃连接的数量;
2)输入分辨率;
3)序列长度和补丁大小;
4)模型缩放。
跳跃连接的数量。如上所述,将类似U-Net的跳跃连接集成到TransUNet中有助于通过恢复低级空间信息来增强更精细的分割细节。这个消融的目标是测试在TransUNet中添加不同数量的跳跃连接的影响。通过将跳跃连接的数量变化为0(R50-ViT-CUP)/1/3,在所有8个测试器官的平均DSC上总结了分割性能,如图2所示。请注意,在“1-skip”设置中,我们只在1/4分辨率尺度上添加了跳跃连接。我们可以看到,添加更多的跳跃连接通常会导致更好的分割性能。通过在CUP的三个中间上采样步骤(输出层之前)插入跳跃连接,即在1/2、1/4和1/8分辨率尺度上(如图1所示),可以获得最佳的平均DSC和HD。因此,我们采用这个配置作为我们的TransUNet。值得一提的是,较小器官(如主动脉、胆囊、肾脏、胰腺)的性能提升比较明显,而较大器官(如肝脏、脾脏、胃)的性能提升相对较小。这些结果加强了我们初步的直觉,即将类似U-Net的跳跃连接集成到Transformer设计中,以实现学习精确的低级细节。
作为一个有趣的研究,我们在跳跃连接中应用了加性Transformer,类似于[13],并发现这种新类型的跳跃连接甚至可以进一步提高分割性能。由于GPU内存的限制,我们在1/8分辨率尺度的跳跃连接中采用了一个轻量级的Transformer,而其他两个跳跃连接保持不变。结果表明,这个简单的改变导致了1.4%的DSC性能提升。
关于输入分辨率的影响。TransUNet的默认输入分辨率为224×224。在这里,我们还提供了在高分辨率512×512上训练TransUNet的结果,如表2所示。当使用512×512作为输入时,我们保持相同的补丁大小(即16),这导致Transformer的序列长度大约增加了5倍。正如[4]所指出的,增加有效序列长度可以带来稳健的改进。对于TransUNet来说,将分辨率从224×224改变为512×512,平均DSC提高了6.88%,但计算成本也大大增加。因此,考虑到计算成本,本文中的所有实验比较都是在默认分辨率224×224下进行的,以展示TransUNet的有效性。
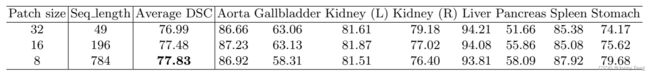
关于补丁大小/序列长度的影响。我们还研究了补丁大小对TransUNet的影响,结果总结在表3中。观察到,通常较小的补丁大小可以获得更高的分割性能。请注意,Transformer的序列长度与补丁大小的平方成反比(例如,补丁大小为16对应序列长度为196,而补丁大小为32对应较短的序列长度为49),因此减小补丁大小(或增加有效的序列长度)可以带来稳健的改进,因为Transformer可以对更长的输入序列中的每个元素之间的更复杂依赖进行编码。遵循ViT [4]中的设置,我们在本文中将16×16作为默认的补丁大小。
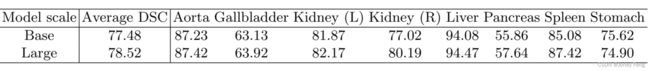
模型缩放。最后,我们对TransUNet的不同模型大小进行了消融研究。具体而言,我们研究了两种不同的TransUNet配置,即“Base”和“Large”模型。对于“Base”模型,隐藏大小D、层数、MLP大小和头数分别设置为12、768、3072和12;而对于“Large”模型,这些超参数分别设置为24、1024、4096和16。从表4中我们可以得出结论,较大的模型可以获得更好的性能。考虑到计算成本,我们在所有实验中采用“Base”模型。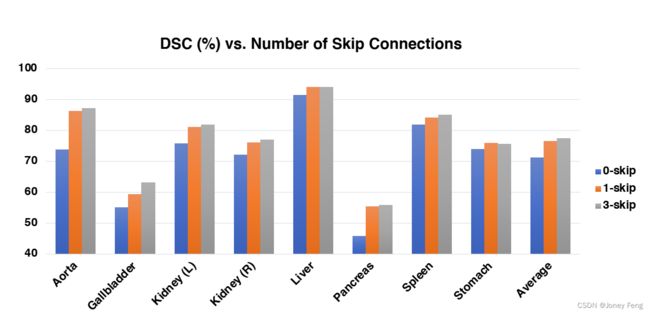
图2:TransUNet中跳跃连接数量的消融研究
图3:通过可视化对不同方法进行的定性比较。从左到右依次为:(a)真实标签,(b)TransUNet,(c)R50-ViT-CUP,(d)R50-AttnUNet,(e)R50-U-Net。我们的方法预测的假阳性较少,并保留了更细致的信息。
表5:在ACDC数据集上的Dice相似系数(DSC)比较(%)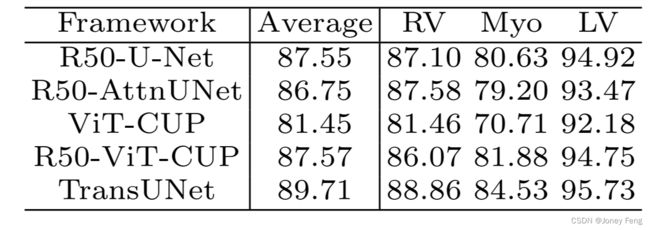
4.5.可视化
我们在Synapse数据集上提供了定性比较的结果,如图3所示。可以看出:
1)纯基于CNN的方法U-Net和AttnUNet更容易出现过分分割或欠分割器官的情况(例如,在第二行中,AttnUNet过分分割了脾脏,而U-Net欠分割了脾脏),这表明基于Transformer的模型,例如我们的TransUNet或R50-ViT-CUP在编码全局上下文和区分语义方面具有更强的能力。
2)第一行的结果显示,与其他方法相比,我们的TransUNet预测的假阳性较少,这表明TransUNet在抑制噪声预测方面比其他方法更具优势。
3)对于Transformer-based模型之间的比较,我们可以观察到与TransUNet相比,R50-ViT-CUP的预测在边界和形状上更加粗糙(例如,第二行中胰腺的预测)。
此外,在第三行中,TransUNet正确预测了左右肾,而R50-ViT-CUP错误地填充了左肾的内部空洞。这些观察结果表明,TransUNet能够进行更精细的分割并保留详细的形状信息。原因是TransUNet同时享受高层次的全局上下文信息和低层次的细节,而R50-ViT-CUP仅依赖于高层次的语义特征。这再次验证了我们将U-Net-like的跳跃连接集成到Transformer设计中以实现精确定位的初步 intu ition。
4.6.泛化到其它数据集
为了展示我们的TransUNet的泛化能力,我们进一步在其他成像模态下进行评估,即MR数据集ACDC,旨在实现自动心脏分割。我们观察到TransUNet相对于纯基于CNN的方法(R50-UNet和R50-AttnUnet)以及其他基于Transformer的基线模型(ViT-CUP和R50-ViT-CUP)都有一致的改进,这与之前在Synapse CT数据集上的结果类似。
5.结论
Transformer被认为是具有强大内在自注意机制的架构。在本文中,我们提出了首个研究探讨使用Transformer进行通用医学图像分割的研究。为了充分发挥Transformer的能力,我们提出了TransUNet,它不仅通过将图像特征视为序列来编码强大的全局上下文,而且还通过U形混合架构设计充分利用了低级别的CNN特征。作为医学图像分割中主导的FCN-based方法的替代框架,TransUNet在各种竞争方法中取得了优越的性能,包括基于CNN的自注意方法。致谢。本工作得到了Lustgarten Foundation for Pancreatic Cancer Research的支持。