J. Med. Chem 2022|TocoDecoy+: 针对机器学习打分函数训练和测试的无隐藏偏差的数据集构建新方法
原文标题:TocoDecoy: A New Approach to Design Unbiased Datasets for Training and Benchmarking Machine-Learning Scoring Functions
论文链接:https://pubs.acs.org/doi/10.1021/acs.jmedchem.2c00460
论文代码:GitHub - 5AGE-zhang/TocoDecoy
参考链接:J. Med. Chem.|TocoDecoy:针对机器学习打分函数训练和测试的无隐藏偏差的数据集构建新方法 - 知乎
MLSFs:基于深度学习的打分函数(针对给定目标的基于结构的虚拟筛选,可以估计配体的结合亲和力和确定结合姿态)。
一、问题
1、传统设计的score functions(SF)存在隐性偏差和数据不足。
分子对接技术被用于:基于结构的虚拟筛选(structure-based virtual screening,SB VS)和计算钓靶(target fishing, TF)。
分子对接可靠性却决于评分函数的准确性。
注:
分子对接技术(Molecular Docking Method, MDM)是指通过计算机模拟将小分子(配体)放置于大分子靶标(受体)的结合区域,再通过计算物理化学参数预测两者的结合力(结合亲和性)和结合方式(构象),进而找到配体与受体在其活性区域相结合时能量最低构象的方法。
计算钓靶(target fishing, TF)是一种能够使用靶标结构信息和生物数据库的数据来识别活性化合物生物靶标的计算方法
诱饵分子:化学性质相似而结构不相似的理论非活性化合物。
2、高质量数据集对于SF和虚拟筛选方法的很重要
PDBbinding数据集(2004):具有相应生物活性和/或结合亲和力的蛋白质-配体复合物共晶结构的统一存储库
DUD数据集(2006)、DUD-E(2012):收集了针对各种目标的活性物质,而且还提供了人工生成的非活性物质(诱饵),这些非活性物质具有与活性物质相似的物理化学性质,但拓扑结构不同。
DEKOIS(2011):自动过程,可为任何给定的活性物创建量身定制的诱饵,2013年开发了DEKOIS 2.0,其中包含81个新的结构多样化的诱饵集
Maximum Unbiased Validation (MUV):无偏数据集,通过收集PubChem BioAssay中经过实验验证的活性和非活性化合物,用于验证VS方法
大量传统的数据集是基于传统SFs,不适合用于benchmark
偏差包括四种主要类型:人工富集、模拟偏差、域偏差和非因果偏差
人工富集:是由于活性物与诱饵物(Decoy)的物理化学性质存在显著差异而引起的一种偏置。
相似偏差:数据集中的化合物结构过于相似,模型的测试表现过于乐观/泛化能力有限。
同样,如果数据集具有有限的化学多样性,那么在该数据集上训练的MLSF/SF可能会遭受域偏差(即,该MLSF/SF仅适用于预测与数据集中的配体共享相似支架的配体)。
域偏差:数据集中的结构多样性太低,模型只适用于预测训练集中出现的特定骨架的化合物。
非因果偏差:由于ML非线性拟合和不可解释的黑盒机制。ML算法很容易通过学习不同的拓扑特征而不是物理上有意义的蛋白质-配体相互作用来区分活性物和诱饵。
为了解决非因果偏差,在以一种方式构造诱饵的训练集上训练模型,并在另一种方式收集诱饵的测试集上测试模型(不是数据集中的拓扑结构,但是模型仍然可以从训练集中学习到非因果偏差),或者只使用交互特征而不是拓扑信息来训练MLSF/SF(尽管在预期的SBVS中表现出色,但缺乏配体结构信息可能会阻止它们的进一步改进)。因此,仍然迫切需要设计无偏和现实的数据集,专门用于基准测试和训练。
第一个相对无偏的数据集,LIT-PCBA(2020)。采用几种脱偏技术,如选择性地将活性和非活性化合物保持在相似的分子性质范围内以去除人工富集。
另一个重要贡献来自于活性分子的三维构象的使用,通过引入DL简单数据增强,解决活性和诱饵之间的大量拓扑差异造成的非因果偏差(适当地减少数据集的偏差)。
诱饵是通过随机旋转和移动DUD-E中活性分子的对接构象(每个活性分子有三个诱饵)或将每个活性分子重新对接到相应的蛋白质口袋中产生的(与活性分子位姿至少有5 Å根均方距离的三个最高位姿被标记为诱饵),增强的数据集可能会迫使ML算法学习蛋白质和配体之间的物理相互作用,而不是配体的性质。此外,作者没有深入研究mlsf是否能够从随机产生的结合姿态中一致地学习蛋白质-配体相互作用。
DeepCoy(深度生成模型):使用有监督的训练过程来利用分子对进行学习。以一个活性分子作为输入,生成一个属性匹配但结构不同的诱饵。然而,DeepCoy生成的数据集可能仍然存在非因果偏差,因为它在与DUDE相同的假设下定义了诱饵。但是,只有25万个分子子集而不是更大的数据集被用于训练,从而多样性存在问题。
二、模型方法MATERIALS AND METHODS
针对4中偏差:在100万数量级的数据集上训练了条件分子生成模型,确保模型能够生成结构多样的分子(去除域偏差);通过条件分子生成模型来控制生成的分子与活性分子物理化学性质相似(去除人工富集);通过T-SNE算法把化合物映射到二维化学空间并进行格点过滤(去除相似偏差);引入两种诱饵构建策略:假设与活性分子结构相似度较低的分子为负样本、假设活性分子与靶标的错误结合构象为负样本(去除非因果偏差)
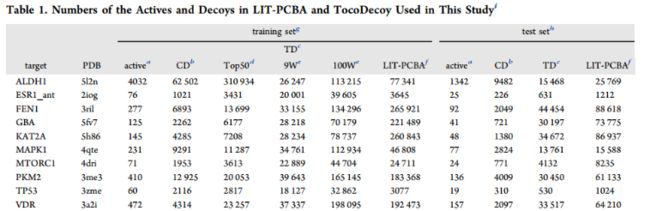
active是target的活性配体的数量,CD活性配体与蛋白质口袋对接时产生的对接得分较差的构象诱饵数量;TD指提供了与从cRNN生成的活性物具有相似物理化学性质但与活性物具有不同拓扑结构的拓扑诱饵的数量。Top50是指top 50拓扑诱饵是基于与相应活性配体的拓扑相似性而不是网格滤波来选择的。xW(9W/100W)表示对诱饵进行网格滤波,网格号为x × 104,其中x为数字。LIT-PCBA表示LIT-PCBA数据集中非活性配体的个数。training set将以4:1的比例进一步分割为训练集和验证集。test测试集:用于模型测试的数据集。TocoDecoy的数量= Active + CD + TD_Top50。TocoDecoy_9W = Active + CD + TD_9W。LIT-PCBA的个数= Active + LIT-PCBA。DUD-E = Active + TD_Top50.
1、数据集
DatasetA: CHEMBL v25. 规则:(1)对ChEMBL数据集中配体的片段、电荷、同位素、立体化学和互变异构状态进行标准化;(2)分子中含有H、C、N、O、F、S、Cl和Br原子,且重原子数量小于50个;(3)将剩余配体进一步划分为训练集和测试集(分别为1 347 173和149 679个配体),cRNN模型的比例为9:1。数据集A用于cRNN建模。
DatasetB: LIT-PCBA, 该数据集中的配体经过了实验验证,对于MLSF的构建和基准测试而言相对无偏。选择了10个靶点子集(即ALDH1、ESR1_ant、FEN1、GBA、KAT2A、MAPK1、MTORC1、PKM2、TP53和VDR)作为数据集B。用LIT-PCBA中的活性分子和靶标生成TocoDecoy数据集。
DatasetC: 即TocoDecoy数据集,是基于数据集B中的活性分子生成的。
DatasetD: 由于数据集B中选择的靶标没有相应的DUD-E数据集,而TD集(数据集C的一个子集)与DUD-E的诱饵生成策略相似,因此选择TD集作为DUD-E数据集的替代方案。
2、数据集生成
(1)将“种子”配体(活性配体)的六个物理化学性质(MW,分子量;logP,油水分配系数;RB,可旋转键数量;HBA,氢键受体数量;HBD,氢键供体数量;HAL,卤键数量)输入到条件循环神经网络(conditional recurrent neural network,cRNN)模型中,以生成属性匹配的decoys。cRNN为每个活性配体共生成了200个有效且不重复的decoys。
(2)尽管生成的decoys可能具有相似的物理化学性质,并且其拓扑结构大概率与其种子(活性)配体不同,但总有一些例外。因此,decoys在与活性配体的理化性质相比,Dice相似性(DS)不符合以下要求将被过滤掉:(i)MW±40 Da,(ii)logP±1.5,(iii)RB±1,(iv)HBA±1,(v)HBD±1,(vi)HAL±1,(vii)DS<0.4。
(3)对每个分子依次计算ECFP和T-SNE向量,然后进行格点过滤,以消除由相似结构引起的相似偏差;保留的decoys形成拓扑诱饵集(Topology Decoys,TD),这些decoys的对接构象是通过对经过结构预处理的蛋白质和配体的分子对接获得的。首先,利用RDKit计算配体的ECFP指纹,然后利用scikit-learn中T-SNE算法将2048位ECFP非线性映射到二维向量上,实现配体在化学空间中的分布可视化。计算每个维度的最小值和最大值,并使用固定的步长将二维向量分割成不同的间隔。二维化学空间被划分为由每个维度的间隔形成的网格,通过去除其他拓扑相似的化合物,每个网格中只保留一个配体。还通过可视化TocoDecoy的化学空间来检查和比较网格过滤前后的模拟偏差。化学空间中的点分布越稀疏,数据集包含的模拟偏差就越小。
(4)按照表S1中列出的相应对接分数阈值来过滤活性配体的对接构象,对接分数低于阈值的构象被作为decoys构象保留,从而产生构成构象诱饵集(Conformation Decoys,CD)。
(5)最后,将TD和CD集整合为最终的TocoDecoy数据集。
3、任务
首先根据LIT-PCBA中的活性分子生成TocoDecoy数据集,然后系统地研究LIT-PCBA和TocoDecoy中的隐藏偏差,包括人工富集、相似偏差、域偏差和非因果偏差。通过基于描述符的XGBoost和基于端到端图的IGN模型进行评估。XGBoost算法可以直接输出样本的预测标签,而IGN模型只能输出样本活跃或不活跃的概率。
MLSFs评价指标:
n为活性物个数,n为配体个数,ri为第i个活性配体的排名,Ra为数据集中活性物的比例,α(本研究为80.5)为与早期识别相关的参数。
BED_ROC (α = 80.5)用于评估mlsf的筛选能力,因为SFs的主要应用之一是其筛选能力是指评分函数在随机分子池中识别给定目标蛋白的真正结合物的能力与结合亲和预测不同,筛选能力更侧重于在得分最高的分子中富集潜在活性分子。
Hidden Biases评价指标:
DOE分数可用于评价活性物与诱饵物之间物理化学性质分布不平衡造成的人工富集,DOE分数越小,人工富集程度越低。
域偏差评价,I(D)值越大,表明数据集中配体的多样性越大:
式6中xi,j,norm为化合物i的理化性质j的归一化值,xj,95%和xj,5%分别为理化性质j的第95和第5百分位。式9中,a、m、i、n分别表示活性配体a、活性配体数、诱饵i、诱饵数。式10中,D为数据集的大小,x和y表示数据集中的配体,|mx∩my|表示配体x和y的共同分子指纹(ECFP)的个数,|mx∩my|表示指纹总数。
三、实验
1、人工富集
在TocoDecoy中,活性物的可旋转键数(RB)、氢键受体数(HBA)、氢键给体数(HBD)和卤素数(HAL)的分布与诱饵物的分布接近。
DOE分数较低的TocoDecoy优于LIT-PCBA
2、模拟偏差、域偏差
对于每个活性配体,导致活性与诱饵的比例为1:100。此外,将原始TocoDecoy数据集分别传递给网格编号为90000(即300 × 300)和1000000(即1000 × 1000)的网格过滤器,从而得到TocoDecoy_9W和TocoDecoy_100W数据集。数据集的大小随着网格数的减少而减小。由于深度学习算法需要大量数据,因此使用90000网格数进行严格滤波以消除模拟偏差,而使用1000000网格数进行松散滤波以保证配体的数量足够大以训练可靠的IGN模型。
为了探索网格过滤器对数据集的影响,我们通过生成包含数据集中每个分子拓扑信息的2048位ECFP来可视化数据集的化学空间,然后T-SNE算法将2048维ECFP降为二维向量,用于绘图。以ALDH1和MAPK1为例,TocoDecoy和LIT-PCBA的偏化学空间和无偏化学空间如图所示。
可以观察到配体在化学空间中的分布是不均匀的,大量结构相似的配体在脱偏前堆叠在一起。此外,TocoDecoy比LIT-PCBA覆盖了更大的化学空间(图中更宽的区域),这表明与LIT-PCBA相比,TocoDecoy可能包含更少的域偏差。
为了进一步探索网格滤波器对去偏的影响,我们在这些数据集上训练IGN模型,并在LIT-PCBA的测试集上进行测试,其性能如图4。
在去偏数据集(TocoDecoy_9W和TocoDecoy_100W)上训练的模型在大多数目标上的表现优于在有偏TocoDecoy数据集上训练的模型。因此,我们得出以下结论:(1)TocoDecoy比LIT-PCBA包含更少的域偏置,(2)网格滤波器有助于消除模拟偏差;(3)用网格滤波去偏数据集训练的模型比用模拟偏数据集训练的模型泛化能力相对更好。
3、非因果偏差
XGBoost模型在CD set和TocoDecoy的Glide SF能量项上训练的性能。TD、CD、TC和LI分别代表TD集、CD集、TocoDecoy集和LIT-PCBA。@之前的数据集是训练集,@之后的数据集是测试集。例如,TC@LI列中的F1分数表示在TocoDecoy上训练并在LIT-PCBA测试集上测试的模型的性能。CD集和TD集从TocoDecoy_9W集中提取。
在CD训练集上训练的XGBoost模型在CD测试集上表现出良好的性能,而在其他测试集上表现不佳,这表明在CD训练集上训练的模型在基于对接分数的分类上存在偏差,泛化能力有限。
当使用TocoDecoy进行训练时,模型在CD测试集上的表现变得更差,这意味着TD集的加入有助于最小化对接分数带来的非因果偏差。同样,XGBoost模型分别使用TD和TocoDecoy中配体的ECFP进行训练,并在LITPCBA、CD、TD和TocoDecoy的测试集上进行测试。
在ECFP上训练的XGBoost模型在TD集和TocoDecoy上的性能。TD、CD、TC、LI分别代表TD集、CD集、TocoDecoy集和LIT-PCBA。@之前的数据集是训练集,@之后的数据集是测试集。例如,TC@LI列中的F1分数表示在TocoDecoy上训练并在LIT-PCBA测试集上测试的模型的性能。CD集和TD集从TocoDecoy_9W集中提取。
基于ECFP在TD集上训练的XGB模型在LITPCBA、CD集和TocoDecoy测试集上的表现要比在TD测试集上的表现差得多。
4、在不同数据集上训练得到的模型在模拟虚拟筛选中的表现
在各种数据集上训练了IGN模型,并在LIT-PCBA的测试集上测试了这些模型。为了更好地了解在TocoDecoy上训练的模型的泛化能力,除了TocoDecoy之外,作者还在DUD-E和LIT-PCBA上训练了IGN模型作为对照。如图A所示,IGN模型在F1分数和BED_ROC方面优于Glide SP,这表明在虚拟筛选中,MLSF优于传统SF。BED_ROC和Precision方面优于TocoDecoy和DUD-E训练的模型,因为LIT-PCBA训练集的数据分布比TocoDecoy和DUD-E的测试集更类似于LIT-PCBA的测试集。显然,在DUD-E上训练的模型偏向于通过分子拓扑结构差异区分活性分子和非活性分子,不能泛化到LIT-PCBA的测试集。同样,在TocoDecoy上训练的模型也并不能在虚拟筛选中很好的泛化。然而,TocoDecoy训练的模型在F1分数、BED_ROC和Precision方面优于DUD-E训练的模型,这表明TocoDecoy训练的模型具有相对更好的泛化能力。
如图6B所示,在十个靶标中的九个(除了ESR1_ant)靶标数据集上,在TocoDecoy上训练的模型比在DUD-E上训练的模型取得了更高的F1分数,在十个靶标中的五个靶标数据集上,在LIT-PCBA上训练的模型预测性能弱于TocoDecoy数据集上训练的模型。
四、总结
TocoDecoy对比了传统的数据集DUD-E和适用于MLSFs评价的无隐藏偏差数据集LIT-PCBA。在四种隐藏偏差的验证中,TocoDecoy表现较另外两个数据集相当/更少的隐藏偏差。在模拟虚拟筛选实验中,在不同数据集上训练的模型的预测精度排名为:LIT-PCBA≈TocoDecoy>DUD-E。尽管TocoDecoy与LIT-PCBA上训练的模型性能相当,但TocoDecoy数据集是可扩展的,生成足够大的无偏TocoDecoy数据集用于MLSF的建模和基准测试是可行的。