用python实现传染病模型传染病模型
用python实现传染病模型
- 1.SI模型
-
- 1.1 代码实现
- 1.2 模型的结果
- 2.SIS (治愈后仍然还是易感者)
-
- 2.1 代码实现
- 2.2模型的结果
- 3 SIR模型(治愈后直接移除)
-
- 3.2代码实现
- 3.2绘制图像:
- 4.SEIR 模型(新增一个人群,叫潜伏者E)
-
- 4.1代码实现
- 4.2模型的结果
参考文章为:https://www.kesci.com/mw/project/60161bf6ac79f40016b7d7d9
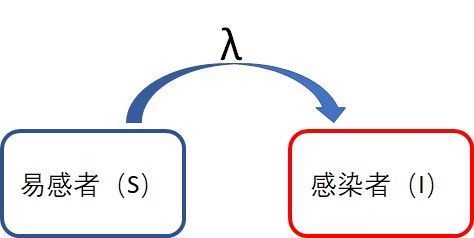
1.SI模型
示意图:
我们假设城市有一千万(N=10的7次方)人,每个患者每天接触感染每天0.8人(lamda=0.8),初始感染人数为45人(i0 = 45/N),我们来模拟70天(T=70)的情况。
1.1 代码实现
import numpy as np
import matplotlib.pyplot as plt
#population
N = 1e7
#simuation Time
T = 70
# susceptiable ratio 易感染比例
s = np.zeros([T])
# infective ratio 感染比例
i = np.zeros([T])
# 由我们的实验可以知道,lamda是有取值范围的,似乎超过1就不可以了
# concat rate
lamda = 0.9
# initial infective people
i[0] = 45.0 / N
s[0] = 1-i[0]
for t in range(T-1):
i[t+1] = i[t] + i[t] * lamda * (1.0 - i[t])
s[t+1] = 1 - i[t+1]
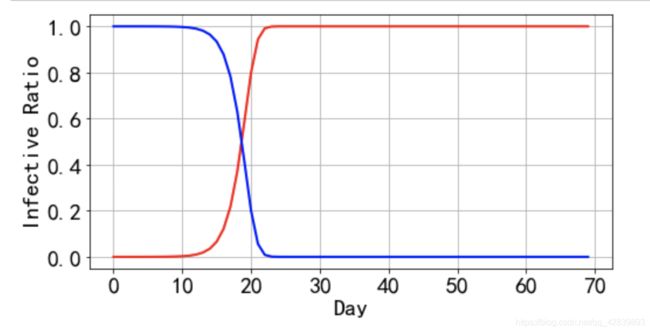
1.2 模型的结果
#感染者随着天数的变化曲线
fig, ax = plt.subplots(figsize=(8,4))
ax.plot(i, c='r', lw=2)
ax.plot(s, c='b', lw=2)
ax.set_xlabel('Day',fontsize=20)
ax.set_ylabel('Infective Ratio', fontsize=20)
ax.grid(1)
plt.xticks(fontsize=20)
plt.yticks(fontsize=20);
由上图可见,大约在25天左右,全部人群都会变成感染者,感染率为1
lamda的现实意义就是该城市的卫生水平,衡量的是消毒,隔离这些措施执行得怎么样。
回到传染病模型,按照SI模型计算的结果,我们全人类都会患病,这好可怕!原因是我们忽略了一个很重要的因素,那就是我们有奋斗在一线的医护人员,我们会被治愈!所以SI模型只适合研究具有高传染风险又不能被治愈的病(比如HIV)。
但是对于其他病,我们是可以靠医疗和自身免疫系统康复的,那么紧接着的一个问题就是,被治愈后还会再被传染上嘛?根据这个问题的回答不同,我们有了两个不同的模型,SIR 和 SIS。现在可以揭晓,SIR的R的含义了,就是移出者(Removed),现实含义就是指被治愈后不会再被感染的人。 而SIS表示治愈后仍然还是易感者。下面我们用python来分别实现这两个模型。
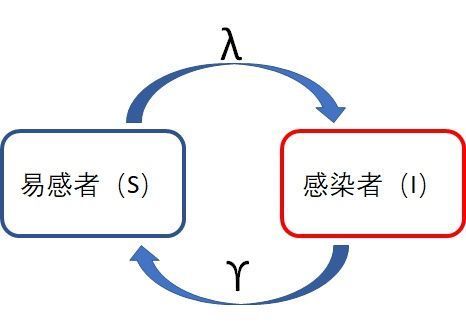
2.SIS (治愈后仍然还是易感者)
为了实现这个模型,我们需要引入新的一个参数,治愈率 γ \gamma γ。好啦,先上我们的新示意图:
和SI模型做比较,区别就是计算感染者的增加数时要减去被治愈的人数。
所以这时候每天的增加的感染者为: λ × i N × s − γ × i N \lambda \times i N \times s-\gamma \times i N λ×iN×s−γ×iN ,
增加的感染率为: λ × i × s − γ i \lambda \times i \times s-\gamma i λ×i×s−γi 。
模型完成啦,修改python代码:
2.1 代码实现
# susceptiable ratio
s = np.zeros([T])
# infective ratio
i = np.zeros([T])
# concat rate(这个其实是有效的感染率)
lamda = 1.0
# recover rate(治愈率)
gamma = 0.5
# initial infective people
i[0] = 45.0 / N
s[0] = 1-i[0]
for t in range(T-1):
i[t+1] = i[t] + i[t] * (1- i[t])* lamda - i[t] * gamma
s[t+1] = 1 - i[t+1]
2.2模型的结果
fig, ax = plt.subplots(figsize=(8,4))
ax.plot(i, c='r', lw=2)
ax.plot(s, c='b', lw=2)
ax.set_xlabel('Day',fontsize=20)
ax.set_ylabel('Infective Ratio', fontsize=20)
ax.grid(1)
plt.xticks(fontsize=20)
plt.yticks(fontsize=20);
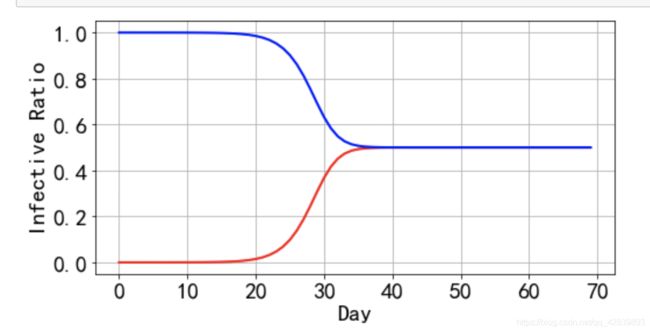
可以看到,达到最大感染率的时间退后10天左右,最后感染和治愈达到动态平衡,人群中有始终有一半的人感染着。所以,SIS模型适合研究具有传染性和反复性的流行病,比如常见流感。同样的,感兴趣的话,改变lamda和gamma的值,观察曲线的变化。和lamda不同的是,gamma的现实意义就是对这种疾病的治疗水平。
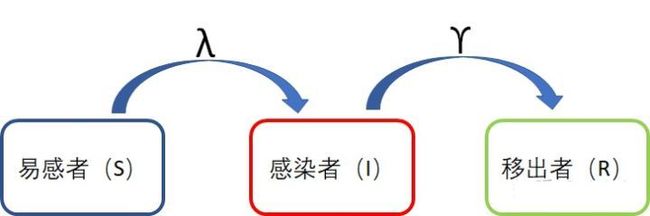
3 SIR模型(治愈后直接移除)
加入了移出者,被治愈的病人不会再被传染,先上我们的新示意图:
SIR 模型
注意到这里,人群被分成了三类,不再只有I和S,所以相比于之前的模型,我们需要找到新的约束关系。现在我们需要分别计算三种人每天的增加量了:
- 易感者:每天都在被传染,所以一直在减少,减少量为被传染的人数: λ N i s \lambda N i s λNis
- 感染者:增加了被感染的人,减少了治愈的人: λ N i s − γ N i \lambda N i s-\gamma N i λNis−γNi
- 移出者:增加了治愈的人: γ N i \gamma N i γNi
建模完成,修改python代码,并且假设人群普遍易感,新型疾病,初始没有移出者。
γ = 0.0821 , λ = 0.2586 , 初 始 易 感 人 数 为 一 千 万 , 初 始 感 染 10 人 , 那 么 我 们 的 城 市 总 人 数 N = 1 e 7 + 10 \gamma=0.0821,\lambda=0.2586 ,初始易感人数为一千万, 初始感染10人,那么我们的城市总人数 N=1 e 7+10 γ=0.0821,λ=0.2586,初始易感人数为一千万,初始感染10人,那么我们的城市总人数N=1e7+10
3.2代码实现
# population
N = 1e7 + 10
# simuation Time / Day
T = 170
# susceptiable ratio
s = np.zeros([T])
# infective ratio
i = np.zeros([T])
#remove ratio
r = np.zeros([T])
# contact rate
lamda = 0.2586
# recover rate
gamma = 0.0821
# initial infective people
i[0] = 10.0 / N
s[0] = 1e7 / N
for t in range(T-1):
i[t+1] = i[t]+i[t]* lamda * s[t] - gamma*i[t]
s[t+1] = s[t] - lamda * i[t]*s[t]
r[t+1] =r[t] + gamma * i[t]
3.2绘制图像:
fig, ax = plt.subplots(figsize=(10,6))
ax.plot(s, c='b', lw=2, label='S')
ax.plot(i, c='r', lw=2, label='I')
ax.plot(r, c='g', lw=2, label='R')
ax.set_xlabel('Day',fontsize=20)
ax.set_ylabel('Infective Ratio', fontsize=20)
ax.grid(1)
plt.xticks(fontsize=20)
plt.yticks(fontsize=20)
plt.legend();
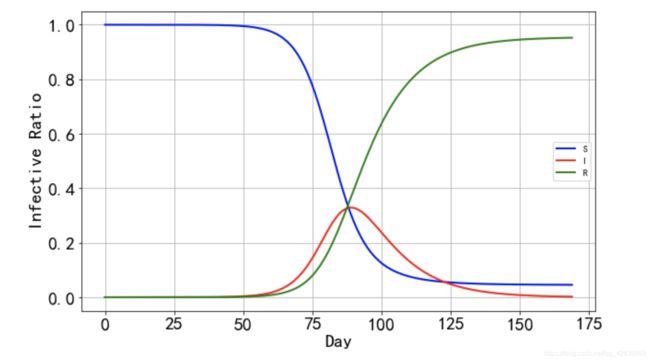
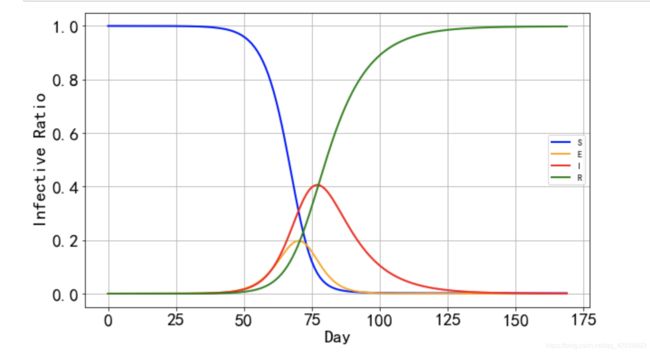
感染人数峰值发生在一个月左右,最大感染人数不到人群的20%, 但是最终人群的80%都会得此病(就是最终的移出者的比例)。SIR模型适合研究没有潜伏期的急性传染病,治疗后能够痊愈并具有抗病性。
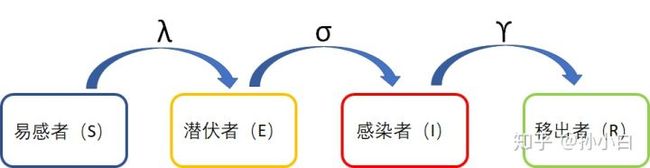
4.SEIR 模型(新增一个人群,叫潜伏者E)
但是,SIR模型和实际情况的出入会比较大,因为忽略了太多因素了,比如说潜伏期,比如说政策调控,药物,出生死亡等等。下面我们可以和前面一样,把潜伏期考虑进去,新增一个人群,叫潜伏者E(exposed):
SEIR模型
同样的我们需要计算各人群每天的增加量:
S:每天减少: λ N i s \lambda N i s λNis
E:每天增加传染,减少发病: λ N i s − σ N e \lambda N i s-\sigma N e λNis−σNe
I:每天增加发病,减少治愈: σ N e − γ N i \sigma N e-\gamma N i σNe−γNi
R:每天增加治愈: γ N i \gamma N i γNi
建模完成,修改我们的python程序,这里的 σ \sigma σ可以理解为潜伏期的倒数。给的4天。新型冠状病毒给目前临床的潜伏期是3-14天。
4.1代码实现
# population
N = 1e7 + 10 + 5
# simuation Time / Day
T = 170
# susceptiable ratio
s = np.zeros([T])
# exposed ratio
e = np.zeros([T])
# infective ratio
i = np.zeros([T])
# remove ratio
r = np.zeros([T])
# contact rate
lamda = 0.5
# recover rate
gamma = 0.0821
# 潜伏期
# exposed period
sigma = 1 / 4
# initial infective people
i[0] = 10.0 / N
s[0] = 1e7 / N
e[0] = 40.0 / N
for t in range(T-1):
s[t + 1] = s[t] - lamda * s[t] * i[t]
e[t + 1] = e[t] + lamda * s[t] * i[t] - sigma * e[t]
i[t + 1] = i[t] + sigma * e[t] - gamma * i[t]
r[t + 1] = r[t] + gamma * i[t]
4.2模型的结果
fig, ax = plt.subplots(figsize=(10,6))
ax.plot(s, c='b', lw=2, label='S')
ax.plot(e, c='orange', lw=2, label='E')
ax.plot(i, c='r', lw=2, label='I')
ax.plot(r, c='g', lw=2, label='R')
ax.set_xlabel('Day',fontsize=20)
ax.set_ylabel('Infective Ratio', fontsize=20)
ax.grid(1)
plt.xticks(fontsize=20)
plt.yticks(fontsize=20)
plt.legend();