【无监督学习之聚类】
聚类
-
- 0.简介
- 距离 和 相似度
- 1. K均值聚类(kmeans)
-
- 模型
- 算法
- 特点
- 2. 谱聚类(Spectral clustering)
-
- 算法思想
- 特点
- 谱聚类的具体步骤:
- 算法步骤:
- 3.小结
- 参考资料
0.简介
聚类:针对给定的样本,依据他们的属性的相似度或距离,将其归并到若干个“簇”或“类”的数据分析问题。
- 类:样本的子集。直观上,相似的样本聚集在同类,不相似的样本分散在不同类。
距离 和 相似度
距离 和 相似度量在聚类中十分重要。
常用的距离度量有:
-
闵可夫斯基距离
d i j = ( ∑ k = 1 m ∣ x k i − x k j ∣ p ) 1 p p ≥ 1 d_{ij}=\left(\sum_{k=1}^m|x_{ki}-x_{kj}|^p\right)^\frac{1}{p}\\p \ge 1 dij=(k=1∑m∣xki−xkj∣p)p1p≥1 -
欧氏距离
-
曼哈顿距离
-
切比雪夫距离
-
马哈拉诺比斯距离
d i j = [ ( x i − x j ) T S − 1 ( x i − x j ) ] 1 2 d_{ij}=\left[(x_i-x_j)^\mathrm TS^{-1}(x_i-x_j)\right]^{\frac{1}{2}} dij=[(xi−xj)TS−1(xi−xj)]21
常用的相似度量有:
-
相关系数
x r i j = ∑ k = 1 m ( x k i − x ˉ i ) ( x k j − x ˉ j ) [ ∑ k = 1 m ( x k i − x ˉ i ) 2 ∑ k = 1 m ( x k j − x ˉ j ) 2 ] 1 2 x ˉ i = 1 m ∑ k = 1 m x k i x ˉ j = 1 m ∑ k = 1 m x k j x r_{ij}=\frac{\sum\limits_{k=1}^m(x_{ki}-\bar x_i)(x_{kj}-\bar x_j)}{\left[\sum\limits_{k=1}^m(x_{ki}-\bar x_i)^2\sum\limits_{k=1}^m(x_{kj}-\bar x_j)^2\right]^\frac{1}{2}}\\\bar x_i=\frac{1}{m}\sum\limits_{k=1}^mx_{ki}\\\bar x_j=\frac{1}{m}\sum\limits_{k=1}^mx_{kj} xrij=[k=1∑m(xki−xˉi)2k=1∑m(xkj−xˉj)2]21k=1∑m(xki−xˉi)(xkj−xˉj)xˉi=m1k=1∑mxkixˉj=m1k=1∑mxkj -
夹角余弦
s i j = ∑ k = 1 m x k i x k j [ ∑ k = 1 m x k i 2 ∑ k = 1 m x k j 2 ] 1 2 s_{ij}=\frac{\sum\limits_{k=1}^m x_{ki}x_{kj}}{\left[\sum\limits_{k=1}^mx_{ki}^2\sum\limits_{k=1}^mx_{kj}^2\right]^\frac{1}{2}} sij=[k=1∑mxki2k=1∑mxkj2]21k=1∑mxkixkj
用距离度量相似度时,距离越小表示样本越相似。用相关系数时,相关系数越大表示样本越相似。
1. K均值聚类(kmeans)
K均值聚类是常用的聚类方法。
模型
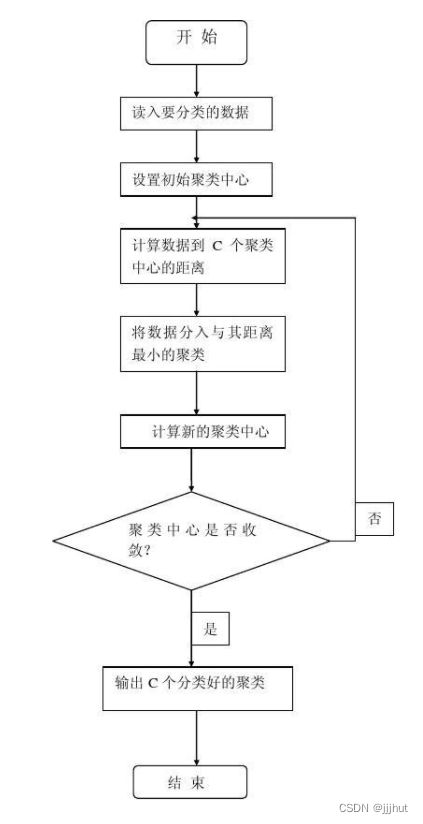
算法
- 首先选择k个类的中心
- 将样本分到与其最近的中心所在的类,得到一个初始的聚类结果
- 然后计算每个类的样本的均值,作为类的新的中心
- 重复以上步骤,直到收敛(类的中心随着迭代不发生位移)为止
import numpy as np
import matplotlib.pyplot as plt
# kmeans
class MyKmeans:
def __init__(self, k, n=20):
self.k = k #质心个数
self.n = n #迭代次数
def fit1(self, x, centers=None):
# step1 : 随机选择 K 个点, 或者指定
if centers is None:
idx = np.random.randint(low=0, high=len(x), size=self.k)
centers = x[idx]
#print(centers)
else:
centers = x[centers]
inters = 0 # 迭代计数器
# 迭代n次或质心不发生移动时结束循环
while inters < self.n:
print(inters)
print(centers)
points_set = {key: [] for key in range(self.k)}#聚类集合
# step2,遍历所有点 P,将 P 放入最近的聚类中心的集合中
for p in x:
nearest_index = np.argmin(np.sum((centers - p) ** 2, axis=1) ** 0.5)#计算最近的质心
points_set[nearest_index].append(p)#将p放入计算出的最近的聚类中心的点集中
# step3,遍历每一个点集,更新每个点集的质心
for i_k in range(self.k):
centers[i_k] = sum(points_set[i_k])/len(points_set[i_k])
inters += 1
return points_set, centers
# 随机生成一个样本数为100的二维点集
np.random.seed(123)
data = np.random.rand(100,2)
data = data*10
# 聚类
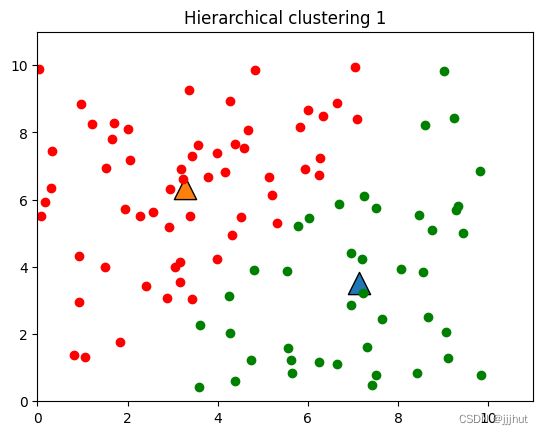
m = MyKmeans(2,50)
points_set1, centers1 = m.fit1(data,centers=[0,1])
# 结果可视化
cat1 = np.asarray(points_set1[0])
cat2 = np.asarray(points_set1[1])
#绘制图像
#plt.figure(1)
for ix, p in enumerate(centers1):
plt.scatter(p[0], p[1], color='C{}'.format(ix), marker='^', edgecolor='black', s=256)
plt.scatter(cat1[:,0], cat1[:,1], color='green')
plt.scatter(cat2[:,0], cat2[:,1], color='red')
plt.title('Hierarchical clustering 1')
plt.xlim(0, 11)
plt.ylim(0, 11)
plt.show()
特点
K均值聚类属于基于划分的聚类方法;
- 类别(簇)数 k k k需要事先设定
- 距离度量或相似度:欧氏距离平方
- 以中心或样本的均值表示类别
- 以样本和其所属类的类中心的距离的总和作为可优化的目标函数。
- 得到的类别是平坦的,分层次化的
- 算法属于迭代算法,不能保证得到全局最优
2. 谱聚类(Spectral clustering)
谱聚类是一种基于图论的聚类算法,在介绍谱聚类算法原理之前,首先介绍下图的相关概念。
算法思想
它的主要思想:把所有数据看成空间中的点,这些点之间可以用边连接起来,距离较远的两个点之间的边权重较低,而距离较近的两个点之间的权重较高,通过对所有数据点组成的图进行切图,让切图后的不同的子图间边权重和尽可能小(即距离远),而子图内的边权重和尽可能高(即距离近)。
特点
谱聚类相较于前面讲到的最最传统的k-means(当然了,也是很好的一个聚类方法,只不过大家都拿它来做个baseline)聚类方法,谱聚类又具有许多的优点:
1.只需要待聚类点之间的相似度矩阵就可以做聚类了。
2.对于不规则的数据(或者说是离群点)不是那么敏感,个人感觉主要体现在最小化图切割的公式中(因为在RatioCut的优化策略中要除以一个分母|A|,表示当前聚类中含有点 的个数;另外一种策略是Ncut,会除以一个分母vol(A),表示的是当前聚类中的点的度的集合;有了这两个分母的话,若是离群点单独成一个聚类,会造成这两个优化函数的值变大)
3.k-means聚类算法比较适合于凸数据集(数据集内的任意两点之间的连线都在该数据集以内,简单理解就是圆形,可能不准确),而谱聚类则比较通用。
谱聚类的具体步骤:
1.相似度矩阵S的构建,构建相似度的矩阵的过程中,可以使用欧氏距离、余弦相似度、高斯相似度等来计算数据点之间的相似度,选用哪个要根据你自己的实际情况来。不过在谱聚类中推荐使用的是高斯相似度,但是我在我的工程中使用的是余弦相似度。
2.相似度图G的构建,主要有一下三种方法:
-
近邻图:在这种图中,我们将距离小于某个阈值的点连接起来。
-
K近邻图:在这种图中,以点i,j为例,如果i是j的k近邻之一,那么将i与j连起来。当然,这会造成相似度图G是一种有向图,因为i是j的k近邻之一,但是j不一定是i的k近邻之一。很像我们生活中:你是我的最好朋友,但是我不一定是你的最好朋友。有两种方法可以将此时的图化为无向图:(1)忽略边的方向性,即只要满足i是j的k近邻之一或者j是i的k近邻之一,那么我们就将与连起来。
(2)只有满足i是j的k近邻之一而且j是i的k近邻之一,我们才将与连起来。依照(1)方法所得的图被称为k近邻图,依照(2)方法所得的图被称为互k近邻图。 -
全连接图:在这种图中,我们将所有的点都相互连接起来,同时所有的边的权重设置为相似度。这种图建立的关键在于相似度函数的确立,相似度函数能够较好地反映实际中的相邻关系,那么效果较好。一个相似度函数的例子为高斯相似度函数:,其中参数的选取是个难点。该方法中的参数的作用与近邻图中的的作用是相似的。
通过构建上述的相似度图G我们可以重新得到一个邻接矩阵W.
3.拉普拉斯矩阵
它的定义很简单,拉普拉斯矩阵。是度矩阵,也就是相似度矩阵的每一行(或者每一列)加和得到的一个对角矩阵。W就是图的邻接矩阵。
4.无向图的切割(也就是求L的特征值和特征向量,之后对特征向量矩阵进行k-means聚类)
算法步骤:
输入:样本集D,相似矩阵的生成方式, 降维后的维度, 聚类方法,聚类后的维度
输出: 簇划分
(1) 根据输入的相似矩阵的生成方式构建样本的相似矩阵S
(2)根据相似矩阵S构建邻接矩阵W,构建度矩阵D
(3)计算出拉普拉斯矩阵L
(4)求L的最小的个特征值所各自对应的特征向量
(6) 将特征向量组成维的特征矩阵F
(7)对F中的每一行作为一个维的样本,共n个样本,用输入的聚类方法进行聚类,聚类维数为。
(8)得到簇划分
import numpy as np
import matplotlib.pyplot as plt
from sklearn import cluster, datasets
from sklearn.preprocessing import StandardScaler
np.random.seed(0)
# 构建数据
n_samples = 1500
noisy_circles = datasets.make_circles(n_samples=n_samples, factor=0.5, noise=0.05)
noisy_moons = datasets.make_moons(n_samples=n_samples, noise=0.05)
blobs = datasets.make_blobs(n_samples=n_samples, random_state=8)
data_sets = [
(
noisy_circles,
{
"n_clusters": 2
}
),
(
noisy_moons,
{
"n_clusters": 2
}
),
(
blobs,
{
"n_clusters": 3
}
)
]
colors = ["#377eb8", "#ff7f00", "#4daf4a"]
affinity_list = ['rbf', 'nearest_neighbors']
plt.figure(figsize=(17, 10))
for i_dataset, (dataset, algo_params) in enumerate(data_sets):
# 模型参数
params = algo_params
# 数据
X, y = dataset
X = StandardScaler().fit_transform(X)
for i_affinity, affinity_strategy in enumerate(affinity_list):
# 创建SpectralCluster
spectral = cluster.SpectralClustering(
n_clusters=params['n_clusters'],
eigen_solver='arpack',
affinity=affinity_strategy
)
# 训练
spectral.fit(X)
# 预测
y_pred = spectral.labels_.astype(int)
y_pred_colors = []
for i in y_pred:
y_pred_colors.append(colors[i])
plt.subplot(3, 4, 4*i_dataset+i_affinity+1)
plt.title(affinity_strategy)
plt.scatter(X[:, 0], X[:, 1], color=y_pred_colors)
plt.show()
3.小结
本文介绍的聚类方法有 K均值聚类方法,谱聚类方法。
K均值聚类属于基于划分的聚类方法;类别(簇)数 k k k需要事先设定;距离度量或相似度:欧氏距离平方,以中心或样本的均值表示类别,以样本和其所属类的类中心的距离的总和作为可优化的目标函数。得到的类别是平坦的,分层次化的,算法属于迭代算法,不能保证得到全局最优
谱聚类只需要数据之间的邻接矩阵,因此对于处理稀疏数据的聚类很有效。这点传统聚类算法比如K-Means很难做到。由于使用了降维,因此在处理高维数据聚类时的复杂度比传统聚类算法好,如果最终聚类的维度非常高,则由于降维的幅度不够,谱聚类的运行速度和最后的聚类效果均不好
聚类效果依赖于邻接矩阵,不同的邻接矩阵得到的最终聚类效果可能很不同。
常用的聚类方法除了这些还有,层次化聚类方法,基于混合分布的方法,基于密度的方法。以上的方法是对于样本的聚类方法,除此之外还有对样本和属性同时聚类的方法,如Co-Clustering。
参考资料
《统计学习方法》李航