- 到底DB::listen(function ($query) { ... });为什么是回调函数?
快点好好学习吧
Laravel数据库
DB::listen(function($query){...});是Laravel中用于监听数据库查询的一个方法。它的核心作用是通过回调函数捕获和处理每个执行的SQL查询及其相关信息。这种设计的选择(使用回调函数)是基于灵活性、解耦性和事件驱动架构的考虑。1.为什么使用回调函数?在DB::listen()方法中,使用回调函数的主要原因包括:a)灵活性回调函数允许开发者以灵活的方式处理每个查询事件
- Android8.0一些系统数据库的变更总结
留给时光吧
1.SettingsProvider之前的一些系统设置内容如亮度、音量大小等都存储在settings.db这个数据库中,但在8.0上数据库不见了。在8.0上都存在几个xml文件中了,其实从6.0开始就已经开始了这种操作,只不过6.0上并没有删除原始数据库,从7.0开始系统删除了原始数据库。简单看一下源码:android\frameworks\base\packages\SettingsProvid
- Python,Go开发光电效应与日常应用APP
Geeker-2025
pythongolang
以下是一个基于Python与Go开发的光电效应科普与应用APP的完整技术方案,结合了物理原理模拟、实时数据处理及生活场景应用,参考了工业级开发实践(如光电实验数据处理和能源设备控制):---###一、系统架构设计```mermaidgraphLRA[Go微服务层]-->B[Python科学计算层]A-->C[数据库/物联网]B-->D[硬件接口]D-->E[传感器/实验设备]subgraph前端A
- [Python] -项目实战5- Python 实现简易学生成绩管理系统
踏雪无痕老爷子
Pythonpython开发语言
一、为什么做这个项目?学习OOP和GUI基础:通过类与对象封装学生信息,熟悉Tkinter构建窗口、表格、按钮等。实用性强:可添加、查询、删除、修改学生记录,是常见管理系统的基本功能。扩展性好:后续可以接入数据库、图表展示、权限控制等功能。二、核心技术与工具tkinter:Python内置的桌面GUI库,用于构建窗口界面、表单和按钮。sqlite3:轻量级关系数据库,适合小型持久化存储,无需部署服
- Kettle--MySQL生产数据库千万、亿级数据量迁移方案及性能优化
m0_67401761
面试学习路线阿里巴巴android前端后端
大家好,我是贾斯汀!【实战前言】(1)不管你是学生,还是已经工作了的小伙伴,可能你在过去、现在或者未来,会遇到这样的问题,公司/项目用的是Oracle/DB2/MySQL等关系型数据库,因公司发展需求,需要完成旧数据库数据安全迁移到新数据库的重要使命,新旧数据库可能是同一种类型的数据库,也可能是不同类型的数据库,相同类型数据库还好,比如都是MySQL数据库,那么你主要只需要考虑如何将数据安全、高效
- 数据库第八次作业--备份和索引
倪旻萱
数据库
一、备份与恢复作业:创库,建表:CREATEDATABASEbooksDB;usebooksDB;CREATETABLEbooks(bk_idINTNOTNULLPRIMARYKEY,bk_titleVARCHAR(50)NOTNULL,copyrightYEARNOTNULL);CREATETABLEauthors(auth_idINTNOTNULLPRIMARYKEY,auth_nameVAR
- PostgreSQL常用命令与工具指南
Mr.小海
Linux服务器postgresql数据库算法架构网络协议linux运维开发
文章目录PostgreSQL常用命令与工具指南简介1.连接与基本操作连接数据库环境变量设置(避免密码输入)常用元命令2.数据库与表管理数据库操作创建数据库删除数据库修改数据库属性表操作创建表修改表结构删除表索引管理创建索引删除索引3.数据操作(CRUD)插入数据查询数据更新数据删除数据事务控制4.账号与权限管理角色/用户操作创建角色修改角色删除角色权限控制授予权限撤销权限查看权限5.常用函数字符串
- 人脸识别:AI 如何精准 “认人”?
田园Coder
人工智能科普人工智能科普
1.人脸识别的基本原理:从“看到脸”到“认出人”1.1什么是人脸识别技术人脸识别是基于人的面部特征信息进行身份认证的生物识别技术。它通过摄像头采集人脸图像,利用AI算法提取面部特征(如眼距、鼻梁高度、下颌轮廓等),再与数据库中的模板比对,最终判断“是否为同一个人”。与指纹识别、虹膜识别等生物识别技术相比,人脸识别的优势在于“非接触性”(无需触碰设备)和“自然性”(符合人类习惯,如刷脸支付无需额外操
- redis-缓存三剑客(缓存击穿,缓存穿透,缓存雪崩)
hzx790688184
redisredis
redis-缓存击穿,缓存穿透,缓存雪崩缓存三剑客(缓存击穿,缓存穿透,缓存雪崩)缓存击穿请求一个不存在的数据时,请求到数据库,数据库不存在该数据,会导致每次请求都会到数据库缓存穿透当热点key过期时,突然大量请求访问,直接访问到数据库缓存雪崩大批量的key同时失效,或redis宕机,导致大量的请求直接访问数据库缓存三剑客(缓存击穿,缓存穿透,缓存雪崩)缓存击穿请求一个不存在的数据时,请求到数据库
- 缓存三剑客解决方案
爱学习的小熊猫_
缓存redis
缓存三剑客解决方案1.缓存雪崩定义:大量缓存数据在同一时间点集体失效,导致所有请求直接穿透到数据库,引发数据库瞬时高负载甚至崩溃。解决方案:设置过期随机值,避免大量缓存同时失效。//缓存雪崩防护:随机过期时间+双层缓存//设置随机过期时间(基础时间+随机偏移)Randomrandom=newRandom();longexpire=baseExpire+random.nextInt(5*60*100
- PHPStorm携手ThinkPHP8:开启高效开发之旅
奔跑吧邓邓子
项目攻略phpstormThinkPHPThinkPHP8php开发
目录一、前期准备1.1开发环境搭建1.2配置Xdebug二、PHPStorm集成ThinkPHP82.1导入ThinkPHP8项目2.2配置PHP解释器2.3配置服务器三、ThinkPHP8项目开发基础3.1项目结构剖析3.2控制器与方法创建3.3视图渲染与数据传递四、数据库操作与模型定义4.1数据库配置4.2模型定义与使用4.3数据库迁移与种子五、高级开发技巧与优化5.1路由优化与管理5.2中间
- 秒杀系统设计思路
先生zeng
昨天遇到这个问题,发现自己临时总结的不是很好,所以现在想重新整理一下思路。分析一下问题:类似淘宝那种做秒杀系统活动,你是如何设计的?场景分析:1.需到达某个时刻才可以开始秒杀(某个时刻之前需要控制拒绝请求)。2.一瞬间大量的请求到后台,服务器,数据库,缓存都会扛不住。(前端拦截、削峰,限流)3.满足条件才可以进行秒杀(最先过滤这些不满足条件的)4.防止恶意刷单请求,网站攻击(SQL注入,CSRF)
- 七、Zabbix — Proxy分布式监控
胖胖不胖、
《Zabbix速学即学即用》zabbix分布式服务器运维监控
目录配置Zabbix-proxy代理1.安装代理2.安装并配置数据库(proxy不能与zabbix-server共享数据库)3.发送zabbix-server源码包中初始化脚本到proxy主机并导入数据库4.修改代理配置文件5.web页面添加并配置代理Zabbix-agent客户端配置1.修改配置文件2.web页面修改,把这些主机修改为通过代理获取数据减少zabbix-server压力便于多地设备
- 某国产数据库“热情过头”的发布会,该收收了!
IT邦德
数据库国产数据库数据库
作者:IT邦德中国DBA联盟(ACDU)成员,15年DBA工作经验Oracle、PostgreSQLACECSDN博客专家及B站知名UP主,全网粉丝15万+擅长主流Oracle、MySQL、PG、高斯及Greenplum备份恢复,安装迁移,性能优化、故障应急处理文章目录01兼容性?02多模融合?03性能玄学?04智能运维?05终极暴击总结哥们儿,还记得十年前DBA圈那场著名论战不?OLTP和OLA
- Java实现简单秒杀功能
在商城项目中,秒杀功能可以说是必不可少的,下面我将使用SpringBoot集成Redis、RabbitMQ、MyBatis-Plus和MySQL来实现一个简单的秒杀系统,系统将包含以下核心功能:使用Redis进行库存预减和用户限流;使用RabbitMQ进行异步下单,提高系统吞吐量;使用MyBatis-Plus操作MySQL数据库;利用Redis执行Lua脚本的原子性防止商品超卖;接口限流(使用Re
- 分布式定时器:原理设计与技术挑战
你一身傲骨怎能输
架构设计分布式
文章摘要分布式定时器用于在分布式系统中可靠、准确地触发定时任务,常见实现方案包括:基于数据库/消息队列的定时扫描、分布式任务调度框架(如Quartz集群、xxl-job)、时间轮/延迟队列(如Redis/Kafka)以及Zookeeper/Etcd协调服务。主要技术挑战包括时钟同步、任务幂等、高可用、负载均衡和故障恢复等。核心难点在于保证任务唯一性、调度精度与分布式一致性,技术选型需权衡轻量级(R
- 常用 SQL 语句摘录
未来无限
C#Winform设计
语句功能--数据操作SELECT--从数据库表中检索数据行和列INSERT--向数据库表添加新数据行DELETE--从数据库表中删除数据行UPDATE--更新数据库表中的数据--数据定义CREATETABLE--创建一个数据库表DROPTABLE--从数据库中删除表ALTERTABLE--修改数据库表结构CREATEVIEW--创建一个视图DROPVIEW--从数据库中删除视图CREATEINDE
- 应用层流量与缓存累积延迟解析
你一身傲骨怎能输
计算机网络缓存
文章摘要应用层流量指OSI模型中应用层协议(如HTTP、gRPC)产生的数据交互,常见于Web请求、微服务通信等场景。缓存累积延迟指多级缓存或消息队列机制中,各级延迟叠加导致数据更新滞后,例如数据库更新后,因消息队列、缓存刷新等环节延迟,用户最终看到的数据可能滞后数秒。两者分别描述了网络通信的数据流机制和分布式系统中的延迟问题。1.应用层流量应用层流量,一般指的是在网络通信的OSI七层模型中,**
- 数据库第四次作业
dustcell.
数据库
1.将mydb9_stusys数据库下的student、sc和course表,备份到本地主机保存为st_msg_bak.sql文件,然后将数据表恢复到自建的db_test数据库中;--备份mysqldump-uroot-pmydb9_stusysstudentsccourse>st_msg_bak.sql#创建目标数据库mysql-uroot-p-e"CREATEDATABASEdb_test;"
- 数据库第五次作业
要求1.定义触发器实现在产品表(product)中每多一个产品,就在操作表(operate)中记录操作方式和时间以及编号记录。注:操作说明:标记执行delete、insert、update2.定义触发器实现在产品表(product)中每更新一个产品,就在操作表(operate)中记录操作方式和时间以及编号记录。3.定义触发器实现在产品表(product)中每删除一个产品就,在操作表(operate
- Java双重检测锁解决MySQL和Redis数据一致性问题
Java双重检测锁解决MySQL和Redis数据一致性问题双重检测锁(Double-CheckedLocking)是一种在多线程环境下优化性能的设计模式,可以用于解决MySQL和Redis之间的数据一致性问题。下面我将介绍如何实现这一方案。问题背景在MySQL和Redis双存储系统中,常见的一致性问题包括:缓存穿透:查询不存在的数据,导致每次请求都打到数据库缓存击穿:热点key失效瞬间,大量请求直
- SQL学习十二、插入数据
沐左
前面学习记录的都是查询数据,那些也是SQL中最常用的语句,这篇我们来学习和积累一下相数据库中插入数据的SQL。另外,需要注意的是,很多数据库对查询操作要求的权限可能低一些,但是对于插入、修改、删除等操作都需要较高的权限。INSERTINTO语句1、插入完整的行例如我们需要插入一条新的订单记录可以这样写:写法一:无需指定要插入数据的列名,只需提供被插入的值即可。SQL格式INSERTINTOtabl
- Jetpack Compose 中 Kotlin 协程的使用
儿歌八万首
jetpackandroidandroiduiComposekotlin协程
本文将带你系统性地理解JetpackCompose中协程的使用场景、API设计理念、生命周期对齐、性能优化与常见误区,助你写出既响应迅速又稳定可靠的现代AndroidUI代码。1.为什么在Compose中离不开协程传统View系统就离不开异步:网络请求、数据库I/O、动画……进入声明式UI时代,异步需求不仅没减少,反而更复杂——状态驱动让数据变化更加频繁。协程凭借以下优势成为Compose首选:语
- SpringBoot架构下智慧物流管理系统设计详解
本文还有配套的精品资源,点击获取简介:本文详细讲解了如何利用SpringBoot框架构建智慧物流管理系统,并涉及关键技术和实现原理。文章首先介绍了SpringBoot的核心组件和工作原理,然后探讨了RESTfulAPI的构建、数据模型与数据库设计、GPS定位服务集成、权限控制和认证、微服务化以及系统测试等关键方面。这一系统结合Java技术优势,提供了一个高效、智能化的物流行业解决方案。1.Spri
- 【设计模式&C#】外观模式(用于解决客户端对系统的许多类进行频繁沟通)
大飞pkz
设计模式设计模式外观模式c#
一种结构性设计模式。特点是将复杂的子系统调用逻辑封装到一个外观类,从而使客户端更容易与系统交互。优点:简化了接口的调用;降低了客户端与子系统的耦合度;封装了子系统的逻辑。缺点:引入了额外的类,可能会增加不必要的复杂性;不适合需要频繁修改的系统。外观类承担的职责过多适合的场景:家庭影院系统;网络服务端的连接;数据库的访问。//Car类,即外观类usingSystem;publicclassCar{/
- 医疗AI与融合数据库的整合:挑战、架构与未来展望(上)
Allen_Lyb
数智化教程(第二期)人工智能数据库架构
引言随着人工智能(AI)在医疗健康领域的广泛应用,数据已成为医疗AI发展的核心驱动力。然而,医疗数据具有极度的异构性(包括结构化电子病历、医学影像向量、基因组JSON/图结构、传感器时序等),传统数据架构难以高效整合。因数据孤岛、复杂ETL流程以及昂贵维护成本,医疗AI平台通常难以充分发挥价值。融合数据库(ConvergedDatabase/多模态一体化数据库)通过支持SQL、JSON、图、向量、
- C#使用Quartz.NET详细讲解
diaochejiang2761
c#
C#使用Quartz.NET详细讲解Quartz.NET是一个开源的作业调度框架,是OpenSymphony的QuartzAPI的.NET移植,它用C#写成,可用于winform和asp.net应用中。它提供了巨大的灵活性而不牺牲简单性。你能够用它来为执行一个作业而创建简单的或复杂的调度。它有很多特征,如:数据库支持,集群,插件,支持cron-like表达式等等。你曾经需要应用执行一个任务吗?这个
- 本地开发用ASP.NET Core Web API项目创建及测试
way_hj
web开发asp.net后端
1.服务端代码(C#)1.1创建ASP.NETCoreWebAPI项目打开VisualStudio2022。选择“创建新项目”。选择“ASP.NETCoreWebAPI”模板,点击“下一步”。输入项目名称(如OracleApi),选择项目位置,点击“创建”。1.2安装Oracle数据库依赖使用NuGet管理包工具在项目中安装Oracle数据库的EFCore依赖:Oracle.EntityFrame
- 【数据库】触发器 Trigger
有事请出门右转
触发器Trigger概念:数据库中的一个对象,相当于JS中的监听器触发器可以监听增删改三个动作比如说我想监听一张表,只要我增删改了这张表中的数据,我就可以触发这个触发器,去往另外一张表中记录一下日志语法:DELIMITER$$CREATETRIGGER`数据库名`.`触发器名`BEFORE/AFTERINSERT/UPDATE/DELETEON`数据库名`.`要监听的表名`FOREACHROW--
- ASP.NET Core Web API 内存缓存(IMemoryCache)入门指南
王维志
asp.netwebapiasp.net缓存后端c#.net
在WebAPI开发中,频繁访问数据库或第三方服务可能会带来性能瓶颈。为了提升接口响应速度并减轻后端压力,使用缓存是非常有效的优化手段。本文将带你快速上手ASP.NETCore提供的内存缓存(IMemoryCache),无需安装额外库,轻量、简单、适合中小项目什么是IMemoryCache?IMemoryCache是ASP.NETCore自带的缓存机制,使用服务器内存来存储数据对象,单机部署即可使用
- PHP,安卓,UI,java,linux视频教程合集
cocos2d-x小菜
javaUIlinuxPHPandroid
╔-----------------------------------╗┆
- zookeeper admin 笔记
braveCS
zookeeper
Required Software
1) JDK>=1.6
2)推荐使用ensemble的ZooKeeper(至少3台),并run on separate machines
3)在Yahoo!,zk配置在特定的RHEL boxes里,2个cpu,2G内存,80G硬盘
数据和日志目录
1)数据目录里的文件是zk节点的持久化备份,包括快照和事务日
- Spring配置多个连接池
easterfly
spring
项目中需要同时连接多个数据库的时候,如何才能在需要用到哪个数据库就连接哪个数据库呢?
Spring中有关于dataSource的配置:
<bean id="dataSource" class="com.mchange.v2.c3p0.ComboPooledDataSource"
&nb
- Mysql
171815164
mysql
例如,你想myuser使用mypassword从任何主机连接到mysql服务器的话。
GRANT ALL PRIVILEGES ON *.* TO 'myuser'@'%'IDENTIFIED BY 'mypassword' WI
TH GRANT OPTION;
如果你想允许用户myuser从ip为192.168.1.6的主机连接到mysql服务器,并使用mypassword作
- CommonDAO(公共/基础DAO)
g21121
DAO
好久没有更新博客了,最近一段时间工作比较忙,所以请见谅,无论你是爱看呢还是爱看呢还是爱看呢,总之或许对你有些帮助。
DAO(Data Access Object)是一个数据访问(顾名思义就是与数据库打交道)接口,DAO一般在业
- 直言有讳
永夜-极光
感悟随笔
1.转载地址:http://blog.csdn.net/jasonblog/article/details/10813313
精华:
“直言有讳”是阿里巴巴提倡的一种观念,而我在此之前并没有很深刻的认识。为什么呢?就好比是读书时候做阅读理解,我喜欢我自己的解读,并不喜欢老师给的意思。在这里也是。我自己坚持的原则是互相尊重,我觉得阿里巴巴很多价值观其实是基本的做人
- 安装CentOS 7 和Win 7后,Win7 引导丢失
随便小屋
centos
一般安装双系统的顺序是先装Win7,然后在安装CentOS,这样CentOS可以引导WIN 7启动。但安装CentOS7后,却找不到Win7 的引导,稍微修改一点东西即可。
一、首先具有root 的权限。
即进入Terminal后输入命令su,然后输入密码即可
二、利用vim编辑器打开/boot/grub2/grub.cfg文件进行修改
v
- Oracle备份与恢复案例
aijuans
oracle
Oracle备份与恢复案例
一. 理解什么是数据库恢复当我们使用一个数据库时,总希望数据库的内容是可靠的、正确的,但由于计算机系统的故障(硬件故障、软件故障、网络故障、进程故障和系统故障)影响数据库系统的操作,影响数据库中数据的正确性,甚至破坏数据库,使数据库中全部或部分数据丢失。因此当发生上述故障后,希望能重构这个完整的数据库,该处理称为数据库恢复。恢复过程大致可以分为复原(Restore)与
- JavaEE开源快速开发平台G4Studio v5.0发布
無為子
我非常高兴地宣布,今天我们最新的JavaEE开源快速开发平台G4Studio_V5.0版本已经正式发布。
访问G4Studio网站
http://www.g4it.org
2013-04-06 发布G4Studio_V5.0版本
功能新增
(1). 新增了调用Oracle存储过程返回游标,并将游标映射为Java List集合对象的标
- Oracle显示根据高考分数模拟录取
百合不是茶
PL/SQL编程oracle例子模拟高考录取学习交流
题目要求:
1,创建student表和result表
2,pl/sql对学生的成绩数据进行处理
3,处理的逻辑是根据每门专业课的最低分线和总分的最低分数线自动的将录取和落选
1,创建student表,和result表
学生信息表;
create table student(
student_id number primary key,--学生id
- 优秀的领导与差劲的领导
bijian1013
领导管理团队
责任
优秀的领导:优秀的领导总是对他所负责的项目担负起责任。如果项目不幸失败了,那么他知道该受责备的人是他自己,并且敢于承认错误。
差劲的领导:差劲的领导觉得这不是他的问题,因此他会想方设法证明是他的团队不行,或是将责任归咎于团队中他不喜欢的那几个成员身上。
努力工作
优秀的领导:团队领导应该是团队成员的榜样。至少,他应该与团队中的其他成员一样努力工作。这仅仅因为他
- js函数在浏览器下的兼容
Bill_chen
jquery浏览器IEDWRext
做前端开发的工程师,少不了要用FF进行测试,纯js函数在不同浏览器下,名称也可能不同。对于IE6和FF,取得下一结点的函数就不尽相同:
IE6:node.nextSibling,对于FF是不能识别的;
FF:node.nextElementSibling,对于IE是不能识别的;
兼容解决方式:var Div = node.nextSibl
- 【JVM四】老年代垃圾回收:吞吐量垃圾收集器(Throughput GC)
bit1129
垃圾回收
吞吐量与用户线程暂停时间
衡量垃圾回收算法优劣的指标有两个:
吞吐量越高,则算法越好
暂停时间越短,则算法越好
首先说明吞吐量和暂停时间的含义。
垃圾回收时,JVM会启动几个特定的GC线程来完成垃圾回收的任务,这些GC线程与应用的用户线程产生竞争关系,共同竞争处理器资源以及CPU的执行时间。GC线程不会对用户带来的任何价值,因此,好的GC应该占
- J2EE监听器和过滤器基础
白糖_
J2EE
Servlet程序由Servlet,Filter和Listener组成,其中监听器用来监听Servlet容器上下文。
监听器通常分三类:基于Servlet上下文的ServletContex监听,基于会话的HttpSession监听和基于请求的ServletRequest监听。
ServletContex监听器
ServletContex又叫application
- 博弈AngularJS讲义(16) - 提供者
boyitech
jsAngularJSapiAngularProvider
Angular框架提供了强大的依赖注入机制,这一切都是有注入器(injector)完成. 注入器会自动实例化服务组件和符合Angular API规则的特殊对象,例如控制器,指令,过滤器动画等。
那注入器怎么知道如何去创建这些特殊的对象呢? Angular提供了5种方式让注入器创建对象,其中最基础的方式就是提供者(provider), 其余四种方式(Value, Fac
- java-写一函数f(a,b),它带有两个字符串参数并返回一串字符,该字符串只包含在两个串中都有的并按照在a中的顺序。
bylijinnan
java
public class CommonSubSequence {
/**
* 题目:写一函数f(a,b),它带有两个字符串参数并返回一串字符,该字符串只包含在两个串中都有的并按照在a中的顺序。
* 写一个版本算法复杂度O(N^2)和一个O(N) 。
*
* O(N^2):对于a中的每个字符,遍历b中的每个字符,如果相同,则拷贝到新字符串中。
* O(
- sqlserver 2000 无法验证产品密钥
Chen.H
sqlwindowsSQL ServerMicrosoft
在 Service Pack 4 (SP 4), 是运行 Microsoft Windows Server 2003、 Microsoft Windows Storage Server 2003 或 Microsoft Windows 2000 服务器上您尝试安装 Microsoft SQL Server 2000 通过卷许可协议 (VLA) 媒体。 这样做, 收到以下错误信息CD KEY的 SQ
- [新概念武器]气象战争
comsci
气象战争的发动者必须是拥有发射深空航天器能力的国家或者组织....
原因如下:
地球上的气候变化和大气层中的云层涡旋场有密切的关系,而维持一个在大气层某个层次
- oracle 中 rollup、cube、grouping 使用详解
daizj
oraclegroupingrollupcube
oracle 中 rollup、cube、grouping 使用详解 -- 使用oracle 样例表演示 转自namesliu
-- 使用oracle 的样列库,演示 rollup, cube, grouping 的用法与使用场景
--- ROLLUP , 为了理解分组的成员数量,我增加了 分组的计数 COUNT(SAL)
- 技术资料汇总分享
Dead_knight
技术资料汇总 分享
本人汇总的技术资料,分享出来,希望对大家有用。
http://pan.baidu.com/s/1jGr56uE
资料主要包含:
Workflow->工作流相关理论、框架(OSWorkflow、JBPM、Activiti、fireflow...)
Security->java安全相关资料(SSL、SSO、SpringSecurity、Shiro、JAAS...)
Ser
- 初一下学期难记忆单词背诵第一课
dcj3sjt126com
englishword
could 能够
minute 分钟
Tuesday 星期二
February 二月
eighteenth 第十八
listen 听
careful 小心的,仔细的
short 短的
heavy 重的
empty 空的
certainly 当然
carry 携带;搬运
tape 磁带
basket 蓝子
bottle 瓶
juice 汁,果汁
head 头;头部
- 截取视图的图片, 然后分享出去
dcj3sjt126com
OSObjective-C
OS 7 has a new method that allows you to draw a view hierarchy into the current graphics context. This can be used to get an UIImage very fast.
I implemented a category method on UIView to get the vi
- MySql重置密码
fanxiaolong
MySql重置密码
方法一:
在my.ini的[mysqld]字段加入:
skip-grant-tables
重启mysql服务,这时的mysql不需要密码即可登录数据库
然后进入mysql
mysql>use mysql;
mysql>更新 user set password=password('新密码') WHERE User='root';
mysq
- Ehcache(03)——Ehcache中储存缓存的方式
234390216
ehcacheMemoryStoreDiskStore存储驱除策略
Ehcache中储存缓存的方式
目录
1 堆内存(MemoryStore)
1.1 指定可用内存
1.2 驱除策略
1.3 元素过期
2 &nbs
- spring mvc中的@propertysource
jackyrong
spring mvc
在spring mvc中,在配置文件中的东西,可以在java代码中通过注解进行读取了:
@PropertySource 在spring 3.1中开始引入
比如有配置文件
config.properties
mongodb.url=1.2.3.4
mongodb.db=hello
则代码中
@PropertySource(&
- 重学单例模式
lanqiu17
单例Singleton模式
最近在重新学习设计模式,感觉对模式理解更加深刻。觉得有必要记下来。
第一个学的就是单例模式,单例模式估计是最好理解的模式了。它的作用就是防止外部创建实例,保证只有一个实例。
单例模式的常用实现方式有两种,就人们熟知的饱汉式与饥汉式,具体就不多说了。这里说下其他的实现方式
静态内部类方式:
package test.pattern.singleton.statics;
publ
- .NET开源核心运行时,且行且珍惜
netcome
java.net开源
背景
2014年11月12日,ASP.NET之父、微软云计算与企业级产品工程部执行副总裁Scott Guthrie,在Connect全球开发者在线会议上宣布,微软将开源全部.NET核心运行时,并将.NET 扩展为可在 Linux 和 Mac OS 平台上运行。.NET核心运行时将基于MIT开源许可协议发布,其中将包括执行.NET代码所需的一切项目——CLR、JIT编译器、垃圾收集器(GC)和核心
- 使用oscahe缓存技术减少与数据库的频繁交互
Everyday都不同
Web高并发oscahe缓存
此前一直不知道缓存的具体实现,只知道是把数据存储在内存中,以便下次直接从内存中读取。对于缓存的使用也没有概念,觉得缓存技术是一个比较”神秘陌生“的领域。但最近要用到缓存技术,发现还是很有必要一探究竟的。
缓存技术使用背景:一般来说,对于web项目,如果我们要什么数据直接jdbc查库好了,但是在遇到高并发的情形下,不可能每一次都是去查数据库,因为这样在高并发的情形下显得不太合理——
- Spring+Mybatis 手动控制事务
toknowme
mybatis
@Override
public boolean testDelete(String jobCode) throws Exception {
boolean flag = false;
&nbs
- 菜鸟级的android程序员面试时候需要掌握的知识点
xp9802
android
熟悉Android开发架构和API调用
掌握APP适应不同型号手机屏幕开发技巧
熟悉Android下的数据存储
熟练Android Debug Bridge Tool
熟练Eclipse/ADT及相关工具
熟悉Android框架原理及Activity生命周期
熟练进行Android UI布局
熟练使用SQLite数据库;
熟悉Android下网络通信机制,S
 https://risteys.finregistry.fi/
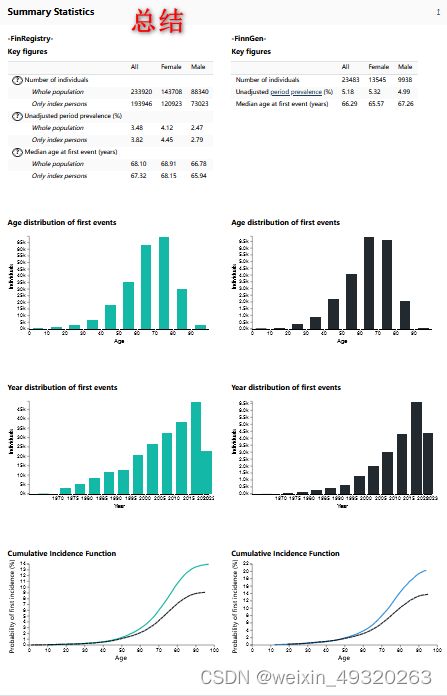
https://risteys.finregistry.fi/ https://finngen.gitbook.io/documentation/data-description#summary-association-statistics
https://finngen.gitbook.io/documentation/data-description#summary-association-statistics