TREC Precision Medicine 使用Terrier进行信息检索
一、实验目标
学会使用一个信息检索系统完成给定的信息检索任务,包括创建索引、选择检索模型并设置参数、评价检索结果等等。
二、实验描述
使用一个信息检索系统,例如Galago、Elastic Search、Terrier、Anserini等,完成TREC 2018 Precision Medicine Track的检索任务,要求至少给出10组不同参数配置或检索方案的结果,并给出每组检索结果的各评价指标的得分,进行分析比较。
三、实验环境
开发环境:Mac OS 10.15.1
软件版本:Java JRE 1.8.0、Python 3.7、Terrier5.0
四、实验过程
4.1 配置Java环境
在oracle官网下载jdk,按提示安装直到安装完成。
Java安装完成后在终端输入java或者java -version查看相关信息,接着在输入echo $JAVA_HOME,此命令将输出Java安装的路径。如果输出为空白,则需要修改zshrc文件,添加exportJAVA_HOME=$(/usr/libexec/java_home)保存退出即可。
![]()
![]()
4.2 安装Terrier
在进行具体的实验操作之前,本小组调研了多个信息检索的开源工具,包括 Lucene、Elasticsearch和Terrier。相比之下,Terrier 更适合用于完成本次任务,其专门为 TREC 比赛定制了接口,而且 Lucene 的评分函数比较简单,适合于工程开发,而 Terrier 对语言模型做了很多优化,更适合科研类任务。根据以往经验,用 terrier 实现要比用 Lucene 实现效果普遍更好。
可以从官网上或者 Github 下载 terrier-project-5.0-bin.tar.gz,下载完后解压即可。解压完成后目录如下图所示:
其中,
bin: 存放运行脚本,在 linux 系统下用.sh,在 windows 下用相应的.bat
doc:帮助说明文档集
etc: 存放建立索引和检索时的配置文件,如 collection.spec, terrier.properties
var: 检索结果的存放位置,存放 index, result,可以通过修改 etc 文件夹下的
terrier.properties 配置文件,定位到其他文件夹
share:存放 stopword-list.txt 等文件
src: 搜索引擎源代码
4.3 数据预处理
在利用 Terrier 进行检索实验之前,首先对给定数据集进行预处理,既是为了满足Terrier 的数据格式要求,也是为了提升查询检索的效果。
4.3.1 处理科学文摘的数据
原始数据集是从Terrier官网下载的xml类型数据,每个数据中包含多个文献,并且有很多对查询影响较低的信息,所以将原数据中的文章编号、文章题目和文章摘要提取出来,生成新的检索数据,处理后的数据格式如下:
4.3.2 处理临床试验的数据
原始数据集是从Terrier官网下载的xml类型数据,每个数据中标签很多,包含很多不必要的信息,所以对其中有价值的标签进行提取。提取的标签主要有索引要求中涉及的,例如brief_title、official_title、brief_summary、detailed_description、MeSH Terms 和 criteria 等,以及查询文件中涉及的标签,例如minimum_age、maximum_age、gender。以 NCT00000102为例,处理后的格式如下:
4.3.3 格式化查询题目
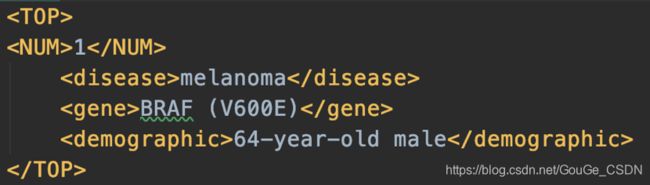
评测的查询输入是半结构化文本,描述了患者的癌症类型、相关的基因变异、年龄性别以及其他可能相关的因素,文件格式如下。需要说明的是,原始查询文件的根标签是 topic和 number,改成 TOP 和 NUM 之后,便于之后处理。
4.4 收集数据文档并建立索引
4.4.1 收集数据文档
收集需要建索引的数据并设置数据的路径。收集完成后,将会在terrier的etc 目录下创建一个collection.spec 文件,文件中包含将要建立索引的数据信息。执行命令格式如下:
bin/trec_setup.sh
即在bin/trec_setup.sh命令后输入需要建立索引数据所在的绝对路径。
4.4.2 建立索引
数据收集完成之后即可建立索引,命令格式如下:
bin/terrier batchindexing
创建完成之后,将会在Terrier中var目录下的index中生成索引文件。通过下面的命令可以查看建立索引的信息:
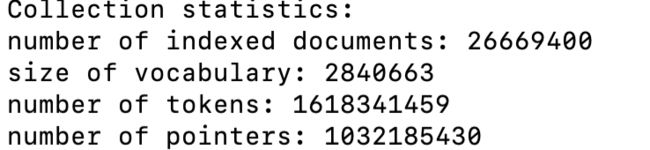
bin/terrier indexstats
以建立科学文摘数据的索引为例,结果如下图所示:
4.5 查询检索
检索命令含有多个参数,可以在etc/terrier.properties文件里预先设好,也可以一个一个在命令行里指定。
基本命令为
bin/terrier batchretrieve
首先要设置查询的位置 通过“trec.topics”属性或者“-t”来指定查询位置,建议使用绝对路径。
其次要设置查询所使用的模型,通过“trec.model”属性或者“-w”来指定查询模型,包括BB2,BM25,LGD等等。Terrier默认的检索模型是DPH。
设置检索模型之后可以通过“-c”等参数设置模型参数(有些模型没有参数)。
以用BB2模型检索科学文摘为例,命令如下图所示:
![]()
检索完成之后,将在var目录下的results文件夹中生成.res文件,格式如下图所示:
4.6 相关反馈
加入伪相关反馈,其命令如下图。相较于上一步的检索命令,增加了 -q 和-Dqe.feedback.filename 参数,用于指定用哪一个模型的检索结果作为反馈的输入。下图中是以IFB2模型的结果作为反馈输入。
本组利用 Terrier 开源工具中已经实现的 Bo1、Bo2、KL 等伪相关反馈模型进行实验。可以在配置文件terrier.properties 中添加相关的配置项,或者是在每一行命令下添加上述参数。
4.7 结果评估
以评估科学文摘的检索结果为例,利用给定的数据qrels-treceval-abstracts.2018.txt,对结果进行评估,命令如下图所示:
该命令执行后会生成一个模型对应的 eval 文件。该文件中包含了 MAP、Bpref、Recall、P_5、P_10 等度量指标值,下图是 BB2.eval 文件内容。
五、实验结果
5.1 模型选择
Terrier 开源工具中集成了许多信息检索模型,可以直接进行接口调用。实验过程中,本小组选择了 13 种模型进行测试,下表展示了不同模型的测试指标值。没有添加相关反馈的情况下,MAP 的最优结果为 0.2050,由IFB2 模型得到;P@5 的最优结果为 0.5640,由 In_expB2和IFB2 模型得到;P@10 的最优结果为 0.5140,由In_expB2 模型得到。经过实验分析,发现各个模型参数的变动对最终结果影响较小,所以在实验中大多数模型使用了默认参数。
| Model |
MAP |
P@5 |
P@10 |
| BM25 |
0.1453 |
0.4800 |
0.4560 |
| BB2 |
0.2024 |
0.5520 |
0.5100 |
| DLH13 |
0.0764 |
0.2760 |
0.2640 |
| In_expB2 |
0.2040 |
0.5640 |
0.5140 |
| DPH |
0.0846 |
0.3120 |
0.3260 |
| Hiemstra_LM |
0.1076 |
0.3320 |
0.3040 |
| IFB2 |
0.2050 |
0.5640 |
0.5100 |
| LGD |
0.0745 |
0.2480 |
0.2360 |
| InL2 |
0.1405 |
0.4200 |
0.3940 |
| PL2 |
0.1228 |
0.3540 |
0.3440 |
| TF_IDF |
0.1402 |
0.4400 |
0.4020 |
| LemurTF_IDF |
0.2030 |
0.5520 |
0.4800 |
| DFR_BM25 |
0.1477 |
0.4560 |
0.4060 |
5.2 相关反馈
选出 4 个结果较好的模型添加相关反馈进行下一步测试,分别是In_expB2、BB2、LemurTF_IDF和 IFB2,也使用了一些初始结果较差的模型进行相关反馈实验。由于组合较多,下表只展示部分结果。
| Model |
MAP |
P@5 |
P@10 |
| IFB2+BB2 |
0.2134 |
0.5680 |
0.5280 |
| BB2+IFB2 |
0.2116 |
0.5680 |
0.5260 |
| In_expB2+BB2 |
0.2129 |
0.5680 |
0.5280 |
| LemurTF+In_expB2 |
0.1934 |
0.5320 |
0.4800 |
在使用伪相关反馈的情况下,MAP 的最优结果为 0.2134,由 IFB2+BB2 模型得到,相较于未添加相关反馈的结果提升了4%;P@5 的最优结果为 0.5680,P@10的最优结果为0.5280,均无明显提升。
经过实验分析,发现利用BB2的查询结果作为反馈,再利用IFB2模型进行查询检索,得出的效果最优。
六、实验基本原理
6.1 评价指标
(1)召回率(Recall): RR/(RR + NR),返回的相关结果数占实际相关结果总数的比率,也称为查全率,R∈[0,1]。
(2) 正确率(Precision): RR/(RR + RN),返回的结果中真正相关结果的比率,也称为查准率, P∈ [0,1]。
(3)Bpref:基本的思想:在相关性判断(Relevance Judgment) 不完全的情况下,计算在进行了相关性判断的文档集合中,在判断到相关文档前,需要判断的不相关文档的篇数。
(4)NDCG:每个文档不仅仅只有相关和不相关两种情况,而是有相关度级别,比如0,1,2,3。我们可以假设,对于返回结果,相关度级别越高的结果越多越好,相关度级别越高的结果越靠前越好。
(5)MAP:平均正确率(Average Precision, AP):对不同召回率点上的正确率进行平均。
(6)P@K:计算前 K 个位置的平均正确率。
6.2 模型介绍
(1)BB2(DFR):用于随机性的Bose-Einstein模型,用于首次归一化的两个伯努利过程的比率,用于术语频率归一化的归一化2。
(2)BM25:BM25概率模型。。
(3)DLH(DFR):DLH超几何DFR模型(无参数)。
(4)DLH13(DFR):DLH的改进版本(无参数)。
(5)DPH(DFR):使用Popper的归一化(无参数)的另一种超几何DFR模型。
(6)Hiemstra_LM:Hiemstra的语言模型。
(7)IFB2(DFR):用于随机性的逆项频率模型,用于首次归一化的两个伯努利过程的比率,用于项频率归一化的归一化2。
(8)In_expB2(DFR):用于随机性的反向期望文档频率模型,用于首次归一化的两个伯努利过程的比率,用于术语频率归一化的归一化2。
(9)InL2(DFR):用于随机性的逆文档频率模型,用于第一归一化的拉普拉斯继承,以及用于术语频率归一化的归一化2。
(10)LemurTF_IDF:Lemur的tf * idf加权功能版本。
(11)LGD(DFR):对数逻辑DFR模型。
(12)PL2(DFR):用于随机性的Poisson估计,用于第一次归一化的Laplace继承和用于项频率归一化的归一化2。
(13)TF_IDF:tf * idf加权函数,其中tf由罗伯逊的tf给出,idf由标准Sparck Jones的idf给出。
参考:
TREC Precision Medicine / Clinical Decision Support Track
Terrier官网