病毒感染检测(数据结构串,c语言版)
病毒感染检测
- 一、实验题目
-
- 1.案例分析
- 2.案例实现
- 3.算法步骤
- 4.算法描述
- 二、工具环境
- 三、实验代码
一、实验题目
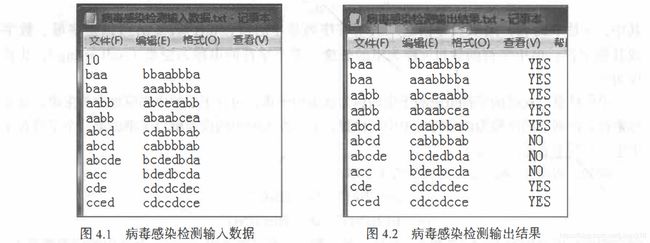
医学研究者最近发现了某些新病毒,通过对这些病毒的分析,得知它们的 DNA 序列都是环状的。现在研究者巳收集了大量的病毒 DNA 和人的 DNA 数据,想快速检测出这些人是否感染了相应的病毒。为了方便研究,研究者将人的 DNA 和病毒 DNA 均表示成由一些字母组成的字符串序列,然后检测某种病毒 DNA 序列是否在患者的 DNA 序列中出现过,如果出现过,则此人感染了该病毒,否则没有感染。 例如, 假设病毒的 DNA 序列为 baa, 患者 1 的 DNA 序列为 aaabbba,则感染, 患者 2 的 DNA 序列为 babbba, 则未感染。(注意,人的 DNA 序列是线性的, 而病毒的DNA 序列是环状的)图 4.1 数据对应的输出结果如图 4.2 所示。
1.案例分析
因为患者的 DNA 和病毒 DNA 均是由一些字母组成的字符串序列, 要检测某种病毒 DNA 序列是否在患者的 DNA 序列中出现过 , 实际上就是字符串的模式匹配问题。可以利用 BF 算法,也可以利用更高效的KMP算法。但与一般的模式匹配问题不同的是,此案例中病毒的 DNA 序列是环状的, 这样需要对传统的 BF 算法或KMP算法进行改进。
下面给出利用 BF 算法实现检测的方案。
2.案例实现
对于每一个待检测的任务, 假设病毒 DNA 序列的长度是 m, 因为病毒 DNA 序列是环状的 ,为了线性取到每个可行的长度为 m 的模式串,可将存储病毒DNA序列的字符串长度扩大为 2m,将病毒DNA序列连续存储两次。然后循环m次,依次取得每个长度为m的环状字符串,将此字符串作为模式串,将人的DNA序列作为主串,调用BF算法进行模式匹配。 只要匹配成功,即可中止循环,表明该人感染了对应的病毒;否则,循环m次结束循环时, 可通过BF算法的返回值判断该人是否感染了对应的病毒。
3.算法步骤
1.从文件中读取待检测的任务数 num。
2.根据 num个数依次检测每对病毒DNA和人的DNA是否匹配,循环 num次,执行以下操作:
• 从文件中分别读取一对病毒DNA序列和人的DNA序列;
• 设置一个标志性变量flag, 用来标识是否匹配成功,初始为0, 表示未匹配;
• 病毒DNA序列的长度是 m, 将存储病毒DNA序列的字符串长度扩大为 2m, 将病毒DNA序列连续存储两次;
• 循环m次,重复执行以下操作:
► 依次取得每个长度为m的病毒DNA环状字符串;
► 将此字符串作为模式串,将人的DNA序列作为主串, 调用BF算法进行模式匹配,将匹配结果返回赋值给flag;
► 若flag非0, 表示匹配成功, 中止循环,表明该人感染了对应的病毒。
• 退出循环时, 判断flag的值,若flag非0, 输出 “YES” , 否则,输出 “NO”。
4.算法描述
void Virus detection ()
{//利用 BF 算法实现病毒检测
ifstream inFile("病毒感染检测输入数据. txt");
ofstream outFile("病毒感染检测输出结果. txt");
inFile>>num; //读取待检测的任务数
while(num--) //依次检测每对病毒 DNA 和人的 DNA 是否匹配
{
inFile>>Virus.ch+1; //读取病毒 DNA 序列, 字符串从下标 1 开始存放
inFile>>Person.ch+1; //读取人的 DNA 序列
Vir=Virus.ch; //将病毒 DNA 临时暂存在Vir中,以备输出
flag=0; //用来标识是否匹配,初始为0, 匹配后为非0
m=Virus.length; //病毒 DNA 序列的长度是 m
for(i=m+1, j=l; j<=m; j++)
Virus.ch[i++]=Virus.ch[j]; //将病毒字符串的长度扩大2倍
Virus.ch[2*m+1]='\0'; //添加结束符号
for(i=0;i<m;i++) //依次取得每个长度为m 的病毒 DNA 环状字符串 temp
{
for (j=1; j<=m; j ++) temp.ch[j]=Virus.ch[i+j];
temp.ch[m+1]='\0'; //添加结束符号
flag=Index_BF (Person, temp, 1); //模式匹配
if(flag) break; //匹配即可退出循环
}// for
if (flag) outFile<<Vir+l<<" "<<Person.ch+1<<" "<<"YES"<<endl;
else outFile<<Vir+l<<" "<<Person.ch+1<<" "<<"NO"<<endl;
}//while
}
二、工具环境
Window10操作系统,Microsoft Visual C++2010学习版 集成开发环境,C语言
三、实验代码
#include