AAU-net: 用于超声图像中乳腺病变分割的自适应注意力U-Net
AAU-net
- 期刊分析
-
- 摘要
- 贡献
- 方法
-
- 整体框架
- 1.Hybrid Adaptive Attention Module
- 2.Channel Self-Attention Block
- 3.Spatial Self-Attention Block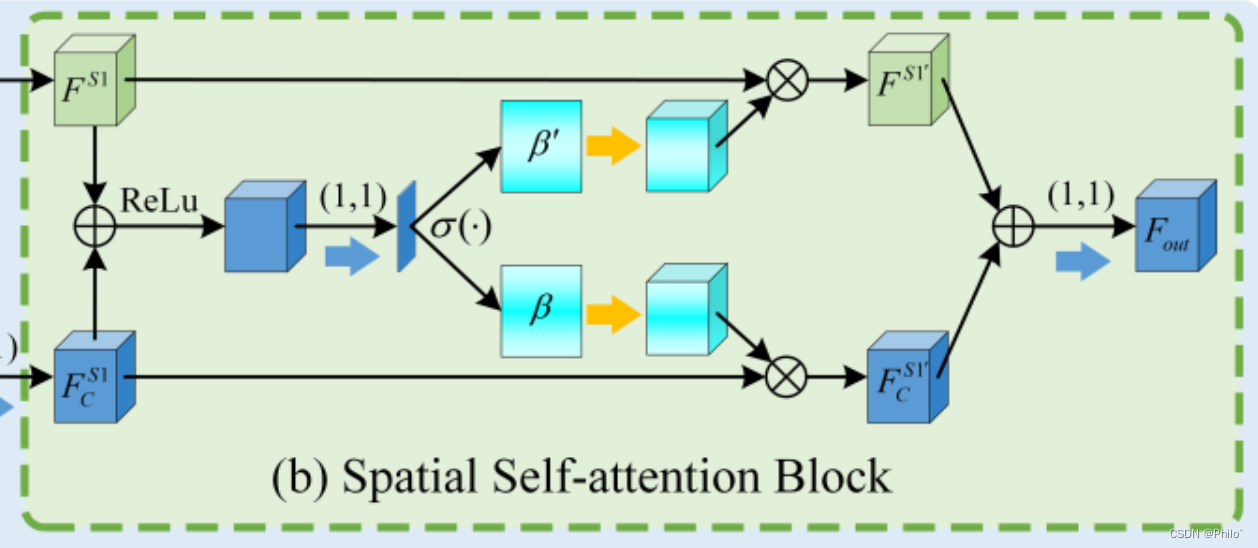
- 实验
-
- 1.消融实验
-
- 1.1 Architecture Ablation
- 1.2 Parameter Ablation
- 2.对比实验
- 3.鲁棒性分析
-
- 3.1 Robustness on Benign and Malignant Lesions
- 3.2 External Validation
- 3.3 Comparison on BUSI with Normal Images
- 3.4 Comparison with different Attention-Based Methods
- 可借鉴参考
期刊分析
期刊名: TRANSACTIONS ON MEDICAL IMAGING
期刊信息: IF: 10.6; JCR: Q1; 中科院一区Top
投稿周期:
本篇:未知; Publish: 2023.5
网站统计:录用比例:很难; 审稿周期:约5.4月
其余信息: LetPub
该论文有代码,Tensorflow版本
摘要
已经提出了各种深度学习方法来从超声图像中分割乳腺病变。然而,相似的强度分布、可变的肿瘤形态和模糊的边界给乳腺病灶分割带来了挑战,特别是对于形状不规则的恶性肿瘤。考虑到超声图像的复杂性,我们开发了自适应注意力U-net(AAU-net)来自动稳定地从超声图像中分割乳腺病变。具体来说,我们引入了一种混合自适应注意模块(HAAM),它主要由通道自注意块和空间自注意块组成,以取代传统的卷积操作。与传统的卷积操作相比,混合自适应注意模块的设计可以帮助我们在不同感受野下捕获更多特征。与现有的注意力机制不同,HAAM模块可以引导网络自适应地选择通道和空间维度上更鲁棒的表示,以应对更复杂的乳腺病变分割。在三个公共乳腺超声数据集上使用几种最先进的深度学习分割方法进行的广泛实验表明,我们的方法在乳腺病变分割方面具有更好的性能。此外,鲁棒性分析和外部实验表明,我们提出的 AAU-net 在乳腺病变分割方面具有更好的泛化性能。而且HAAM模块可以灵活应用到现有的网络框架中。源代码可在 https://github.com/CGPxy/AAU-net 上获取
贡献
- 设计了一种新颖的混合自适应注意模块,它可以从通道和空间维度自适应地选择不同尺度的感受野。
- 开发了一种新颖的自适应注意力 U 网,用于从超声图像中分割乳腺病变。该网络可以通过学习 BUS 图像的通用表示来改进乳腺病变分割。
- 对三个公共 BUS 数据集的大量实验表明,我们的方法持续提高了乳腺病变的分割精度,优于强大的基线和最先进的医学图像分割方法。
方法
整体框架
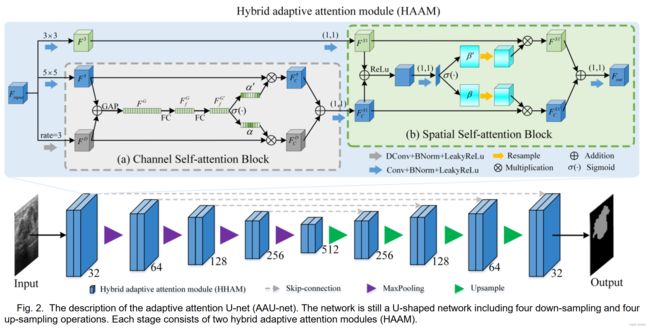
这个网络写的言简意赅。重点是将UNet中的基础卷积换成混合自适应卷积模块,和之前解读的SegNetr很像。同时整个HAAM很像一个混合版的SK-Block(可以在另一篇写的注意力模块博文中找到)
1.Hybrid Adaptive Attention Module

1. 是三个特征,两个阶段的混合;
2. 三个特征分别使用普通的3×3卷积、5×5卷积、3×3空洞卷积(空洞率为3);
3. 三个特征中两个先使用通道注意力模块,然后将第三个结合进来传入到空间注意力模块;

个人认为这个图是有问题的,其中5×5卷积核3×3的空洞卷积示意图画错了
2.Channel Self-Attention Block

就是一个简化版的SK-Net,将分支数设为2,其余的变化很少
3.Spatial Self-Attention Block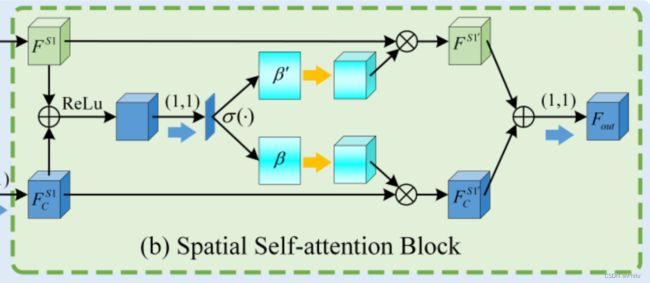
也是一个简化版的SK-Net,将中间的特征使用1×1卷积和softmax得到空间权重进行特征增强
总体感觉创新点不足,不是很突出;但是实验做的是真的多,与我自己的实验结果来看,结果可信度很高,但应该是没有达到SOTA的。
实验
损失函数:BCE;公开的三个数据集:BUSI、BUS、STU(具体的可在文章中找到下载方式);
1.消融实验
1.1 Architecture Ablation
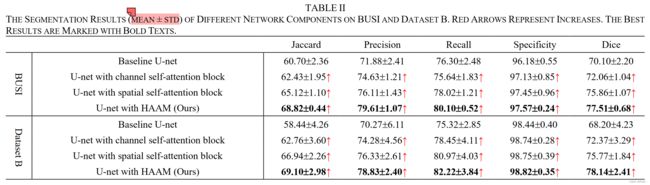
1. 相较于基础模型,空间注意力模块的提升效果比通道注意力的好
2. 终于知道他们±符号的具体定义了,以下给出一个例子进行演示



1.2 Parameter Ablation
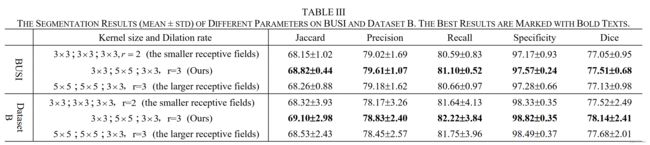
就是将卷积核大小改变,空洞卷积率改变。结果表明,作者提出的卷积方案最优,但是我们可以看出啦,影响的效果不大(尤其是在BUSI数据集上)
2.对比实验



1. 这个结果分布和我当时做出了的结果分布很像,但是只是对比CNN -Base模型,没有与Combined-VIT模型对比,其实很难说达到了SOTA效果。
2. 自己也做了这两个数据集,效果是比较好的。这里要吐槽一下,同样的数据集,我的最好结果竟然是别人的benchmark,,而且没给出数据集划分和代码。
3. 可视化结果中,ROC和P-R曲线是我需要学习的内容,以后可以放在论文中
4. 可视化分割结果图中,对比结果很好,但是其本身达到的精度有待提升
3.鲁棒性分析
3.1 Robustness on Benign and Malignant Lesions
1. 将乳腺良恶性病例分开进行分割测试,这样的操作可以清晰的看出作者提出的模型对哪一类的病例提升效果明显,这也是后期自己实验的方向
2. ROC曲线,得学一下咋画的
3.2 External Validation

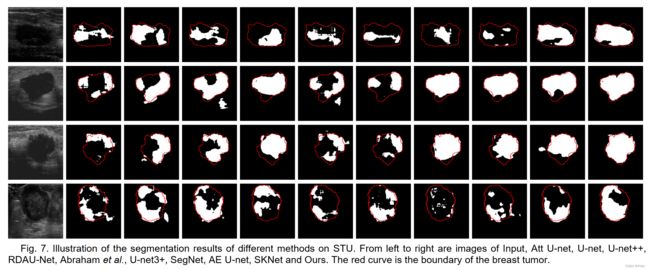
将在BUS数据集(Dataset B)上训练的模型用来分割外部数据STU得到的结果,结合表IV可以看出,其分割效果比在DatasetB的效果好
3.3 Comparison on BUSI with Normal Images
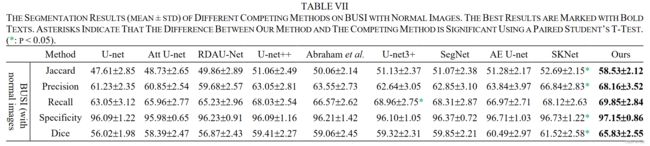
BUSI数据集本身是含有266例正常乳腺病例(及GT图像无mask),大多数论文都是在数据集引入的时候排除掉的,没想到作者在这竟然还放进去讨论了
3.4 Comparison with different Attention-Based Methods

主要是使用主流的注意力模块替换作者提出的HAAM模块
结果显示,在DatasetB上的效果要比在BUSI上的效果突出很多
可借鉴参考
-
定量实验结果呈现mean±std
-
P值检验的具体流程
from scipy import stats
# UNet的Dice结果
unet_dice = [0.83, 0.835, 0.850]
# Att-UNet的Dice结果
att_unet_dice = [0.886, 0.899, 0.865]
# 执行独立样本t检验
t_stat, p_value = stats.ttest_ind(unet_dice, att_unet_dice)
# 输出p值
print("P值为:", p_value)
-
阅读 2022
R. Huang, M. Lin, H. Dou, Z. Lin, Q. Ying, X. Jia, W. Xu, Z. Mei, X. Yang, and Y. Dong, “Boundary-rendering Network for Breast Lesion Segmentation in Ultrasound Images,” Med. Image Anal., p. 102478, 2022. -
阅读MIA 2021
C. Xue, L. Zhu, H. Fu, X. Hu, X. Li, H. Zhang, and P. Heng, “Global guidance network for breast lesion segmentation in ultrasound images,” Med. Image Anal., vol. 70, p. 101989, 2021.