ComplexHeatmap热图专栏 | 1.1 基础热图绘制
1 写在前面
最近在作图,一直在寻找《小杜的生信笔记》前期发表的代码。众所周知,小杜的教程基本都是平时自己用到的绘图教程,也是自己一个分享和总结。
自己在后期作图的时候,也会去寻找自己前期的教程作为基础,进行不断的修改。没错,自己的教程,在后期作图的时候也会不断的修改。

2 热图
热图作为组学分析中最最最常见的图形之一。我们前提也推出好几个绘制热图的教程,如ComplexHeatmap绘制Pathway热图、热图绘制篇 | 高颜值热图总汇、如何绘制带有“正负号”标记的热图?、Pheatmap包绘制热图教程、热图添加指定的基因标记(代码重现)、ComplexHeatmap热图绘制|指定基因在热图中标记。那么我们也推出相关ComplexHeatmap包绘制热图的教程(其实也不算是教程,作者已经给出详细的帮助文档),也算是再次系统性的学习ComplexHeatmap包绘制热图。
在此也感谢ComplexHeatmap包开发作者Zuguang Gu。
Citation:
- Zuguang Gu, et al.,
Complex heatmaps reveal patterns and correlations in multidimensional genomic data, Bioinformatics, 2016. - Zuguang Gu.
Complex Heatmap Visualization, iMeta, 2022.
本教程详细内容可以点击
安装
library(devtools)
install_github("jokergoo/ComplexHeatmap")
1.1 热图的设计
单个热图由热图主体和热图组件组成。热图主体可以按行和列进行分割。热图组件包括标题、树状图、行/列名称/标签和热图注释,它们被放置在堆主体的四个侧面。根据所述热图主体对所述热图组件进行重新排序或拆分。
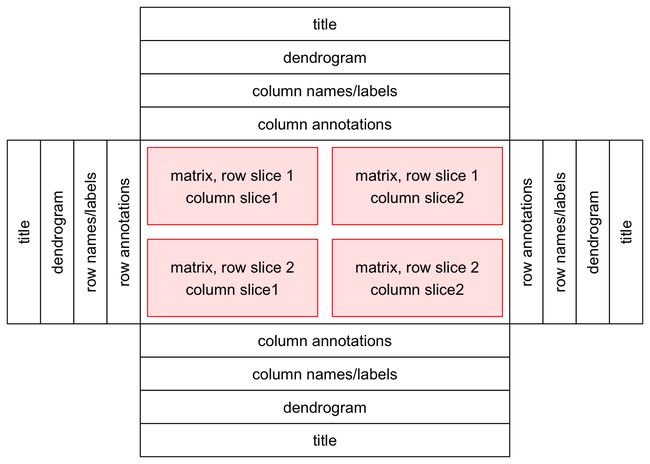
2 A Single Heatmap
单个热图是数据可视化最常用的方法。尽管 ComplexHeat map 包的“闪光点”在于它可以并行地可视化一个热图列表,但是,作为热图列表的基本单元,良好地配置单个热图仍然非常重要。
随机生成一个数据矩阵:
set.seed(123)
nr1 = 4; nr2 = 8; nr3 = 6; nr = nr1 + nr2 + nr3
nc1 = 6; nc2 = 8; nc3 = 10; nc = nc1 + nc2 + nc3
mat = cbind(rbind(matrix(rnorm(nr1*nc1, mean = 1, sd = 0.5), nr = nr1),
matrix(rnorm(nr2*nc1, mean = 0, sd = 0.5), nr = nr2),
matrix(rnorm(nr3*nc1, mean = 0, sd = 0.5), nr = nr3)),
rbind(matrix(rnorm(nr1*nc2, mean = 0, sd = 0.5), nr = nr1),
matrix(rnorm(nr2*nc2, mean = 1, sd = 0.5), nr = nr2),
matrix(rnorm(nr3*nc2, mean = 0, sd = 0.5), nr = nr3)),
rbind(matrix(rnorm(nr1*nc3, mean = 0.5, sd = 0.5), nr = nr1),
matrix(rnorm(nr2*nc3, mean = 0.5, sd = 0.5), nr = nr2),
matrix(rnorm(nr3*nc3, mean = 1, sd = 0.5), nr = nr3))
)
mat = mat[sample(nr, nr), sample(nc, nc)] # random shuffle rows and columns
rownames(mat) = paste0("row", seq_len(nr))
colnames(mat) = paste0("column", seq_len(nc))
head(mat)
> head(mat)
column1 column2 column3 column4 column5 column6 column7 column8 column9 column10 column11 column12 column13
row1 0.90474160 -0.35229823 0.5016096 1.26769942 0.8251229 0.16215217 -0.2869867 0.68032622 -0.1629658 0.8254537 0.7821773 -0.49625358 -0.0895258
row2 0.90882972 0.79157121 1.0726316 0.01299521 0.1391978 0.46833693 1.2814948 0.38998256 -0.3473535 1.3508922 1.1183375 2.05005447 1.3770269
row3 0.28074668 0.02987497 0.7052595 1.21514235 0.1747267 0.20949120 -0.6423579 -0.31395304 0.2175907 -0.2973086 0.4322058 -0.25803192 -0.5686518
row4 0.02729558 0.75810969 0.5333504 -0.49637424 -0.5261114 0.56724357 0.8127096 -0.01427338 1.0844780 0.2426662 0.8783874 1.38452112 0.8376570
row5 -0.32552445 1.03264652 1.1249573 0.66695147 0.4490584 1.04236865 2.6205200 0.75823530 -0.2333277 1.3439584 0.8517620 0.85980233 1.9986067
row6 0.58403269 -0.47373731 0.5452483 0.86824798 -0.1976372 -0.03565404 -0.3203530 1.05534136 0.7771690 0.4594983 0.2550648 -0.02778098 -0.2112484
我们可以替换成绘制热图的数据即可。
下面的命令包含 Heatmap()函数的最小参数,该函数将矩阵可视化为具有默认设置的热图。与其他热图工具非常相似,它绘制树状图、行/列名称和热图图例。默认的颜色模式是“蓝-白-红”,它映射到矩阵中的最小均值-最大值。图例的标题使用内部索引号分配。
Heatmap(mat)
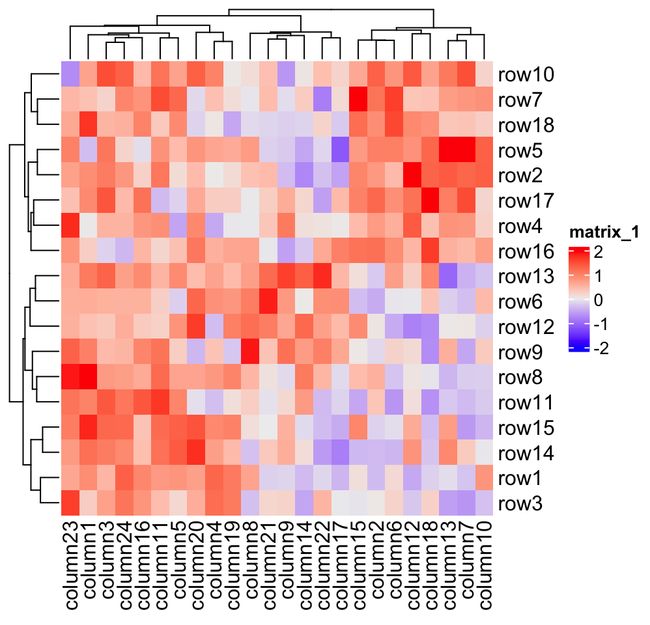
默认情况下,图例的标题取自热图的“名称”。每个热图都有一个名字,就像热图的唯一标识符一样,。在后面的章节中,你会发现热图名称是用来设置“主热图”,并用于装饰热图。如果未分配该名称,则将一个内部名称以matrix_%d的形式分配给热图。在本章的下面的例子中,我们给热图命名垫(在下一个图中你会看到图例标题的变化)。
2.1 更改颜色
用circlize::ColorRamp2()函数在Heatmap()中生成颜色映射函数。ColorRamp2()的两个参数是中断值的向量和相应颜色的向量。ColorRamp2()通过LAB颜色空间在每个区间内线性插值颜色。此外,使用color Ramp2()还可以帮助生成带有适当刻度标记的图例。
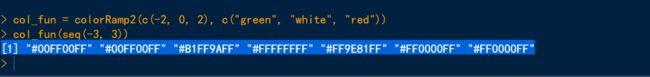
在下面的例子中,-2和2之间的值被线性插值以获得相应的颜色,大于2的值都映射为红色,小于 -2的值都映射为绿色。
library(circlize)
col_fun = colorRamp2(c(-2, 0, 2), c("green", "white", "red"))
col_fun(seq(-3, 3))
[1] "#00FF00FF" "#00FF00FF" "#B1FF9AFF" "#FFFFFFFF" "#FF9E81FF" "#FF0000FF" "#FF0000FF"
##'@2.1 更改颜色
library(circlize)
col_fun = colorRamp2(c(-2, 0, 2), c("green", "white", "red"))
col_fun(seq(-3, 3))
## 绘图
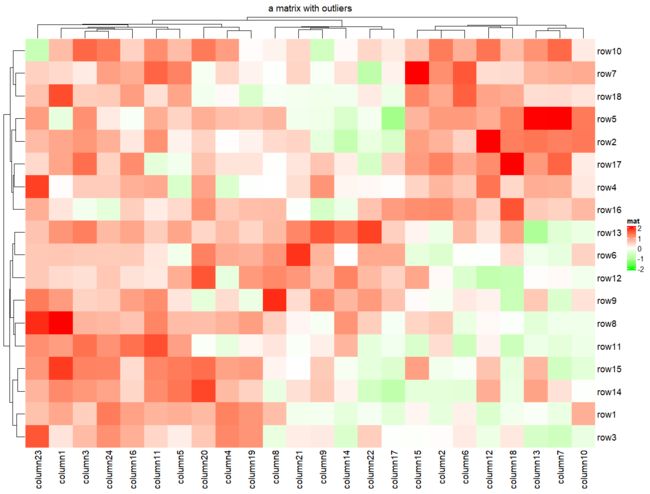
Heatmap(mat, name = "mat", col = col_fun)
2.2 热图标题
Heatmap(mat, name = "mat", col = col_fun,
## 标题的修改
column_title = "a matrix with outliers")
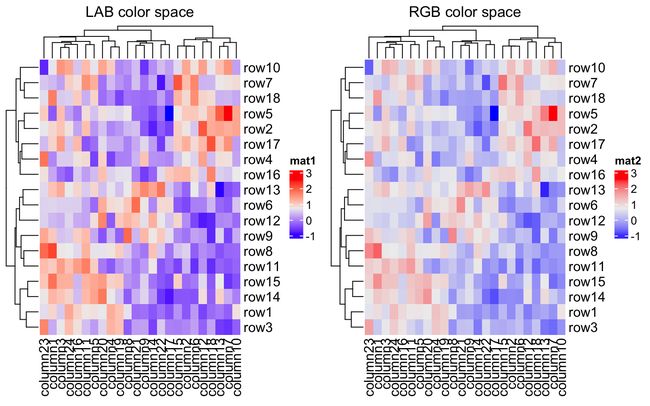
2.3 不使用colorRamp2()更改颜色
颜色设置:
f1 = colorRamp2(seq(min(mat), max(mat), length = 3), c("blue", "#EEEEEE", "red"))
f2 = colorRamp2(seq(min(mat), max(mat), length = 3), c("blue", "#EEEEEE", "red"), space = "RGB")
Heatmap(mat, name = "mat1", col = f1, column_title = "LAB color space")
Heatmap(mat, name = "mat2", col = f2, column_title = "RGB color space")
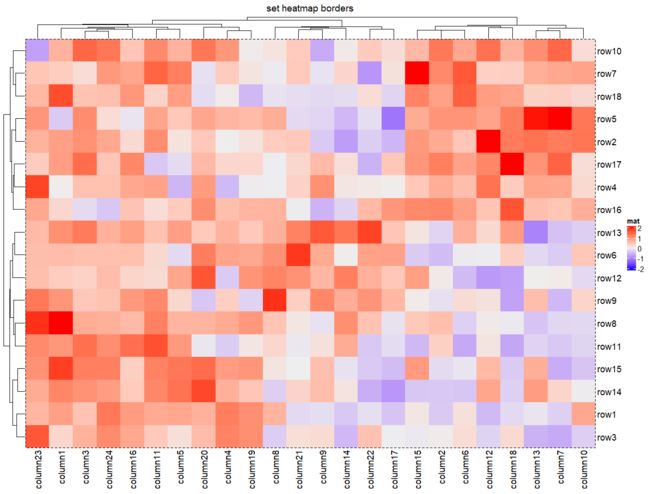
2.4 添加热图边框颜色
2.4.1 设置热图主体全局边界
热图边框的颜色可以通过border/border_gp和rect_gp参数设置。border/border_gp控制热图主体的全局边界,rect_gp控制热图中网格/单元格的边界。
##'@2.4 添加热图边框颜色
Heatmap(mat, name = "mat",
##设置颜色
border_gp = gpar(col = "black", ## 设置颜色
lty = 2), ## lty = 2设置虚线距离
column_title = "set heatmap borders")
2.4.2 设置热图网格/单元格的边界
Heatmap(mat, name = "mat", rect_gp = gpar(col = "white", ## 设置颜色
lwd = 2),
column_title = "set cell borders")
往期文章:
1. 复现SCI文章系列专栏
![]()
3. 最全WGCNA教程(替换数据即可出全部结果与图形)
-
WGCNA分析 | 全流程分析代码 | 代码一
-
WGCNA分析 | 全流程分析代码 | 代码二
-
WGCNA分析 | 全流程代码分享 | 代码三
4. 精美图形绘制教程
- 精美图形绘制教程
5. 转录组分析教程
转录组上游分析教程[零基础]
小杜的生信筆記,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!