- Elasticsearch 聚合查询源码解读与架构方法论
北漂老男人
Elasticsearchelasticsearch架构大数据搜索引擎全文检索
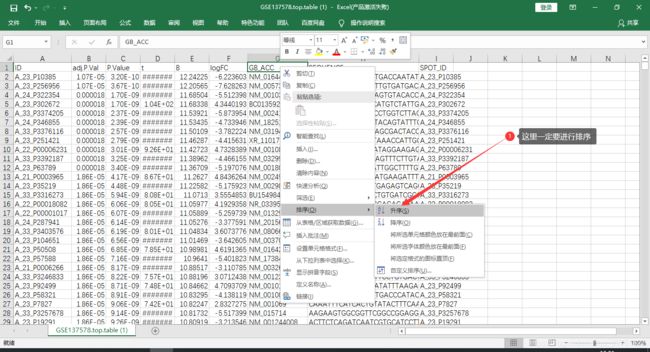
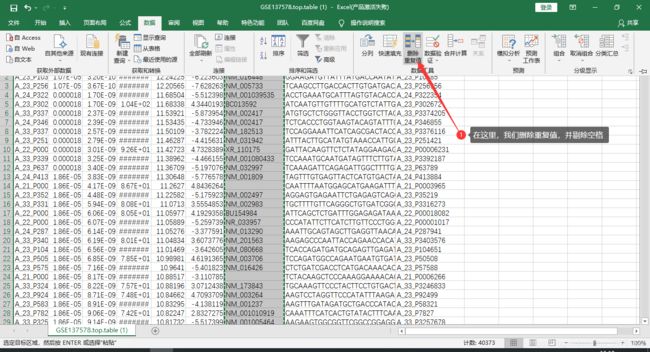
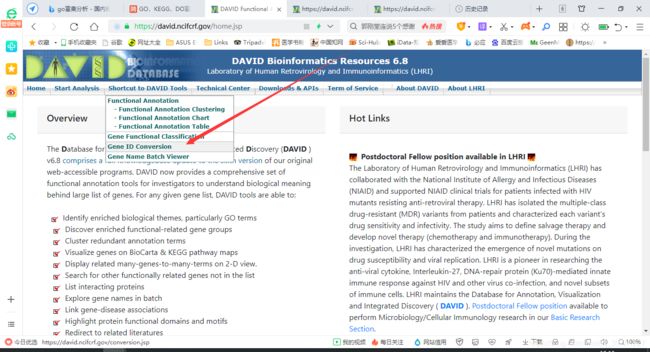
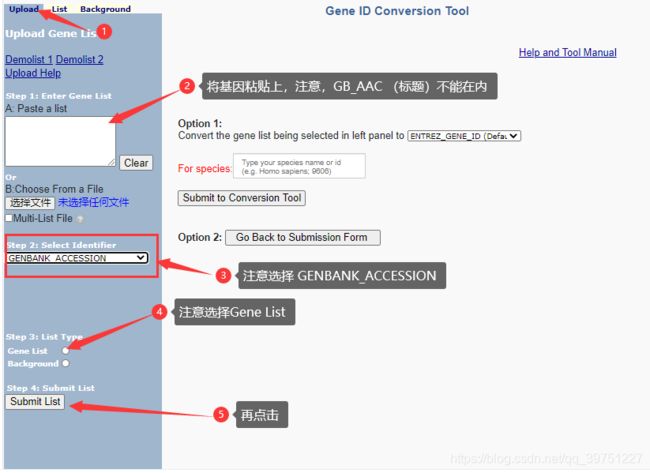
Elasticsearch聚合查询源码解读与架构方法论01.引言Elasticsearch的聚合查询(Aggregation)是大规模分布式数据分析的核心能力。理解其源码结构与设计方法论,不仅有助于高效使用聚合,也能为自定义扩展、性能优化、集群运维等提供理论与实践基础。本文将从源码结构、核心模块、关键实现、行级注释与方法论出发,系统剖析聚合查询的底层原理。02.源码结构与核心模块2.1聚合相关源码
- 【Redis(8)】Spring Boot整合Redis和Guava,解决缓存穿透、缓存击穿、缓存雪崩等缓存问题
在缓存技术的挑战及设计方案我们介绍了使用缓存技术可能会遇到的一些问题,那么如何解决这些问题呢?在构建缓存系统时,SpringBoot和Redis的结合提供了强大的支持,而Guava的LoadingCache则为缓存管理带来了便捷的解决方案。下面我将介绍如何通过整合SpringBoot、Redis和Guava来实现一个解决缓存穿透、缓存击穿、缓存雪崩、缓存污染和缓存数据一致性问题的缓存方案。一、整合
- 零基础学习性能测试第一章:核心性能指标-吞吐量QPS/TPS
试着
性能测试学习性能测试零基础性能指标QPSTPS
目录零基础学习性能测试:第一章-核心性能指标:吞吐量(QPS/TPS)一、吞吐量核心概念解析1.吞吐量定义与分类2.核心区别与关系二、吞吐量关键价值与工作应用1.吞吐量的业务意义2.实际工作场景应用三、吞吐量测试实战指南1.测试工具选择2.JMeter吞吐量测试全流程3.关键配置参数四、吞吐量瓶颈分析与优化1.瓶颈定位四步法2.常见瓶颈及解决方案3.优化案例:电商系统吞吐量提升五、工作应用模板与工
- 零基础学习性能测试第一章:核心性能指标-响应时间
试着
性能测试学习性能测试零基础性能指标响应时间
目录核心学习理念:聚焦实战、理解本质、快速应用**第一阶段:理解响应时间的本质(1-2小时)第二阶段:学习如何测量响应时间(动手实践,2-4小时)第三阶段:将响应时间应用到实际工作(核心目标)第四阶段:快速应用的关键技巧与注意事项总结与行动清单(今天就能开始做!)零基础学习性能测试的核心指标——响应时间,并能快速应用到工作中,这个目标很明确!下面我将为你设计一个结构化的学习路径,从概念到实践,让你
- 零基础学习性能测试第一章:性能需求分析
试着
性能测试学习数据库服务器性能测试零基础需求分析
目录**核心学习理念****模块1:理解性能需求分析的价值(1小时)****1.1为什么必须做需求分析?****1.2性能需求四要素**(附企业级模板)**模块2:四步挖掘性能需求(实战核心)****步骤1:识别关键业务场景(2小时)****步骤2:量化业务负载(3小时)****步骤3:定义性能指标(2小时)****步骤4:明确环境与数据要求(1小时)****模块3:输出需求文档(企业级模板)**
- 零基础学习性能测试第三章:执行性能测试
试着
性能测试学习性能测试零基础
以下是针对零基础学习性能测试的第三章:执行性能测试的详细学习内容设计,聚焦实战操作与快速应用,助你高效上手:第三章核心目标:学会独立执行完整性能测试,产出有效结果关键原则:标准化流程>工具操作>数据解读>风险规避学习模块1:测试前准备——决定成败的关键(占40%精力)1.1环境搭建标准化为什么重要:环境差异会导致结果失真(最常见失败原因)操作清单:硬件:确保测试服务器配置(CPU/内存/磁盘)≥生
- 零基础学习性能测试第一章-性能测试和功能测试的区别
试着
性能测试学习功能测试性能测试零基础
目录零基础学习性能测试:性能测试与功能测试的核心区别一、核心概念对比(本质区别)1.测试目标差异2.测试方法对比3.工作流程差异二、实际工作场景应用指南1.何时使用功能测试?(适用场景)2.何时使用性能测试?(关键场景)3.协同应用模式三、工具链对比与实践1.工具选择矩阵2.工具使用对比(以用户登录为例)3.报告输出差异四、工作场景决策树决策指南:五、协同工作最佳实践1.项目各阶段配合2.协作工作
- 算力网赋能工业互联网:智能调度与安全融合创新
智能计算研究中心
其他
内容概要工业互联网的深化发展正面临数据处理效率与系统安全性的双重挑战,而算力网通过技术融合与资源协同,成为破解这一难题的核心路径。当前,工业场景中设备互联规模持续扩大,实时数据流处理需求激增,传统集中式计算架构在响应速度与能耗控制方面显现瓶颈。算力网通过构建智能调度机制,实现任务优先级动态划分与资源弹性分配,在智能检测、故障预测等场景中,将平均响应延迟降低40%以上。行业观察:Gartner研究显
- 如何通过 WebSocket 接口订阅实时外汇行情数据(PHP 示例)
quant_1986
websocketphp网络协议开发语言后端网络金融
步骤1:准备工作确保已安装PHP和Composer安装WebSocket客户端库:composerrequiretextalk/websocket步骤2:编写代码订阅行情以下是最简可运行的PHP示例,订阅EUR/USD的1分钟K线数据:60]);//构造订阅请求$initMessage=["code"=>10004,"trace"=>uniqid(),"data"=>["arr"=>[["type
- 【原生JS教程】第3节:运算符与表达式
全栈前端老曹
原生JS教程与实战前端教程javascript开发语言ecmascript前端
第3课:运算符与表达式引言运算符是JavaScript中用于操作数据的基本工具,表达式则是由变量、常量、运算符等构成的可求值代码片段。掌握运算符的分类和使用方式,是编写逻辑判断、数据处理和复杂计算的基础。通过本节课的学习,你将掌握:JavaScript中常见的运算符类型(算术、比较、逻辑、三元等)运算符的优先级与结合性表达式的基本概念与使用场景常见运算符陷阱与最佳实践本节内容概要✅1.算术运算符(
- 6+,基于免疫原性细胞死亡的非肿瘤分型文章,投稿到接收仅一个多月,肿瘤的热点已经传导至非肿瘤生信文章中!
生信小课堂
影响因子:6.147本文从投稿到接收仅一个多月关于非肿瘤生信,我们也解读过很多,主要有以下类型1单个疾病WGCNA+PPI分析筛选hub基因。2单个疾病结合免疫浸润,热点基因集,机器学习,分子分型等。3两种相关疾病联合分析,包括非肿瘤结合非肿瘤,非肿瘤结合肿瘤或者非肿瘤结合泛癌分析目前非肿瘤生信发文的门槛较低,有需要的朋友欢迎交流!研究概述:脑卒中是世界上死亡和残疾的主要原因之一,缺血性中风占80
- 从代码到终端部署:Prompt如何颠覆传统DevOps流程
LCG元
工具运维promptdevops运维
文章目录基于Prompt工程的DevOps架构重构实践一、架构演进与技术对比1.1架构演进路径1.2核心流程对比二、核心实现方案2.1Prompt解析引擎实现(Python)2.2Kubernetes集成部署(YAML模板)三、生产部署实践3.1安全增强方案3.2性能优化数据四、技术前瞻与演进4.1未来三年技术路线图五、完整技术图谱六、核心代码实现(TypeScript前端)七、部署验证测试基于P
- 解锁Prompt+DevOps新姿势:终端系统重塑的三大核心策略
文章目录引言:Prompt驱动的DevOps范式迁移核心策略一:智能决策流水线构建横向架构对比纵向实现流程Python实现示例核心策略二:自适应终端部署体系TypeScript客户端实现YAML部署配置模板核心策略三:智能运维闭环构建安全审计实现方案性能对比分析技术前瞻性分析附录:完整技术图谱技术架构部署验证引言:Prompt驱动的DevOps范式迁移在云原生与AI工程化交汇的今天,Prompt技
- 系统架构设计师教程 第二章 计算机系统基础知识-2.9 系统性能
AncleLeen
软考-系统架构设计师-学习路线系统架构软考-系统架构师
系统架构设计师教程第二章计算机系统基础知识-2.9系统性能2.9.1.性能指标2.9.1.1计算机的性能指标2.9.1.2路由器的性能指标(了解即可)2.9.1.3交换机的性能指标(了解即可)2.9.1.4网络的性能指标2.9.1.5操作系统的性能指标2.9.1.6数据库管理系统的性能指标2.9.1.7Web服务器的性能指标2.9.2.性能计算2.9.3.性能设计2.9.3.1性能调整2.9.3.
- commons-pool2对象池原理简析
月落亦莫离
所谓对象池,即一个放对象的池子。目的是为了复用对象,以减少创建对象的开销,如连接池、线程池等。commons-pool2是apache下的一款对象池开源组件,在学习它的原理前,首先考虑下如果我们自实现对象池,会有哪些问题需要考虑?底层用什么数据结构来做对象池的容器?对象池要有什么属性,支持哪些方法?对象在对象池中的生命周期是什么样的?从对象池获取/归还的步骤?接下来我们带着这些问题去学习commo
- Leetcode703. 数据流中的第K大元素
LonnieQ
题目设计一个找到数据流中第K大元素的类(class)。注意是排序后的第K大元素,不是第K个不同的元素。你的KthLargest类需要一个同时接收整数k和整数数组nums的构造器,它包含数据流中的初始元素。每次调用KthLargest.add,返回当前数据流中第K大的元素。示例:intk=3;int[]arr=[4,5,8,2];KthLargestkthLargest=newKthLargest(
- Java-数构链表
2301_81674311
java链表开发语言
1.链表1.1链表的概念和结构链表是一种物理存储结构上非连续存储结构,数据元素的逻辑顺序是通过链表中引用链接次序实现的。这里大多讨论无头单向非循环链表。这种结构,结构简单,一般与其他数据结构结合,作为其他数据结构的子数据。1.2链表的实现publicclassMysingleList{staticclassListNode{publicintval;//节点的值域publicListNodenex
- 顶刊潜力!国家产融合作试点城市DID
皮皮学姐分享-ppx
人工智能其他经验分享大数据科技
1632顶刊潜力!国家产融合作试点城市DID数据来源国家产融合作试点城市是由工业和信息化部、财政部、中国人民银行、金融监管总局、中国证监会联合组织的项目。国家产融合作试点城市旨在通过产融合作的方式,引导金融资源有效支持实体经济,推动产业与金融的协调发展,探索产融合作新模式新路径,强化对新型工业化的金融支撑。试点城市将采取设立专项基金、提供贷款支持、建设产融信息对接服务平台等措施,促进产业链与金融
- 上位机知识篇---Prompt&PowerShell Prompt
Atticus-Orion
上位机知识篇promptpowershell
在Anaconda环境中,AnacondaPrompt和AnacondaPowerShellPrompt是两个常用的命令行工具,它们的核心功能都是为了方便管理Python环境和执行相关命令,但底层依赖的命令行解释器不同,因此在使用场景和语法上存在一些区别。下面详细介绍两者的差异:1.底层依赖的命令行解释器不同这是两者最根本的区别,决定了它们的语法规则和功能范围:AnacondaPrompt基于Wi
- 三月三日之研报
只是意外一凡凡
今天上班,空闲的时候看了《手把手教你读财报》,和班班请教,明白了成本和权益的区别,也明白了权益的计算方式。看到了班班留的作业,《雨虹的好公司分析》,翻出了之前班班提供的20份研报,看的是一头雾水,之前也只是大致的浏览了目录,好多的信息,好像都全是重点又好像都不是重点,看的脑壳疼……有参照着劲嘉的好公司分析,在理杏仁里把那些基础数据找了出来,试着写了一点,真的是继续不下去……怎么办?很没有头绪和重点
- 零基础学习性能测试第一章:核心性能指标-并发量
试着
性能测试学习性能测试零基础性能指标并发量
目录零基础学习性能测试:第一章-核心性能指标:并发量一、并发量核心概念解析1.并发量定义与分类2.并发量关键特性二、并发量测试的核心价值1.业务意义三角模型2.实际工作场景应用三、并发量测试实战指南1.测试工具配置要点2.并发测试四步流程3.JMeter并发测试实操四、并发瓶颈分析与优化1.并发瓶颈定位矩阵2.常见并发问题解决方案3.电商系统并发优化案例五、工作应用模板与工具1.并发测试报告模板2
- 济源36家亲子鉴定机构地址整理(附2024年汇总鉴定)
国医基因柯主任
济源亲子鉴定机构地址在哪里?济源亲子鉴定机构地址:济源市健康路58号(济源国医基因130-1550-4309)。亲子鉴定作为现代科学技术发展的产物,越来越受到公众的关注。特别是在家庭法律纠纷、财产继承纠纷、移民、以及个人疑虑等方面,亲子鉴定都发挥着不可或缺的作用。那么,对于生活在河南省济源市的市民来说,济源市的亲子鉴定机构地址在哪里呢?济源36家亲子鉴定机构地址整理(附2024年汇总鉴定)济源亲子
- 浅谈EXT2文件系统----inode table
巭犇
文件系统linux数据库运维
Inodetable概述在EXT2文件系统中,inode表(InodeTable)是一个非常重要的结构,用于存储文件和目录的元数据。每个文件和目录都由一个inode(索引节点)来表示,inode中包含了关于该文件或目录的关键信息,如文件的大小、权限、所属用户、时间戳以及指向数据块的指针等。EXT2文件系统将所有inode结构集中存储在inode表中。内核源码structext2_inode{__l
- 数据结构排序算法总结(C语言实现)
xienda
排序算法数据结构算法
以下是常见排序算法的总结及C语言实现,包含时间复杂度、空间复杂度和稳定性分析:1.冒泡排序(BubbleSort)思想:重复比较相邻元素,将较大元素向后移动。时间复杂度:O(n²)(最好O(n),最坏O(n²))空间复杂度:O(1)稳定性:稳定voidbubbleSort(intarr[],intn){for(inti=0;iarr[j+1]){//交换相邻元素inttemp=arr[j];arr
- 分布式推客系统全栈开发指南:SpringCloud+Neo4j+Redis实战解析
wx_ywyy6798
oracle数据库推客系统推客小程序推客系统开发推客小程序开发推客分销系统
一、推客系统概述与市场背景推客系统(或称"推荐客"系统)是一种基于社交关系和内容分发的推荐营销平台,近年来在电商、内容平台和社交媒体领域迅速崛起。根据最新统计数据,2023年全球社交电商市场规模已达1.2万亿美元,其中推客模式的贡献率超过35%。1.1推客系统的核心价值推客系统通过以下机制创造商业价值:社交裂变:利用用户社交网络实现指数级传播精准推荐:基于用户行为和关系链的个性化内容分发激励机制:
- 萤火虫实践队问卷拟定圆满成功
像风吹过七千里
(通讯员朱海鹏2022-1-4)为后续的寒调工作能够顺利进行,萤火虫实践队全体成员于8时15分在腾讯会议平台集中讨论了关于“双减政策”的课题问卷,多维度了分析本课题的调研问卷构成,并由队长安排了后期的相关工作。会议开始,萤火虫实践队成员按时打卡,然后在队长成汪洋的带领下对本次课题展开了探讨,在会议中,萤火虫实践队的成员们分别讲解了自己和本组另外一名组员的问卷初稿,问卷内容包括学生、家长、老师方面。
- 解析进程 /proc/pid/maps 和 /proc/pid/smaps
目录/proc//maps背景具体描述代码实现实践/proc/pid/smapssmaps各子项详解代码实现代码调用的路径如下:小结/proc//maps背景相对于/proc/meminfo和dumpsysmeminfo可以看到系统整体的内存信息,我们还需要能够具体到每一个进程内存占用统计的信息。在分析内存问题的时候,会经常依赖kernel的proc文件系统下各个进程的文件节点,从中获取当前进程的
- mysql.createPool(db)_nodejs解决mysql和连接池(pool)自动断开问题
会咕咕咕的小夫爷
最近在做一个个人项目,数据库尝试使用了mongodb、sqlite和mysql。分享一下关于mysql的连接池用法。项目部署于appfog,项目中我使用连接池链接数据库,本地测试一切正常。上线以后,经过几次请求两个数据接口总是报503。一直不明就里,今天经过一番排查终于顺利解决了。1.mysql链接普通模式varmysql=require('mysql'),env={host:'localhost
- 边缘计算物联网关如何优化数据处理流程-天拓四方
职业影评人
边缘计算物联网人工智能
在物联网技术日新月异的今天,数据的产生、传输与处理已成为推动行业智能化转型的关键。边缘计算物联网关,作为这一生态系统中的核心组件,正以其独特的优势,在数据处理效率、实时性、安全性及成本效益等方面展现出非凡的潜力。本文将聚焦于边缘计算物联网关如何优化数据处理流程,深入探讨其技术原理、应用优势及未来发展趋势。一、边缘计算物联网关概述边缘计算物联网关,简而言之,是位于物联网设备边缘,负责数据收集、初步处
- #零基础学画画#第『111』天 各位早安!
Loura明珠
昨晚下雨,车的防盗器在楼下半夜响,空调定时停了,打蚊子,整晚没睡好。早上下雨,装备齐全出发,到小区门口车车坏了,安乔童自己走路去学校,推车回家赶出门。天气不好,温差大,会造成出行不便,身体不适,大家注意哦。#零基础学画画#第『111』天各位早安!图片发自App
- 二分查找排序算法
周凡杨
java二分查找排序算法折半
一:概念 二分查找又称
折半查找(
折半搜索/
二分搜索),优点是比较次数少,查找速度快,平均性能好;其缺点是要求待查表为有序表,且插入删除困难。因此,折半查找方法适用于不经常变动而 查找频繁的有序列表。首先,假设表中元素是按升序排列,将表中间位置记录的关键字与查找关键字比较,如果两者相等,则查找成功;否则利用中间位置记录将表 分成前、后两个子表,如果中间位置记录的关键字大于查找关键字,则进一步
- java中的BigDecimal
bijian1013
javaBigDecimal
在项目开发过程中出现精度丢失问题,查资料用BigDecimal解决,并发现如下这篇BigDecimal的解决问题的思路和方法很值得学习,特转载。
原文地址:http://blog.csdn.net/ugg/article/de
- Shell echo命令详解
daizj
echoshell
Shell echo命令
Shell 的 echo 指令与 PHP 的 echo 指令类似,都是用于字符串的输出。命令格式:
echo string
您可以使用echo实现更复杂的输出格式控制。 1.显示普通字符串:
echo "It is a test"
这里的双引号完全可以省略,以下命令与上面实例效果一致:
echo Itis a test 2.显示转义
- Oracle DBA 简单操作
周凡杨
oracle dba sql
--执行次数多的SQL
select sql_text,executions from (
select sql_text,executions from v$sqlarea order by executions desc
) where rownum<81;
&nb
- 画图重绘
朱辉辉33
游戏
我第一次接触重绘是编写五子棋小游戏的时候,因为游戏里的棋盘是用线绘制的,而这些东西并不在系统自带的重绘里,所以在移动窗体时,棋盘并不会重绘出来。所以我们要重写系统的重绘方法。
在重写系统重绘方法时,我们要注意一定要调用父类的重绘方法,即加上super.paint(g),因为如果不调用父类的重绘方式,重写后会把父类的重绘覆盖掉,而父类的重绘方法是绘制画布,这样就导致我们
- 线程之初体验
西蜀石兰
线程
一直觉得多线程是学Java的一个分水岭,懂多线程才算入门。
之前看《编程思想》的多线程章节,看的云里雾里,知道线程类有哪几个方法,却依旧不知道线程到底是什么?书上都写线程是进程的模块,共享线程的资源,可是这跟多线程编程有毛线的关系,呜呜。。。
线程其实也是用户自定义的任务,不要过多的强调线程的属性,而忽略了线程最基本的属性。
你可以在线程类的run()方法中定义自己的任务,就跟正常的Ja
- linux集群互相免登陆配置
林鹤霄
linux
配置ssh免登陆
1、生成秘钥和公钥 ssh-keygen -t rsa
2、提示让你输入,什么都不输,三次回车之后会在~下面的.ssh文件夹中多出两个文件id_rsa 和 id_rsa.pub
其中id_rsa为秘钥,id_rsa.pub为公钥,使用公钥加密的数据只有私钥才能对这些数据解密 c
- mysql : Lock wait timeout exceeded; try restarting transaction
aigo
mysql
原文:http://www.cnblogs.com/freeliver54/archive/2010/09/30/1839042.html
原因是你使用的InnoDB 表类型的时候,
默认参数:innodb_lock_wait_timeout设置锁等待的时间是50s,
因为有的锁等待超过了这个时间,所以抱错.
你可以把这个时间加长,或者优化存储
- Socket编程 基本的聊天实现。
alleni123
socket
public class Server
{
//用来存储所有连接上来的客户
private List<ServerThread> clients;
public static void main(String[] args)
{
Server s = new Server();
s.startServer(9988);
}
publi
- 多线程监听器事件模式(一个简单的例子)
百合不是茶
线程监听模式
多线程的事件监听器模式
监听器时间模式经常与多线程使用,在多线程中如何知道我的线程正在执行那什么内容,可以通过时间监听器模式得到
创建多线程的事件监听器模式 思路:
1, 创建线程并启动,在创建线程的位置设置一个标记
2,创建队
- spring InitializingBean接口
bijian1013
javaspring
spring的事务的TransactionTemplate,其源码如下:
public class TransactionTemplate extends DefaultTransactionDefinition implements TransactionOperations, InitializingBean{
...
}
TransactionTemplate继承了DefaultT
- Oracle中询表的权限被授予给了哪些用户
bijian1013
oracle数据库权限
Oracle查询表将权限赋给了哪些用户的SQL,以备查用。
select t.table_name as "表名",
t.grantee as "被授权的属组",
t.owner as "对象所在的属组"
- 【Struts2五】Struts2 参数传值
bit1129
struts2
Struts2中参数传值的3种情况
1.请求参数绑定到Action的实例字段上
2.Action将值传递到转发的视图上
3.Action将值传递到重定向的视图上
一、请求参数绑定到Action的实例字段上以及Action将值传递到转发的视图上
Struts可以自动将请求URL中的请求参数或者表单提交的参数绑定到Action定义的实例字段上,绑定的规则使用ognl表达式语言
- 【Kafka十四】关于auto.offset.reset[Q/A]
bit1129
kafka
I got serveral questions about auto.offset.reset. This configuration parameter governs how consumer read the message from Kafka when there is no initial offset in ZooKeeper or
- nginx gzip压缩配置
ronin47
nginx gzip 压缩范例
nginx gzip压缩配置 更多
0
nginx
gzip
配置
随着nginx的发展,越来越多的网站使用nginx,因此nginx的优化变得越来越重要,今天我们来看看nginx的gzip压缩到底是怎么压缩的呢?
gzip(GNU-ZIP)是一种压缩技术。经过gzip压缩后页面大小可以变为原来的30%甚至更小,这样,用
- java-13.输入一个单向链表,输出该链表中倒数第 k 个节点
bylijinnan
java
two cursors.
Make the first cursor go K steps first.
/*
* 第 13 题:题目:输入一个单向链表,输出该链表中倒数第 k 个节点
*/
public void displayKthItemsBackWard(ListNode head,int k){
ListNode p1=head,p2=head;
- Spring源码学习-JdbcTemplate queryForObject
bylijinnan
javaspring
JdbcTemplate中有两个可能会混淆的queryForObject方法:
1.
Object queryForObject(String sql, Object[] args, Class requiredType)
2.
Object queryForObject(String sql, Object[] args, RowMapper rowMapper)
第1个方法是只查
- [冰川时代]在冰川时代,我们需要什么样的技术?
comsci
技术
看美国那边的气候情况....我有个感觉...是不是要进入小冰期了?
那么在小冰期里面...我们的户外活动肯定会出现很多问题...在室内呆着的情况会非常多...怎么在室内呆着而不发闷...怎么用最低的电力保证室内的温度.....这都需要技术手段...
&nb
- js 获取浏览器型号
cuityang
js浏览器
根据浏览器获取iphone和apk的下载地址
<!DOCTYPE html>
<html>
<head>
<meta charset="utf-8" content="text/html"/>
<meta name=
- C# socks5详解 转
dalan_123
socketC#
http://www.cnblogs.com/zhujiechang/archive/2008/10/21/1316308.html 这里主要讲的是用.NET实现基于Socket5下面的代理协议进行客户端的通讯,Socket4的实现是类似的,注意的事,这里不是讲用C#实现一个代理服务器,因为实现一个代理服务器需要实现很多协议,头大,而且现在市面上有很多现成的代理服务器用,性能又好,
- 运维 Centos问题汇总
dcj3sjt126com
云主机
一、sh 脚本不执行的原因
sh脚本不执行的原因 只有2个
1.权限不够
2.sh脚本里路径没写完整。
二、解决You have new mail in /var/spool/mail/root
修改/usr/share/logwatch/default.conf/logwatch.conf配置文件
MailTo =
MailFrom
三、查询连接数
- Yii防注入攻击笔记
dcj3sjt126com
sqlWEB安全yii
网站表单有注入漏洞须对所有用户输入的内容进行个过滤和检查,可以使用正则表达式或者直接输入字符判断,大部分是只允许输入字母和数字的,其它字符度不允许;对于内容复杂表单的内容,应该对html和script的符号进行转义替换:尤其是<,>,',"",&这几个符号 这里有个转义对照表:
http://blog.csdn.net/xinzhu1990/articl
- MongoDB简介[一]
eksliang
mongodbMongoDB简介
MongoDB简介
转载请出自出处:http://eksliang.iteye.com/blog/2173288 1.1易于使用
MongoDB是一个面向文档的数据库,而不是关系型数据库。与关系型数据库相比,面向文档的数据库不再有行的概念,取而代之的是更为灵活的“文档”模型。
另外,不
- zookeeper windows 入门安装和测试
greemranqq
zookeeper安装分布式
一、序言
以下是我对zookeeper 的一些理解: zookeeper 作为一个服务注册信息存储的管理工具,好吧,这样说得很抽象,我们举个“栗子”。
栗子1号:
假设我是一家KTV的老板,我同时拥有5家KTV,我肯定得时刻监视
- Spring之使用事务缘由(2-注解实现)
ihuning
spring
Spring事务注解实现
1. 依赖包:
1.1 spring包:
spring-beans-4.0.0.RELEASE.jar
spring-context-4.0.0.
- iOS App Launch Option
啸笑天
option
iOS 程序启动时总会调用application:didFinishLaunchingWithOptions:,其中第二个参数launchOptions为NSDictionary类型的对象,里面存储有此程序启动的原因。
launchOptions中的可能键值见UIApplication Class Reference的Launch Options Keys节 。
1、若用户直接
- jdk与jre的区别(_)
macroli
javajvmjdk
简单的说JDK是面向开发人员使用的SDK,它提供了Java的开发环境和运行环境。SDK是Software Development Kit 一般指软件开发包,可以包括函数库、编译程序等。
JDK就是Java Development Kit JRE是Java Runtime Enviroment是指Java的运行环境,是面向Java程序的使用者,而不是开发者。 如果安装了JDK,会发同你
- Updates were rejected because the tip of your current branch is behind
qiaolevip
学习永无止境每天进步一点点众观千象git
$ git push joe prod-2295-1
To
[email protected]:joe.le/dr-frontend.git
! [rejected] prod-2295-1 -> prod-2295-1 (non-fast-forward)
error: failed to push some refs to '
[email protected]
- [一起学Hive]之十四-Hive的元数据表结构详解
superlxw1234
hivehive元数据结构
关键字:Hive元数据、Hive元数据表结构
之前在 “[一起学Hive]之一–Hive概述,Hive是什么”中介绍过,Hive自己维护了一套元数据,用户通过HQL查询时候,Hive首先需要结合元数据,将HQL翻译成MapReduce去执行。
本文介绍一下Hive元数据中重要的一些表结构及用途,以Hive0.13为例。
文章最后面,会以一个示例来全面了解一下,
- Spring 3.2.14,4.1.7,4.2.RC2发布
wiselyman
Spring 3
Spring 3.2.14、4.1.7及4.2.RC2于6月30日发布。
其中Spring 3.2.1是一个维护版本(维护周期到2016-12-31截止),后续会继续根据需求和bug发布维护版本。此时,Spring官方强烈建议升级Spring框架至4.1.7 或者将要发布的4.2 。
其中Spring 4.1.7主要包含这些更新内容。