KiU-Net: Overcomplete Convolutional Architectures for Biomedical Image and Volumetric Segmentation
KiU-Net: Overcomplete Convolutional(过完备卷积) Architectures for Biomedical Image(生物医学图像) and Volumetric Segmentation(血管分割)
论文:KiU-Net: Overcomplete Convolutional Architectures for Biomedical Image and Volumetric Segmentation
源码:https://github.com/jeya-maria-jose/KiU-Net-pytorch
Q:什么是过完备(Overcomplete)?
A:指的是基函数的个数超过输入信号的采样个数,以减少信息丢损,同时也有利于发掘数据中的结构。
目录
一、摘要
二、创新点
三、方法
1. Overcomplete Networks(过完备网络 Kite-Net)
2. Undercomplete Networks(不完备网络 U-Net 3D)
3. Cross Residual Feature Block(CRFB模块)
四、实验
1. 定量分析
2. 定性分析
五、讨论
1. Res-KiUNet
2. Dense-KiUNet
六、总结
一、摘要
研究背景:大多数医学图像分割方法都使用 U-Net 或其变体,因为它们在大多数应用中都取得了成功。在对这些基于编码器-解码器的“传统”方法进行详细分析后,发现它们在检测较小结构方面表现不佳,并且无法精确分割边界区域。
这个问题可以归因于随着深入编码器,感受野大小的增加。对学习高级特征的额外关注,导致基于 U-Net 的方法学习较少的低级特征信息,而低级特征对于检测小结构至关重要。
主要工作:为了克服这个问题,作者提出使用一个过完备的卷积架构,将输入图像投影到一个更高的维度,这样就可以限制感受野在网络的深层增加。设计了一种新的图像分割架构 KiU-Net,它有两个分支:
(1) 上分支:学习捕获输入的精细细节和精确边缘的过完备卷积网络 Kite-Net。
(2) 下分支:学习高级特征的 U-Net。
此外,还提出了 KiU-Net 3D,这是一种用于体积分割的三维卷积架构。
研究成果:通过在覆盖各种图像模式的五个不同数据集上进行实验,对 KiU-Net 进行了详细的研究。以更少的参数和更快的收敛速度的额外好处实现了良好的性能。我们还证明了基于残差块和密集块的 KiU-Net 扩展可以进一步提高性能。
二、创新点
1. 提出了KiU-Net 3D,这是KiU-Net用于体积分割的扩展。使用基于3D卷积的过完备网络来有效提取低级信息。
2. 提出了Res-KiUNet和Dense-KiUNet架构,分别使用剩余连接和密集块来改善网络的学习。
3. 评估了所提出的架构在5个数据集上进行体积和图像分割的性能:脑肿瘤分割(BraTS)、肝肿瘤分割(LiTS)、腺体分割(GlaS)、视网膜图像血管树提取(RITE)和脑解剖分割。这些数据集分别对应于5种不同的模态:超声(US)、磁共振成像(MRI)、计算机断层扫描(CT)、显微镜(microscopic)和眼底图像(fundus)。通过在多个数据集上进行这些额外的实验,证明了所提出的方法可以很好地推广到不同的模态。
三、方法
用于3D体积分割的KiU-Net 3D的架构,主要由Kite-Net 3D,CRFB模块和U-Net 3D组成,如下图所示:
1. Overcomplete Networks(过完备网络 Kite-Net)
Kite-Net,它是 U-Net 的一个过完备版本。
目的:使用基于3D卷积的过完备网络来捕获输入的精细细节和精确边缘(低级的小特征)。
方法:过完备架构Kite-Net在编码器中具有上采样层,在解码器中具有最大池化层,这确保了网络深层过滤器的感受野大小不会像U-Net那样增加,从而便于Kite-Net提取边界的精细细节以及更深层的小结构。
过程:在Kite-Net 3D分支的编码器中,每个conv块都有一个conv 3D层,后面是一个系数为2的三线性上采样层和ReLU激活(共三层)。在解码器中,每个conv块都有一个conv 3D层,后面是一个系数为2的3D最大池化层和ReLU激活(共三层)。此外,同层之间还有跳连接进行特征融合。
2. Undercomplete Networks(不完备网络 U-Net 3D)
目的: 提取高级特征,弥补Kite-Net缺乏提取高级特征的过滤器的缺陷。
过程:在U-Net 3D分支的编码器中,每个conv块都有一个conv 3D层,后面是一个系数为2的3D最大池化层和ReLU激活。在解码器中,每个conv块都有一个conv 3D层,后面是一个系数为2的三线性上采样层和ReLU激活。此外,同层之间也有跳连接进行特征融合。
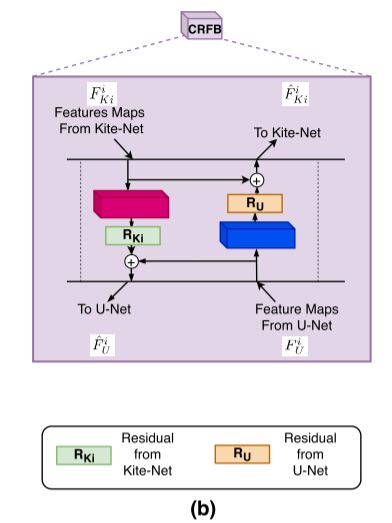
3. Cross Residual Feature Block(CRFB模块)
CRFB模块(交叉残差特征模块),如下图所示:
目的:将以上两种网络在多个尺度上的特征结合起来,以此从两个网络中学习互补的特征,进一步提高单个网络学习到的特征的质量。
方法:在 CRFB中,学习 Kite-Net 3D的残差特征并将其添加到 U-Net 3D的特征中,将互补特征转发给U-Net,反之亦然。
过程:将来自U-Net 3D的第 i 个特征图表示为![]() ,来自Kite-Net 3D的第 i 个特征图表示为
,来自Kite-Net 3D的第 i 个特征图表示为![]() 。首先使用conv块从
。首先使用conv块从![]() ,
,![]() 中提取交叉残差特征
中提取交叉残差特征![]() 和
和![]() 。
。![]() 被转发到的conv块具有conv 3D层和最大池化层的组合。
被转发到的conv块具有conv 3D层和最大池化层的组合。![]() 被转发通过的conv块具有conv 3D层和上采样层的组合。然后将这些跨层残差特征添加到原始特征
被转发通过的conv块具有conv 3D层和上采样层的组合。然后将这些跨层残差特征添加到原始特征![]() 和
和![]() (残差连接),以获得互补特征
(残差连接),以获得互补特征![]() 和
和![]() ,公式定义如下:
,公式定义如下:
![]() ,
,
![]()
![]() 传入U-Net 3D的下一层和
传入U-Net 3D的下一层和 ![]() 传入Kite-Net 3D的下一层。
传入Kite-Net 3D的下一层。
四、实验
数据集:脑解剖分割 (超声) 数据集,收集了 20 个不同早产儿的 US 扫描图,收集的图像总数为 1629 张带有标注的图像,其中1300张被分配用于训练,以及329张用于测试;腺体分割 (显微镜) 数据集,Gland分割(GLAS)数据集共包含 165 张图像,其中85张用于训练,80张用于测试;视网膜神经分段 (眼底) 数据集,RITE 数据集包含 40 张图像,分为20张用于训练,20张用于测试。以上数据集,图像预处理为大小128 x 128,用于KiU-Net训练。
脑肿瘤分割 (磁共振成像) 数据集,注释分为四类--增强肿瘤、瘤周水肿和坏死及非增强肿瘤核心,总共有 335 次MRI扫描;肝脏分割 (CT) 数据集,包含增强腹部CT扫描以及肝脏和肝脏病变的标注,使用数据集的109个CT扫描进行训练,并在27到48之间的21个CT扫描上进行测试。图像预处理为大小255 x 255,用于KiU-Net 3D训练。
损失函数:KiU-Net使用binary cross entropy,KiU-Net 3D使用cross entropy,定义公式如下:
其中,w 和 h 是图像的维度,p(x, y)对应于图像,![]() 表示在特定像素位置(x, y)的输出预测。
表示在特定像素位置(x, y)的输出预测。
超参数:设置batch-size为1,学习率为0.001。使用Adam优化器进行训练。训练提出的网络 300 次或直到收敛取决于数据集。
实验细节:使用Python中的Pytorch框架进行的。实验使用两块NVIDIA-RTX 2080Ti gpu 进行。
评估指标:dice系数 (F1-score)。
1. 定量分析
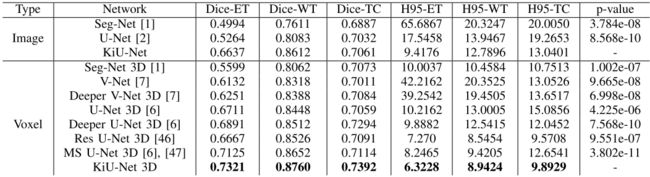
KiU-Net,KiU-Net 3D与先进网络之间的对比试验:
与所有对比网络相比,KiU-Net和KiU-Net 3D在性能方面实现了显着提升。
2. 定性分析
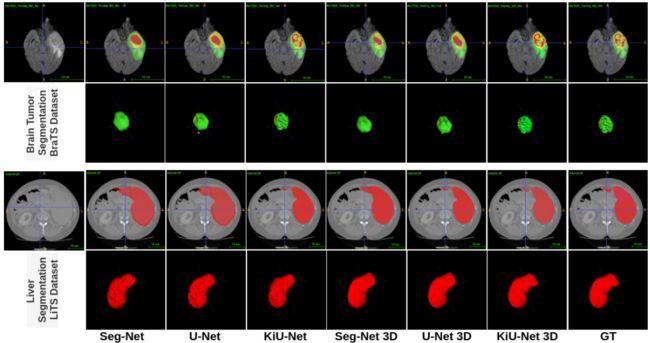
KiU-Net,KiU-Net 3D与先进网络之间预测结果的对比分析:
我们可以观察到 KiU-Net 能够精确地分割小脑室,而其他传统的网络无法做到这一点。KiU-Net 3D也能够比任何其他网络明显更好地分割肿瘤的表面和边缘,并且更接近真值。
五、讨论
两种不同的改进思路:基于残差模块和密集模块的改进。
1. Res-KiUNet
在每个分支在每个层上都添加残差连接。
2. Dense-KiUNet
每个分支在每个层上都添加密集模块。密集模块包含4个conv层,每个conv层都只输出k/4特征图,最后将所有的conv层的输出进行连接获得一个完整的特征图。
效果对比:
可以观察到,与KiU-Net相比,Dense-KiUNet 和 Res-KiUNet 提供了进一步的性能改进。
六、总结
1. 根据感受野的增大不利于学习细小的低级特征的结论,作者提出了超完备卷积架构(Kite-Net ),用于抑制感受野,更精确地学习小掩模和表面和边缘的更精细细节。
2. 为了学习低级特征的同时,学习高级特征,作者使用了双分支的网络结构,网络下分支使用U-Net进行高级特征的提取。
3. 为了在多个尺度上将低级特征和高级特征结合起来,作者提出了CRFB模块,使用残差连接的方式将跨层特征进行融合。