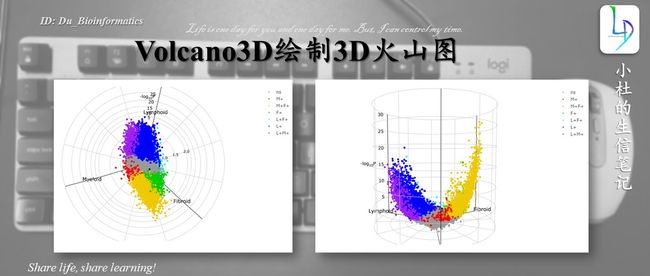
Volcano3D绘制3D火山图
一边学习,一边总结,一边分享!
本期教程内容
**注:**本教程详细内容
Volcano3D绘制3D火山图
一、前言
火山图是做差异分析中最常用到的图形,在前面的推文中,我们也推出了好几期火山图的绘制教程,以及很多火山图的教程也可以参考。R语言绘制精美图形 | 火山图 | 学习笔记、各类差异基因火山图汇总 | 学习笔记等等。大家根据自己的需求绘制相关的火山图即可。
今天,我们学习使用volcano3D绘制3D火山图,我们会给出详细的绘图过程。
注意: 小杜的生信笔记分享的教程是结合自己的需求进行分享,难免会有错误或遗漏,望见谅!
二、GitHub网址
https://github.com/KatrionaGoldmann/volcano3D
三、 加载R包和数据
3.1 安装volcano3D包
### 安装volcano3D包
library(devtools)
#install_github("KatrionaGoldmann/volcano3D")
### 或是使用以下方法进行安装
# repos = getOption("repos")
# repos["volcano3Ddata"] = "http://KatrionaGoldmann.github.io/volcano3Ddata"
# options(repos = repos)
#install.packages("volcano3Ddata")
library(volcano3Ddata)
3. 2 安装R包时报错问题处理
我自己在安装volcano3D1时出现的报错,安装不了。我开了VPN,但也是报错。后看了是如下的问题,volcano3D相关的包vctrs版本太低了,然后进行升级后即可安装。

3.3 加载数据包
devtools::install_github("KatrionaGoldmann/volcano3Ddata")
citation("volcano3D")
3.4 加载R包
library(volcano3D)
library(volcano3Ddata)
# Basic DESeq2 set up
library(DESeq2)
library(ggplot2)
library(kableExtra)
3.5 数据展示
结合示例数据进行准备数据,基本与我们平时做差异分析数据保持一致,或是你直接使用DESep2做差异分析,使用其结果进行绘图。
citation("volcano3D")
##
## To cite package 'volcano3D' in publications use:
##
## Goldmann K, Lewis M (2020). _volcano3D: 3D Volcano Plots and Polar
## Plots for Three-Class Data_.
## https://katrionagoldmann.github.io/volcano3D/index.html,
## https://github.com/KatrionaGoldmann/volcano3D.
##
## A BibTeX entry for LaTeX users is
##
## @Manual{,
## title = {volcano3D: 3D Volcano Plots and Polar Plots for Three-Class Data},
## author = {Katriona Goldmann and Myles Lewis},
## year = {2020},
## note = {https://katrionagoldmann.github.io/volcano3D/index.html, https://github.com/KatrionaGoldmann/volcano3D},
## }
data("syn_data")
data("syn_txi")
3.6 差异分析
创建数据集
counts <- matrix(rnbinom(n=3000, mu=100, size=1/0.5), ncol=30)
rownames(counts) <- paste0("gene", 1:100)
cond <- rep(factor(rep(1:3, each=5), labels = c('A', 'B', 'C')), 2)
resp <- factor(rep(1:2, each=15), labels = c('non.responder', 'responder'))
metadata <- data.frame(drug = cond, response = resp)
counts[1:10,1:5]
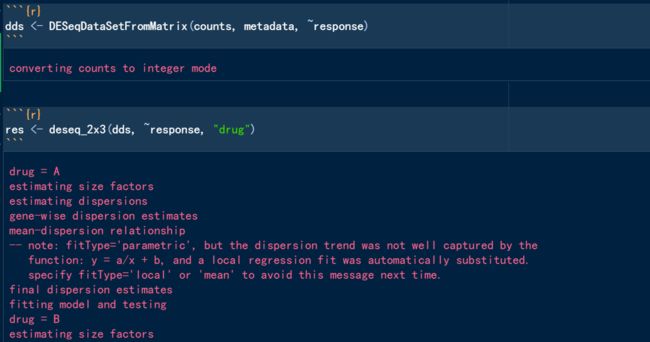
dds <- DESeqDataSetFromMatrix(counts, metadata, ~response)
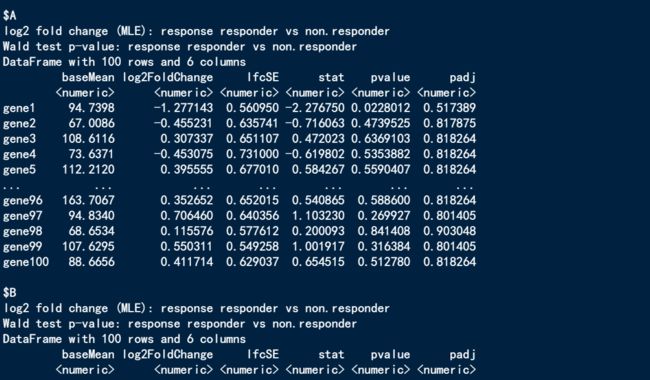
res <- deseq_2x3(dds, ~response, "drug")
head(res)
函数deseq_2x3()返回一个包含3个DESeq2对象的列表,其中包含3种药物中每一种的响应分析。这些响应与非响应差异表达式比较可以通过R软件包easylabel的easyVolcano()函数快速可视化,该软件包专为DESeq2和limma对象设计,并使用交互式。 R/发光界面。
#install.packages("easylabel")
library(easylabel)
df <- as.data.frame(res[[1]]) # results for the first drug
easyVolcano(df)
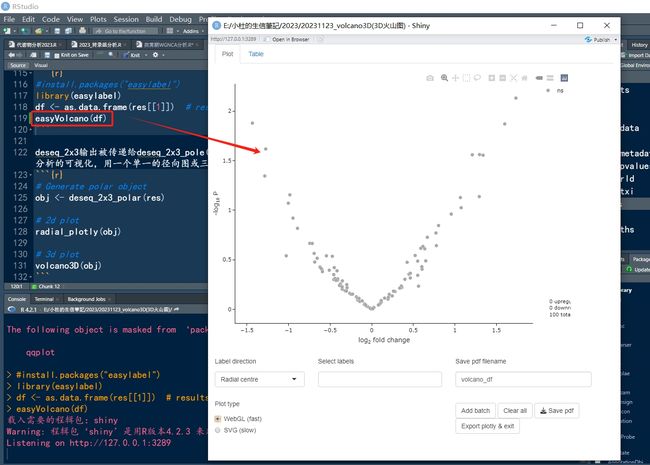
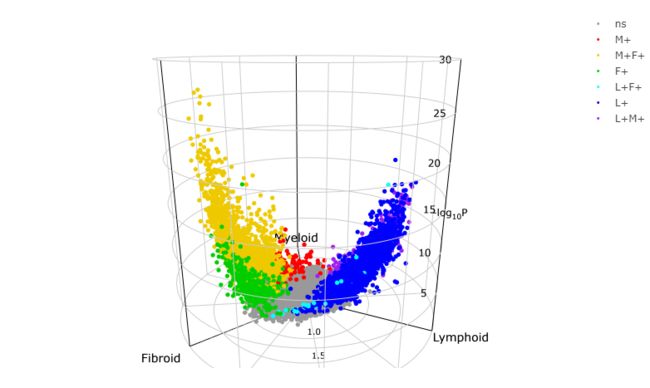
deseq_2x3输出被传递给deseq_2x3_pole()以生成一个volc3d类对象用于绘图。因此,三维径向图和三维火山图简化了二乘三维分析的可视化,用一个单一的径向图或三维火山图取代了三个火山图。
# Generate polar object
obj <- deseq_2x3_polar(res)
# 2d plot
radial_plotly(obj)
# 3d plot
volcano3D(obj)
四、绘制不同药理的图形
kable(table(syn_metadata$Pathotype), col.names = c("Pathotype", "Count"))
syn_metadata$Pathotype <- factor(syn_metadata$Pathotype,
levels = c('Lymphoid', 'Myeloid', 'Fibroid'))
##设置原始变量
dds <- DESeqDataSetFromTximport(txi = syn_txi,
colData = syn_metadata,
design = ~ Pathotype + Batch + Gender)
##差异分析
dds_DE <- DESeq(dds)
##病理类型
dds_LRT <- DESeq(dds, test = "LRT", reduced = ~ Batch + Gender, parallel = TRUE)
#创建'volc3d' 数据集
res <- deseq_polar(dds_DE, dds_LRT, "Pathotype")
##绘制三维散点图
volcano3D(res)
五、构建一个3D shiny
构建一个shiny,也可以单独放在一个R脚本中
Volcano3D绘制3D火山图
往期文章:
1. 复现SCI文章系列专栏
2. 《生信知识库订阅须知》,同步更新,易于搜索与管理。
3. 最全WGCNA教程(替换数据即可出全部结果与图形)
-
WGCNA分析 | 全流程分析代码 | 代码一
-
WGCNA分析 | 全流程分析代码 | 代码二
-
WGCNA分析 | 全流程代码分享 | 代码三
-
WGCNA分析 | 全流程分析代码 | 代码四
-
WGCNA分析 | 全流程分析代码 | 代码五(最新版本)
4. 精美图形绘制教程
- 精美图形绘制教程
5. 转录组分析教程
转录组上游分析教程[零基础]
小杜的生信筆記 ,主要发表或收录生物信息学的教程,以及基于R的分析和可视化(包括数据分析,图形绘制等);分享感兴趣的文献和学习资料!!