R语言基于Matching包进行倾向评分匹配(PSM)
倾向评分匹配(Propensity Score Matching,简称PSM)是一种统计学方法,用于处理观察研究(Observational Study)的数据,在SCI文章中应用非常广泛。在观察研究中,由于种种原因,数据偏差(bias)和混杂变量(confounding variable)较多,倾向评分匹配的方法正是为了减少这些偏差和混杂变量的影响,以便对实验组和对照组进行更合理的比较。
为什么需要做倾向评分匹配?
我们知道RCT的证据力度高,是因为对患者进行了严格的筛选。我们的回顾性研究都是过去的数据,很难像RCT一样进行严格的筛选出两组患者基线相近的基础资料,但我们可以通过倾向评分匹配把回归性的数据进行筛选,把基线资料相近的患者进行匹配,得到近似RCT的效果。
应用场景
1.基线资料不平
2.开展病例对照研究病阳性例数较少,如罕见病研究
3.将众多混杂因素变为一个变量:倾向值
以下为一个实例,没进行匹配前两组患者基线资料相差很大,进行倾向评分匹配后,基线资料近似一致了

既往我们已经介绍了《利用SPSS进行PSM倾向性评分配对》,今日继续介绍R语言进行PSM倾向性评分配对,可以进行PSM的R包很多,我们今天来介绍Matching包,继续使用我们的早产数据,我们先导入R包和数据
library(Matching)
library(tableone)
bc<-read.csv("E:/r/test/zaochan.csv",sep=',',header=TRUE)
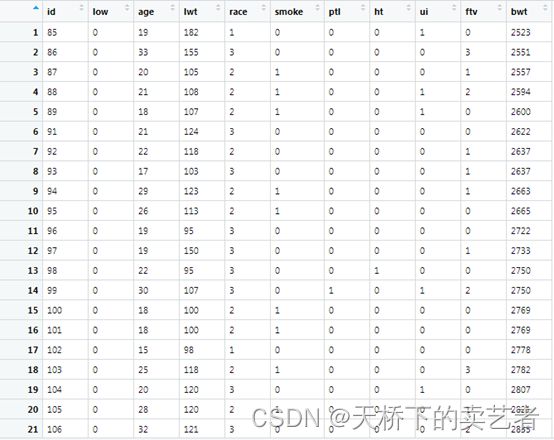
这是一个关于早产低体重儿的数据(公众号回复:早产数据,可以获得该数据),低于2500g被认为是低体重儿。数据解释如下:low 是否是小于2500g早产低体重儿,age 母亲的年龄,lwt 末次月经体重,race 种族,smoke 孕期抽烟,ptl 早产史(计数),ht 有高血压病史,ui 子宫过敏,ftv 早孕时看医生的次数,bwt 新生儿体重数值。
我们先把分类变量转成因子
bc$race<-ifelse(bc$race=="black",1,ifelse(bc$race=="white",2,3))
bc$smoke<-ifelse(bc$smoke=="nonsmoker",0,1)
bc$low<-factor(bc$low)
bc$race<-factor(bc$race)
bc$ht<-factor(bc$ht)
bc$ui<-factor(bc$ui)
假设我们研究的是有无高血压(ht)对生出低体重儿(low)的影响,我们先绘制一个患者基线表
dput(names(bc))##输出变量名
allVars <-c("age", "lwt", "race", "smoke", "ptl", "ui",
"ftv")###所有变量名
fvars<-c("race", "smoke","ui")#分类变量定义为fvars
tab2 <- CreateTableOne(vars = allVars, strata = "ht" , data = bc, factorVars=fvars,
addOverall = TRUE )###绘制基线表
print(tab2)#输出表格

我们可以看到基线资料中有高血压组和没有高血压组除了末次月经的体重不一样,其他资料基本都是配平的。
我们需要进行倾向评分匹配,所以要先生成倾向评分,先建立方程
fit1<- glm(ht ~age + lwt + race + smoke + ptl + ui + ftv, data=bc,
family=binomial(link = "logit"))
倾向评分就是fit1$fitted,你用predict生成也是一样的,先生成Match的3个参数
X <- fit1$fitted
Y <- bc$low
Tr<- (bc$ht == 1)
其实Y这个参数,生成不生成都是一样的,M=1代表默认1:1匹配
rr <- Match(Y=Y, Tr=Tr, X=X, M=1)
生成了一个rr的列表(下图),匹配的数据在index.treated和index.control里面

我们把这两个数据提取出来
bcMatched <- bc[unlist(rr [c("index.treated","index.control")]), ]
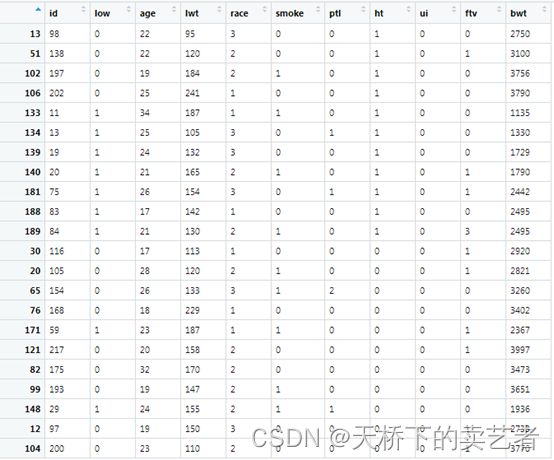
我们来看看提取出来的数据(下图),一共提取出30个数据,但是我们看到id为98的数据重复匹配了

我们进一步设置一下,设置卡钳和replace,但这样进一步要求会继续损失数据
rr1 <- Match(Tr=Tr, X=X, M=1,caliper=0.2,replace = FALSE)#Y不设置也可以
bcMatched2<- bc[unlist(rr1 [c("index.treated","index.control")]), ]
tab3 <- CreateTableOne(vars = allVars, strata = "ht" , data = bcMatched2, factorVars=fvars,
addOverall = TRUE )###绘制基线表
print(tab3)#输出表格

我们可以看到,lwt已经被配平了,之前在没配平前时是小于0.05的,这样患者的基线的年龄和体重就被配平了。我们在这个基础上还可以进行逆概率加权,还可以绘制差异SMD,我这里就不弄了,感兴趣的可以参看我的其他文章。