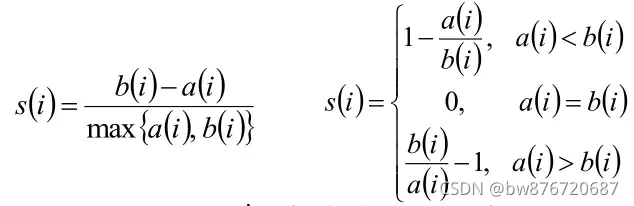机器学习——Kmeans聚类算法
目录
- 简介
- 手肘法
-
- 手肘法核心思想
- 轮廓系数
- 代码举例1
- 代码举例2
- 实例
简介
K均值聚类算法是先随机选取K个对象作为初始的聚类中心。然后计算每个对象与各个种子聚类中心之间的距离,把每个对象分配给距离它最近的聚类中心。聚类中心以及分配给它们的对象就代表一个聚类。每分配一个样本,聚类的聚类中心会根据聚类中现有的对象被重新计算。这个过程将不断重复直到满足某个终止条件。终止条件可以是没有(或最小数目)对象被重新分配给不同的聚类,没有(或最小数目)聚类中心再发生变化,误差平方和局部最小。
手肘法
最关键的问题是其中k值的确定方式
核心指标:SSE(sum of the squared errors,误差平方和)
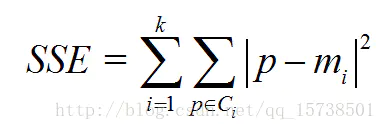
- Ci是第i个簇
- p是Ci中的样本点
- mi是Ci的质心(Ci中所有样本的均值)
- SSE是所有样本的聚类误差,代表了聚类效果的好坏。
手肘法核心思想
-
随着聚类数k的增大,样本划分会更加精细,每个簇的聚合程度会逐渐提高,那么误差平方和SSE自然会逐渐变小。
-
当k小于真实聚类数时,由于k的增大会大幅增加每个簇的聚合程度,故SSE的下降幅度会很大,而当k到达真实聚类数时,再增加k所得到的聚合程度回报会迅速变小,所以SSE的下降幅度会骤减,然后随着k值的继续增大而趋于平缓,也就是说SSE和k的关系图是一个手肘的形状,而这个肘部对应的k值就是数据的真实聚类数
import pandas as pd
from sklearn.cluster import KMeans
import matplotlib.pyplot as plt
df_features = pd.read_csv(r'C:\预处理后数据.csv',encoding='gbk') # 读入数据
'利用SSE选择k'
SSE = [] # 存放每次结果的误差平方和
for k in range(1,9):
estimator = KMeans(n_clusters=k) # 构造聚类器
estimator.fit(df_features[['R','F','M']])
SSE.append(estimator.inertia_) # estimator.inertia_获取聚类准则的总和
X = range(1,9)
plt.xlabel('k')
plt.ylabel('SSE')
plt.plot(X,SSE,'o-')
plt.show()
轮廓系数
但是问题就出在于如何通过图像确定k值?
直接通过肉眼判断是不可信的,因此选择使用轮廓系数
使用轮廓系数(silhouette coefficient)来确定,选择使系数较大所对应的k值
方法:
-
计算样本i到同簇其他样本的平均距离ai。ai 越小,说明样本i越应该被聚类到该簇。将ai 称为样本i的簇内不相似度。
簇C中所有样本的a i 均值称为簇C的簇不相似度。 -
计算样本i到其他某簇Cj 的所有样本的平均距离bij,称为样本i与簇Cj 的不相似度。定义为样本i的簇间不相似度:bi =min{bi1, bi2, …, bik}
bi越大,说明样本i越不属于其他簇。 -
判断:
-
轮廓系数范围在[-1,1]之间。该值越大,越合理。
si接近1,则说明样本i聚类合理;
si接近-1,则说明样本i更应该分类到另外的簇;
若si 近似为0,则说明样本i在两个簇的边界上。 -
所有样本的s i 的均值称为聚类结果的轮廓系数,是该聚类是否合理、有效的度量。
-
使用轮廓系数(silhouette coefficient)来确定,选择使系数较大所对应的k值
-
sklearn.metrics.silhouette_score sklearn中有对应的求轮廓系数的API
增加轮廓系数改进之后判断代码:
def cluster_para_est(train_x):
k_num = 50
sse = []
for k in range(1, k_num):
# kmeans算法
kmeans = KMeans(n_clusters=k)
kmeans.fit(train_x)
# 计算inertia簇内误差平方和
sse.append(kmeans.inertia_)
x = range(1, k_num)
plt.xlabel('K')
plt.ylabel('SSE')
plt.plot(x, sse, 'o-')
plt.show()
for idx in range(len(sse) - 1):
if sse[idx] - sse[idx + 1] <= 1:
print(idx + 1)
return idx + 1
代码举例1
1、便于理解,首先创建一个明显分为2类20*2的例子(每一列为一个变量共2个变量,每一行为一个样本共20个样本):
import numpy as np
c1x=np.random.uniform(0.5,1.5,(1,10))
c1y=np.random.uniform(0.5,1.5,(1,10))
c2x=np.random.uniform(3.5,4.5,(1,10))
c2y=np.random.uniform(3.5,4.5,(1,10))
x=np.hstack((c1x,c2x))
y=np.hstack((c2y,c2y))
X=np.vstack((x,y)).T
print(X)
2、引用Python库将样本分为两类(k=2),并绘制散点图:
#只需将X修改即可进行其他聚类分析
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
kemans=KMeans(n_clusters=2)
result=kemans.fit_predict(X) #训练及预测
print(result) #分类结果
plt.rcParams['font.family'] = ['sans-serif']
plt.rcParams['font.sans-serif'] = ['SimHei'] #散点图标签可以显示中文
x=[i[0] for i in X]
y=[i[1] for i in X]
plt.scatter(x,y,c=result,marker='o')
plt.xlabel('x')
plt.ylabel('y')
plt.show()
3、如果K值未知,可采用肘部法选择K值(假设最大分类数为9类,分别计算分类结果为1-9类的平均离差,离差的提升变化下降最抖时的值为最优聚类数K)
import matplotlib.pyplot as plt
from sklearn.cluster import KMeans
from scipy.spatial.distance import cdist
K=range(1,10)
meanDispersions=[]
for k in K:
kemans=KMeans(n_clusters=k)
kemans.fit(X)
#计算平均离差
m_Disp=sum(np.min(cdist(X,kemans.cluster_centers_,'euclidean'),axis=1))/X.shape[0]
meanDispersions.append(m_Disp)
plt.rcParams['font.family'] = ['sans-serif']
plt.rcParams['font.sans-serif'] = ['SimHei'] #使折线图显示中文
plt.plot(K,meanDispersions,'bx-')
plt.xlabel('k')
plt.ylabel('平均离差')
plt.title('用肘部方法选择K值')
plt.show()
代码举例2
文件读取的部分可以写一个独立的函数判断更加方便
import numpy as np
import matplotlib.pyplot as plt
# 加载数据
def loadDataSet(fileName):
data = np.loadtxt(fileName,delimiter='\t')
return data
# 欧氏距离计算
def distEclud(x,y):
return np.sqrt(np.sum((x-y)**2)) # 计算欧氏距离
# 为给定数据集构建一个包含K个随机质心的集合
def randCent(dataSet,k):
m,n = dataSet.shape
centroids = np.zeros((k,n))
for i in range(k):
index = int(np.random.uniform(0,m)) #
centroids[i,:] = dataSet[index,:]
return centroids
# k均值聚类
def KMeans(dataSet,k):
m = np.shape(dataSet)[0] #行的数目
# 第一列存样本属于哪一簇
# 第二列存样本的到簇的中心点的误差
clusterAssment = np.mat(np.zeros((m,2)))
clusterChange = True
# 第1步 初始化centroids
centroids = randCent(dataSet,k)
while clusterChange:
clusterChange = False
# 遍历所有的样本(行数)
for i in range(m):
minDist = 100000.0
minIndex = -1
# 遍历所有的质心
#第2步 找出最近的质心
for j in range(k):
# 计算该样本到质心的欧式距离
distance = distEclud(centroids[j,:],dataSet[i,:])
if distance < minDist:
minDist = distance
minIndex = j
# 第 3 步:更新每一行样本所属的簇
if clusterAssment[i,0] != minIndex:
clusterChange = True
clusterAssment[i,:] = minIndex,minDist**2
#第 4 步:更新质心
for j in range(k):
pointsInCluster = dataSet[np.nonzero(clusterAssment[:,0].A == j)[0]] # 获取簇类所有的点
centroids[j,:] = np.mean(pointsInCluster,axis=0) # 对矩阵的行求均值
print("Congratulations,cluster complete!")
return centroids,clusterAssment
def showCluster(dataSet,k,centroids,clusterAssment):
m,n = dataSet.shape
if n != 2:
print("数据不是二维的")
return 1
mark = ['or', 'ob', 'og', 'ok', '^r', '+r', 'sr', 'dr', ', 'pr']
if k > len(mark):
print("k值太大了")
return 1
# 绘制所有的样本
for i in range(m):
markIndex = int(clusterAssment[i,0])
plt.plot(dataSet[i,0],dataSet[i,1],mark[markIndex])
mark = ['Dr', 'Db', 'Dg', 'Dk', '^b', '+b', 'sb', 'db', ', 'pb']
# 绘制质心
for i in range(k):
plt.plot(centroids[i,0],centroids[i,1],mark[i])
plt.show()
dataSet = loadDataSet("kk.txt")
k = 5
centroids,clusterAssment = KMeans(dataSet,k)
showCluster(dataSet,k,centroids,clusterAssment)
实例
已完成有空再整理。