notes_sklearn_交叉验证
目录
- 1. 简介
- 2. 计算CV指标
-
- 2.1 cross_val_score()
- 2.2 预处理 & Pipeline
- 2.3 cross_validate()
- 2.4 cross_val_predict()
- 3. CV迭代器
-
- 3.1 独立同分布数据的CV迭代器
-
- 1)k-fold
- 2)Repeated K-Fold
- 3)Leave One Out (LOO)
- 4)Leave P Out (LPO)
- 5)Random permutations (Shuffle & Split)
- 3.2 基于分类标签的分层CV迭代器
-
- 1)Stratified k-fold
- 2)Stratified Shuffle Split
- 3.3 分组数据的CV迭代器
-
- 1)Group k-fold
- 2)StratifiedGroupKFold
- 3)Leave One Group Out
- 4)Leave P Groups Out
- 5)Group Shuffle Split
- 3.4 其它
-
- 1)Predefined Fold-Splits / Validation-Sets
- 2)time series data
- 4. shuffling
- 5. 置换检验得分
- 参考
1. 简介
模型训练和测试使用相同数据集会导致过拟合。因而通常做法是在进行实验时,划出数据集的一部分作为测试集。train_test_split() 用于完成训练集和测试集的随机拆分。
import numpy as np
from sklearn.model_selection import train_test_split
from sklearn import datasets
from sklearn import svm
X, y = datasets.load_iris(return_X_y=True)
print(X.shape, y.shape)
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.4, random_state=0)
print(X_train.shape, y_train.shape, X_test.shape, y_test.shape)
clf = svm.SVC(kernel='linear', C=1).fit(X_train, y_train)
clf.score(X_test, y_test)
在评估不同超参数时,需要调整超参数,直到达到最佳性能,这也会导致过拟合。为此,可以将数据集的另一部分划分为验证集:在训练集上训练,然后在验证集上评估,最后在测试集上进行最终评估。
然而,通过将可用数据分成三个子集,可用于训练的样本数量减少,而且结果可能取决于特定随机选择的(训练集、验证集)对。一种解决方案是交叉验证(CV)。在进行最终评估时,仍应保留测试集,但在进行 CV 时不再需要验证集。
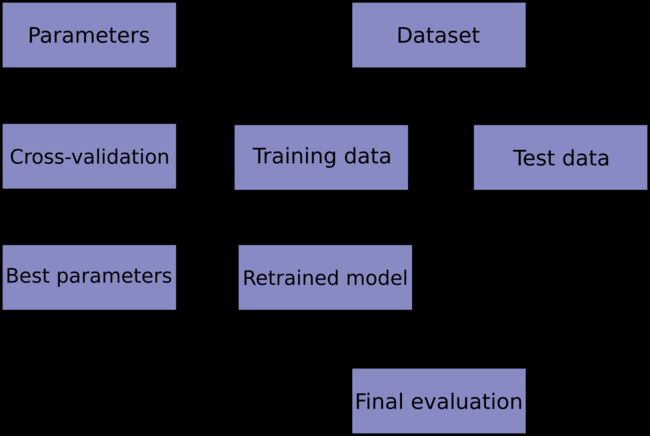
模型训练中典型的交叉验证工作流程图。最佳参数可通过网格搜索技术确定。
在被称为k-fold CV的基本方法中,训练集被分成 k 个较小的集(其它方法见下文,但一般遵循相同原则)。每折都遵循以下程序:
- 使用k-1折(训练集)训练模型
- 在其余数据上(验证集)对模型进行验证
k折交叉验证所报告的性能指标是循环中计算值的平均值。这种方法的计算成本可能很高,但不会浪费太多数据,这在小样本逆推理等的问题上是一大优势。

2. 计算CV指标
2.1 cross_val_score()
使用CV的最简单方法是在estimator和数据集上调用cross_val_score()。下面的例子演示了如何通过拆分数据、拟合模型和连续计算 5 次分数来估计SVM在鸢尾数据集上的准确性。
from sklearn.model_selection import cross_val_score
clf = svm.SVC(kernel='linear', C=1, random_state=42)
scores = cross_val_score(clf, X, y,
cv=5,
scoring='accurary')
print("%0.2f accuracy with a standard deviation of %0.2f" % (scores.mean(), scores.std()))
①当 cv 参数为整数时,默认使用KFold策略或StratifiedKFold策略(ClassifierMix estimator)。也可以通过传递CV迭代器使用其它策略:,见下:
from sklearn.model_selection import ShuffleSplit
#n_samples = X.shape[0]
cv = ShuffleSplit(n_splits=5, test_size=0.3, random_state=0)
cross_val_score(clf, X, y, cv=cv)
或者使用生成器,将(训练、测试)拆分结果作为索引数组,见下:
def custom_cv_2folds(X):
n = X.shape[0]
i = 1
while i <= 2:
idx = np.arange(n * (i - 1) / 2, n * i / 2, dtype=int)
yield idx, idx
i += 1
custom_cv = custom_cv_2folds(X)
cross_val_score(clf, X, y, cv=custom_cv)
②每次CV迭代计算的分数是estimator的score方法。可以使用scoring参数来更改。分类问题中该参数的预定义值如下:

2.2 预处理 & Pipeline
除了estimator的预测需要在测试集上进行,预处理(如标准化、特征选择等)和数据转换也应从训练集中学习,在测试集上预测或转换。
from sklearn import preprocessing
X_train, X_test, y_train, y_test = train_test_split(X, y, test_size=0.4, random_state=0)
scaler = preprocessing.StandardScaler().fit(X_train)
X_train_transformed = scaler.transform(X_train)
clf = svm.SVC(C=1).fit(X_train_transformed, y_train)
X_test_transformed = scaler.transform(X_test)
clf.score(X_test_transformed, y_test)
Pipeline可以简化estimators创建,CV示例如下:
from sklearn.pipeline import make_pipeline
clf = make_pipeline(preprocessing.StandardScaler(), svm.SVC(C=1))
cross_val_score(clf, X, y, cv=cv)
2.3 cross_validate()
| # metrics | rerurn | |
|---|---|---|
| cross_val_score() | 单个 | 列表 |
| cross_validate() | 支持多个 | 字典,包括test_*, fit-times, score-times, … |
示例1. 使用单个metrics
scores = cross_validate(clf, X, y, scoring='precision_macro', cv=5, return_estimator=True)
##return_train_score:是否保存训练集上的得分;
##return_estimator:是否保存在训练集上拟合的estimator;
##return_indices:是否保存训练集和测试集索引;
sorted(scores.keys())
#['estimator', 'fit_time', 'score_time', 'test_score']
示例2. 指定多个metrics:list/tuple/scorer set/dict
from sklearn.model_selection import cross_validate
from sklearn.metrics import recall_score
scoring = ['precision_macro', 'recall_macro']
clf = svm.SVC(kernel='linear', C=1, random_state=0)
scores = cross_validate(clf, X, y, scoring=scoring)
sorted(scores.keys())
#['fit_time', 'score_time', 'test_precision_macro', 'test_recall_macro']
2.4 cross_val_predict()
cross_val_predict()的接口与cross_val_score()类似,但对输入中的每个元素返回该元素的预测值。只有将所有元素准确分配到测试集一次的CV策略才能使用(否则会报错)?
cross_val_score()得到的是CV折的平均值,而cross_val_predict()只返回几个不同模型的标签或概率。因此,cross_val_predict()并不是衡量泛化误差的合适指标。
3. CV迭代器
3.1 独立同分布数据的CV迭代器
独立同分布(i.i.d.):假设所有样本都来自相同的生成过程,且生成过程对过去生成的样本没有记忆。
1)k-fold
KFold将所有样本分成大小相等(如果可能的话)的k组/折/fold。预测函数使用k-1学习,用其余1折测试。

#示例:4个样本的2折CV
import numpy as np
from sklearn.model_selection import KFold
X = ["a", "b", "c", "d"]
kf = KFold(n_splits=2)
for train, test in kf.split(X):
print("%s %s" % (train, test))
2)Repeated K-Fold
RepeatedKFold将K-Fold重复n次,每次重复产生不同的划分。
#示例:重复2次的2折CV
import numpy as np
from sklearn.model_selection import RepeatedKFold
X = np.array([[1, 2], [3, 4], [1, 2], [3, 4]])
rkf = RepeatedKFold(n_splits=2, n_repeats=2, random_state=101)
for train, test in rkf.split(X):
print("%s %s" % (train, test))
3)Leave One Out (LOO)
将设共有n个样本,LeaveOneOut用n-1个样本训练,用剩下的1个测试。该方法不会浪费大量数据。
from sklearn.model_selection import LeaveOneOut
X = [1, 2, 3, 4]
loo = LeaveOneOut()
for train, test in loo.split(X):
print("%s %s" % (train, test))
使用 LOO 进行模型选择时应权衡一些已知的注意事项。相较kfold,无论是模型数,模型训练时的样本数,LOO计算成本更高。在准确性方面,LOO通常导致高方差。但是,如果学习曲线对于相关的训练规模很陡峭,那么 5或10折CV可能会高估泛化误差。一般来说,大多数作者和经验证据都认为5或10折CV应优于LOO。
4)Leave P Out (LPO)
LeavePOut每次使用n-p个样本训练,用剩下的p个样本测试。 不同于LeaveOneOut和KFold,当 p>1 时,测试集将重叠。
#示例:4样本的Leave-2-Out
from sklearn.model_selection import LeavePOut
X = np.ones(4)
lpo = LeavePOut(p=2)
for train, test in lpo.split(X):
print("%s %s" % (train, test))
5)Random permutations (Shuffle & Split)
ShuffleSplit将产生用户指定数量的独立划分。样本首先被打乱,然后划分成训练集和测试集。

from sklearn.model_selection import ShuffleSplit
X = np.arange(10)
ss = ShuffleSplit(n_splits=5, test_size=0.25, random_state=0)
## 可以通过设定random_state伪随机数发生器种子来控制随机性,以保证结果的可重复性。
for train_index, test_index in ss.split(X):
print("%s %s" % (train_index, test_index))
3.2 基于分类标签的分层CV迭代器
有些分类问题的目标类别分布可能会出现严重失衡:例如,负样本可能比正样本多几倍。在这种情况下,建议使用StratifiedKFold和StratifiedShuffleSplit中实现的分层抽样,以确保在每个训练和验证折中大致保留相对类别频率。
1)Stratified k-fold
StratifiedKFold是k-fold的一个变种,返回分层折:每个集合包含的每个目标类别样本的百分比与完整集合大致相同。

#示例:50样本的分层3折
from sklearn.model_selection import StratifiedKFold, KFold
import numpy as np
X, y = np.ones((50, 1)), np.hstack(([0] * 45, [1] * 5))
skf = StratifiedKFold(n_splits=3)
for train, test in skf.split(X, y):
print('train - {} | test - {}'.format(np.bincount(y[train]), np.bincount(y[test])))
kf = KFold(n_splits=3)
for train, test in kf.split(X, y):
print('train - {} | test - {}'.format(np.bincount(y[train]), np.bincount(y[test])))
2)Stratified Shuffle Split
StratifiedShuffleSplit是ShuffleSplit的一个变体,返回分层划分。

3.3 分组数据的CV迭代器
如果数据生成过程具有分组结构(样本来自不同的受试者、实验、测量设备),使用分组交叉验证会更安全。
数据分组和特定领域相关。例如,从多个病人身上收集的医疗数据,每个病人身上都有多个样本。而这些数据可能取决于各个分组。该例中,每个样本的患者ID将是其组ID。
如果想知道在特定组别上训练出来的模型是否能很好地泛化到未见过的组别上,我们需要确保验证折中的所有样本都来自训练折中完全没有呈现的组。
1)Group k-fold
GroupKFold是 k-fold 的一种变体,它可以确保测试集和训练集中不包含同一群体。例如,如果数据来自不同的受试者,每个受试者都有多个样本,如果模型能灵活地从高度个体化的特征中学习,就可能无法泛化到新的受试者。GroupKFold 可以检测这种过拟合。
假设有三个受试者:
from sklearn.model_selection import GroupKFold
X = [0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 8.8, 9, 10]
y = ["a", "b", "b", "b", "c", "c", "c", "d", "d", "d"]
groups = [1, 1, 1, 2, 2, 2, 3, 3, 3, 3]
gkf = GroupKFold(n_splits=3)
for train, test in gkf.split(X, y, groups=groups):
print("%s %s" % (train, test))
每个受试者处于不同的测试折中,同一受试者只能处于测试或训练折中。
需要注意由于数据的不平衡,折的大小并不完全相同。
如果类别比例必须在不同折中保持平衡,那么StratifiedGroupKFold是更好的选择。
与KFold不同,GroupKFold完全没有随机化,而KFold在shuffle=True时是随机化的。?
2)StratifiedGroupKFold
如果数据集不平衡,仅使用GroupKFold可能会产生有偏拆分。而StratifiedGroupKFold是结合了StratifiedKFold和GroupKFold的CV方案。其原理是将每个分组保持在单个划分的同时,尽量保持每个分组中类的分布。
from sklearn.model_selection import StratifiedGroupKFold
X = list(range(18))
y = [1] * 6 + [0] * 12
groups = [1, 2, 3, 3, 4, 4, 1, 1, 2, 2, 3, 4, 5, 5, 5, 6, 6, 6]
sgkf = StratifiedGroupKFold(n_splits=3)
for train, test in sgkf.split(X, y, groups=groups):
print("%s %s" % (train, test))
-
当前的实现在大多数情况下无法实现完全打乱。当 shuffle=True 时,会发生以下情况:
- 打乱所有组。
- 使用稳定排序按类的标准差对组进行排序。
- 遍历排序后的组并分配给折。
这意味着只有类别分布标准差相同的组才会被打乱,当每个组只有一个类别时,这可能会有用。
-
算法会将每个组贪婪地分配到n_splits测试集之一,选择能使各个测试集类别分布差异最小的测试集。组分配从类别频率方差最大的组开始,即先分配在一个或几个类别上达到峰值的大组。
-
从某种意义上说,这种分法是次优的,因为即使可以实现完美的分层,它也可能产生不平衡的分法。如果每个组中的类别分布相对接近,使用
GroupKFold会更好。

3)Leave One Group Out
LeaveOneGroupOut方法在每次划分时,都会保留属于一个特定组的样本。
from sklearn.model_selection import LeaveOneGroupOut
X = [1, 5, 10, 50, 60, 70, 80]
y = [0, 1, 1, 2, 2, 2, 2]
groups = [1, 1, 2, 2, 3, 3, 3]
logo = LeaveOneGroupOut()
for train, test in logo.split(X, y, groups=groups):
print("%s %s" % (train, test))
4)Leave P Groups Out
from sklearn.model_selection import LeavePGroupsOut
X = np.arange(6)
y = [1, 1, 1, 2, 2, 2]
groups = [1, 1, 2, 2, 3, 3]
lpgo = LeavePGroupsOut(n_groups=2)
for train, test in lpgo.split(X, y, groups=groups):
print("%s %s" % (train, test))
5)Group Shuffle Split
GroupShuffleSplit是ShuffleSplit和LeavePGroupsOut组合,可生成一系列随机分区,其中每次分区都会保留一个组的子集。每次训练/测试划分都是独立的,这意味着连续测试集之间不存在任何必然关系。
from sklearn.model_selection import GroupShuffleSplit
X = [0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 0.001]
y = ["a", "b", "b", "b", "c", "c", "c", "a"]
groups = [1, 1, 2, 2, 3, 3, 4, 4]
gss = GroupShuffleSplit(n_splits=4, test_size=0.5, random_state=0)
for train, test in gss.split(X, y, groups=groups):
print("%s %s" % (train, test))

上述分组CV函数也可用于将数据集拆分为训练集和测试集。train_test_split()是ShuffleSplit的封装,因此只能进行分层划分(使用类标签),而不能适用分组。
要进行训练和测试划分,请使用CV划分器split()方法输出的生成器生成的训练和测试集的索引:
import numpy as np
from sklearn.model_selection import GroupShuffleSplit
X = np.array([0.1, 0.2, 2.2, 2.4, 2.3, 4.55, 5.8, 0.001])
y = np.array(["a", "b", "b", "b", "c", "c", "c", "a"])
groups = np.array([1, 1, 2, 2, 3, 3, 4, 4])
train_indx, test_indx = next(GroupShuffleSplit(random_state=7).split(X, y, groups))
X_train, X_test, y_train, y_test = X[train_indx], X[test_indx], y[train_indx], y[test_indx]
3.4 其它
1)Predefined Fold-Splits / Validation-Sets
对于某些数据集,已经存在将数据分成训练和验证折或多个交叉验证折的预定义划分。使用PredefinedSplit可以使用这些折。
例如,在使用验证集时,将属于验证集的所有样本的test_fold设置为0,将所有其他样本的test_fold设置为-1。
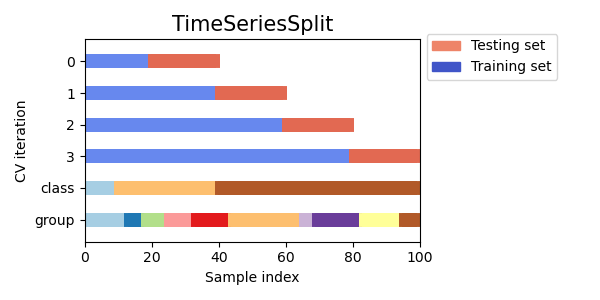
2)time series data
时间序列数据的特点是时间相近的观测值之间存在相关性(自相关性)。然而,经典的交叉验证技术假设样本是独立且同分布的,这将导致时间序列数据的训练实例和测试实例之间存在不合理的相关性(导致泛化误差估计值较差)。因此,要在"未来"观测数据上评估时序模型,而"未来"观测数据至少要与用于训练模型的观测数据相同。为此,TimeSeriesSplit提供了一种解决方案。
TimeSeriesSplit是 k-fold 的一种变体,它将前 k 折作为训练集,将第 k+1 折作为测试集。连续的训练集是之前训练集的超集。此外,它还会将所有剩余数据添加到第一个训练分区,该分区始终用于训练模型。
4. shuffling
如果数据排序不是任意的(例如,具有相同标签的样本是连续的),要获得有意义的CV结果,对数据进行打乱可能是必不可少的。但是,如果样本的分布不是i.i.d,则情况可能相反。例如,如果样本对应新闻文章,并按其发布时间排序,那么打乱很可能会导致模型过拟合和验证分数膨胀:它将在与训练样本相似(时间上接近)的样本上进行测试。
一些CredV迭代器(如 KFold)有一个内置选项,可在划分数据之前对数据索引进行打乱。默认情况下,包括通过在cross_val_score中指定K折CV、网格搜索等,都不会进行数据打乱。train_test_split返回的仍是随机划分。
CV splitters的随机性通过random_state控制,但是split方法也存在随机性??
5. 置换检验得分
permutation_test_score提供了另一种评估分类器性能的方法。它提供了一个基于 permutation 的 p 值,表示分类器观察到的性能在偶然情况下获得的可能性有多大。该检验的零假设是分类器未能利用特征和标签之间的任何统计依赖关系来对测试数据做出正确预测。小p值证明数据集包含特征与标签之间的真实依赖关系,分类器能够利用这种依赖关系获得良好的结果。高p值可能是由于特征和标签之间缺乏依赖性(不同类别之间的特征值没有差异),也可能是由于分类器无法利用数据中的依赖性。后者使用更合适的分类器能导致更低的 p 值。
permutation_test_score通过计算数据的 n_permutations 不同排列来生成零分布。在每次排列中,标签都会被随机洗牌,从而消除了特征与标签之间的任何依赖关系。输出的 p 值是模型平均CV得分优于模型使用原始数据得到的CV得分的排列的分数。要获得可靠的结果,n_permutations 通常应大于100,cv 在3-10倍之间。
CV提供了分类器泛化程度的信息,特别是分类器的预期误差范围。然而,在没有结构的高维数据集上训练的分类器在CV中的表现仍可能会好于预期,这只是随机现象,通常发生在样本少于几百个的小数据集上。
permutation_test_score提供了分类器是否找到了真正的类结构的信息,有助于评估分类器的性能。需要注意,即使数据中只存在微弱的结构,该检验也会产生较低的 p 值,因为在相应的置换数据集中完全不存在结构。因此,该检验只能显示模型可靠地优于随机猜测的情况。此外,permutation_test_score使用蛮力计算,需要拟合 (n_permutations + 1) * n_cv 个模型。因此,它只适用于拟合单个模型速度非常快的小型数据集。
参考
3.1. Cross-validation: evaluating estimator performance
10. Common pitfalls and recommended practices