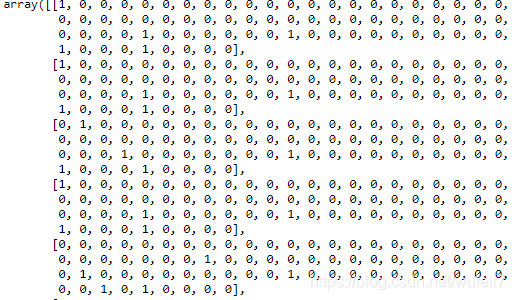Deepchem的分子特征化方法之图神经网络特征化 (ConvMolFeaturizer / WeaveFeaturizer)解析
by wufeil
在Deepchem提供的分子特征化方法中,有两个方法是适用于图神经网络的,分别为:ConvMolFeaturizer 和WeaveFeaturizer。
本期我们就来解析一下这两种图神经网络分子特征化的方法。
一、ConvMolFeaturizer
根绝官方文档的介绍,这是基于“Duvenaud, David K., et al. “Convolutional networks on graphs for learning molecular fingerprints.” Advances in neural information processing systems. 2015.”文章打造的图卷积分子特征化的API接口。其模型原理与fingerprint的对比如下:

仔细一看原理还是比较简单的。感兴趣的可以取查看一下原文献。
ConvMolFeaturizer目前还没有中文名字,这里我暂且叫它图卷积特征化。
有时候,我们会觉得deepchem中的模型并不好用,希望可以用到自己使用torch或者tensorflow编写的模型,但是deepchem中的特征化化以后是一个convmol对象,而没有直接给出邻接矩阵节点特征矩阵等信息;此外,模型好不好,输入也是一个很关键的因素,了解输入包含了哪些信息,在分子特征化过程中丢失了那些信息,是进一步改进模型的基础。所以,这里我们更关心它的用法,特别是其返回的邻接矩阵,节点特征如何提取,以及它到底包含了一个分子什么样的信息。
1.1 ConvMolFeaturizer的用法
import deepchem as dc
import rdkit
#分子指纹表示的化学分子
mols=['C', 'CC', 'CCC', 'CCCN', 'CCNC']
#分子指纹转化为mol对象
mols = [rdkit.Chem.MolFromSmiles(i) for i in mols]
#先补个氢
mols = [rdkit.Chem.AddHs(i) for i in mols]
实例化ConvMolFeaturizer,并调用featurize方法实现分子特征化
#实例化ConvMolFeaturizer
feature = dc.feat.ConvMolFeaturizer()
X = feature.featurize(mols)
X
X是一个列表,保存了四个分子图卷积特征化以后的ConvMol对象。
1.2 提取ConvMolFeaturizer的邻接矩阵和节点特征矩阵
图卷积特征化以后产生的ConvMol对象保存了用于表示分子的邻接矩阵,节点特征矩阵的信息。先来来提取它, 提取成我们经常看见的np.array矩阵。
'''
首先,我们先提取一个ConvMol对象
'''
conmol = X[2]
conmol
conmol就是一个图卷积特征化(ConvMolFeaturizer)产生的ConvMol对象。其原来的分子是一个丙烷CH3CH2CH3。
分子中原子的数量
conmol.get_num_atoms()
输出为11,因为丙烷有11个原子。
邻接矩阵
k = conmol.get_adjacency_list()
k
输出为:

输出还是比较奇怪的,并不是正常的邻接矩阵 (丙烷有11个原子,所以其邻接矩阵应该是一个11*11的矩阵)。
其实,conmol.get_adjacency_list()的返回值的邻接矩阵要这么理解:首先,deepchem已经将原子按照度有小到大的顺序排列,所以最后尾的节点的长度要长。此外,例如矩阵中0号节点,也就是[8]第一行,只与8号节点相邻,在邻接矩阵k中k[0,8]=1。这样子那就好理解了,现在就来复原成我们熟悉的稀疏矩阵表示的邻接矩阵。
import scipy
k = conmol.get_adjacency_list()
#k转换为正常矩阵
k_ = np.zeros((len(k), len(k)))
for i in range(len(k)):
for j in k[i]:
k_[i,j] = 1
k = k_
print (k)
复原的邻接矩阵为:

那么,复原的邻接矩阵对不对呢?我们可以通过DGL构建一个图,然后用networkx来可视化一下,看它是不是一个丙烷。
import dgl
import networkx as nx
k = scipy.sparse.coo_matrix(k)
graph = dgl.DGLGraph(k)
nx.draw(graph.to_networkx(), with_labels=True)
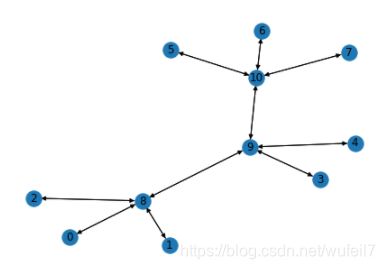
根绝输出的结果来看,这确实是一个丙烷。其中,0~7号节点都是H,8,9,10三个节点分别C。这样看来,我们对图卷积特征化的邻接矩阵的理解是没有错的。
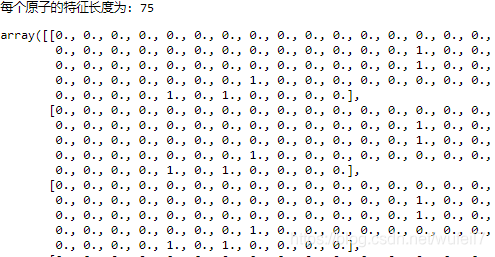
原子节点的特征矩阵
# 每个原子的特征,已经按照度的由小到大顺序进行排列
atom_feature = conmol.get_atom_features()
print('每个原子的特征长度为:',atom_feature.shape[1])
atom_feature
关于节点特征的一些思考
下面来检查一下,8,9,10号节点的特征向量,是否都一致?
通过分子图的构象,8,9,10号节点都是碳原子,但是9号碳原子的与8,10号是有差别的。在图卷积特征化过程中能体现出这一点吗?我们现在来检查一下。
atom_feature_8 = atom_feature[8]
atom_feature_9 = atom_feature[9]
atom_feature_10 = atom_feature[10]
assert all(atom_feature_10 == atom_feature_9)
assert all(atom_feature_8 == atom_feature_9)
运行结果没有报错,图卷积特征化认为,8,9,10号节点都是C原子,其特征都是一致的。
再来检验一次,这次使用分子指纹为:'CCNC’的分子检验一下。
conmol=X[3]
k = conmol.get_adjacency_list()
#获取原子的特征
feature = conmol.get_atom_features()
#k转换为正常矩阵
k_ = np.zeros((len(k), len(k)))
for i in range(len(k)):
for j in k[i]:
k_[i,j] = 1
k = k_
print (k)
k = scipy.sparse.coo_matrix(k)
graph = dgl.DGLGraph(k)
nx.draw(graph.to_networkx(), with_labels=True)
'''
判断一下,N原子旁边相邻的C原子(12号节点)与其他C原子特征之间的差别
'''
feature_c_12 = feature[12]
feature_c_11 = feature[11]
feature_c_10 = feature[10]
assert all(feature_c_12 == feature_c_11)
assert all(feature_c_12 == feature_c_10)
#N原子的特征
feature_N = feature[9]
assert all(feature_N == feature_c_11), 'Atom Features not exactly same'
assert all(feature_N == feature_c_10), 'Atom Features not exactly same'
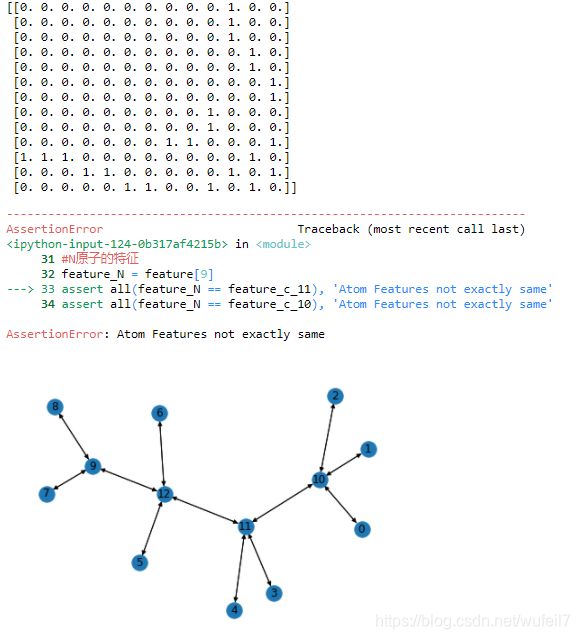
从分子结构上看,9号节点是一个N原子。12,11,10号节点都是C原子。但是,12,11,10号节点的特征都是相同!!!其实我们明眼一看,12号C原子由于旁边是一个N原子,其环境自然是不一样的,其节点特征也应该是不一样的。
也许是,节点的特征仅仅是考虑了键的类型,这里C周围的价键都是单键。
对于不同的原子类型,例如9号节点与11号节点的特征确实是不同的。也就是说图卷积特征化的方法,可以区分分子中不同原子的种类,赋予不同的特征。
小伙伴们可以再拿其他的例子试一下,把结果告诉我。
另外,我们也看到,节点的特征长度高达,75位。这75位我怀疑是基于fingerprint产生的,依据其都为R范围以内的Fingerprint。这一点有人研究明白了也留言告诉我哈。
二、WeaveFeaturizer
WeaveFeaturizer,叫它编织卷积特征化吧。WeaveFeaturizer是另一种deepchem提供的分子图神经网络特征化的方法。其用法与ConvMolFeaturizer类似。
#分子指纹表示的化学分子
mols=['C', 'CC', 'CCC', 'CCCN', 'CCNC']
#分子指纹转化为mol对象
mols = [rdkit.Chem.MolFromSmiles(i) for i in mols]
mols = [rdkit.Chem.AddHs(i) for i in mols]
#实例化ConvMolFeaturizer
feature = dc.feat.WeaveFeaturizer()
X = feature.featurize(mols)
conmol=X[4]
print(type(conmol))
要注意,WeaveFeaturizer编织卷积特征化的方法产生的是deepchem.feat.mol_graphs.WeaveMol对象,与ConvMolFeaturizer产生的ConvMol对象是不一样的。
关于WeaveMol对象中的特征,使用方法,以后在分析。因为deepchem中的这一模块似乎是新开发的,接口信息很不明确,需要看源代码。WeaveFeaturizer好像要比ConvMolFeaturizer要复杂不少。
看了WeaveFeaturizer的源代码发现,WeaveFeaturizer也可以生成节点的特征,节点的特征与ConvMolFeaturizer相同。此外,WeaveFeaturizer也可以生成边的特征。下面就来解析一下WeaveFeaturizer产生的那些特征和用法。
节点特征
conmol.nodes
conmol.get_num_features() #特征的数量也为75个
输出为:75
边的特征
print('边的特征:', conmol.get_pair_features().shape)
输出为:边的特征: (13, 13, 14)
注意:根据原文档的解释,即使两个节点(原子)之间, 即使没有价键连接也是有特征的。
其边特征计算了:
bond_feats = [
bt == Chem.rdchem.BondType.SINGLE, bt == Chem.rdchem.BondType.DOUBLE,
bt == Chem.rdchem.BondType.TRIPLE, bt == Chem.rdchem.BondType.AROMATIC,
bond.GetIsConjugated(),
bond.IsInRing()
]
if use_chirality:
bond_feats = bond_feats + one_of_k_encoding_unk(
str(bond.GetStereo()), GraphConvConstants.possible_bond_stereo)
包括:是否是单键,双键,三键,共轭,环, 还有就是关于手性的信息。
总结:
deepchem中有两种分子适用于图神经网络特征化的方法:ConvMolFeaturizer和WeaveFeaturizer两种。ConvMolFeaturizer仅计算了节点的特征;WeaveFeaturizer不仅计算了节点的特征,还计算了边的特征。注意,ConvMolFeaturizer与WeaveFeaturizer的节点特征的计算方法是一致的。
美中不足的是,这些特征都是one-hot类型的。这些特征实际上就是RDKIT的描述符。