深度学习--AutoEncoder异常值处理
整体的算法思路:
1.将正常样本与异常样本切分为:训练集X,训练集Y,测试集X,测试集Y
2.AutoEncoder建模:建模
3.用正样本数据训练AutoEncoder:因为AutoEncoder是要想办法复现原有数据,因此要确保AutoEncoder看到的都只是自身正常的数据,这样当异常的数据到来时,就会出现很突兀的状况,这也是我们要的效果。
4.计算阈值:因为异常样本会造成很突兀的效果,但是突兀的程度有多大,我们认为是异常样本,需要确定:让测试样本通过Autoencoder模型,复现后的Loss超过阈值,就被认为是异常样本,没有超过阈值则为正常样本
一、载入并处理数据
1.引入库:os 库用于操作文件和目录,pandas 库用于读取 Excel 表格,numpy 库用于进行数值计算,tensorflow 库用于构建自编码器模型,matplotlib 库和 seaborn 库用于数据可视化,sklearn 库用于数据预处理。
2.这里我们把前40%作为正常数据(自定义),把剩下的划分为测试数据
3.MinMaxScaler() 函数用于对数据进行归一化处理,该函数将原始数据缩放到 [0, 1] 范围内。fit_transform() 函数则用于在训练集上计算数据的均值和方差,并将其应用于训练集和测试集上。
4.sample() 函数将训练集中的数据随机打乱,可以提高训练效果。transform() 函数用于将测试集数据进行归一化处理。
import os
import pandas as pd # 引入 Pandas 库,用于读取 Excel 表格
import numpy as np # 引入 NumPy 库,用于数值计算
import tensorflow as tf # 引入 TensorFlow 库,用于构建自动编码器模型
import matplotlib.pyplot as plt
import seaborn as sns
sns.set(color_codes=True)
from sklearn import preprocessing
# 从 Excel 表格中读取数据
df = pd.read_excel('SUM8.07-9.29.xlsx',sheet_name='Analysis')
# 选择要分析的特定列并进行预处理
column_name = 'ene_1' # 指定要分析的列名
data = df[column_name].fillna(0).values.reshape(-1, 1) # 填充缺失值并转换为 NumPy
# 划分测试集和训练集,训练集采取正常数据,测试集采取剩下的数据,按照时间节点划分
# 确定划分点
split_point = int(0.4 * len(data)) # 假设按照前40%作为训练集,后60%作为测试集
# 划分训练集和测试集
dataset_train_data = data[:split_point]
dataset_test_data = data[split_point:]
# 转为DataFrame数据
dataset_train=pd.DataFrame(dataset_train_data)
dataset_test=pd.DataFrame(dataset_test_data)
# 归一化
scaler = preprocessing.MinMaxScaler()
X_train = pd.DataFrame(scaler.fit_transform(dataset_train), # Find the mean and standard deviation of X_train and apply them to X_train
columns=dataset_train.columns,
index=dataset_train.index)
# 随机训练数据
X_train.sample(frac=1)
X_test = pd.DataFrame(scaler.transform(dataset_test),
columns=dataset_test.columns,
index=dataset_test.index)
二、构建模型
自编码模型一般的神经网络,其内部结构呈现一定对称性。这里我们创建三层神经网络:第一层有10个节点(编码器),中间层有2个节点(编码),第三层有10个节点(解码器)
过程:
创建一个空的 Sequential 模型对象 model,并通过调用 AutoEncoder_build 函数来构建自编码器模型,并将构建好的模型赋值给 model。传入的参数包括模型对象 model、训练数据 X_train 和激活函数 act_func。这样就完成了构建自编码器模型的过程。
# 建立自编码模型
def AutoEncoder_build(model, X_train, act_func):
tf.random.set_seed(10) # 设置随机种子,确保结果的可重复性
# 创建一个空的 Sequential 模型对象,用于存储神经网络的结构
model = tf.keras.Sequential()
# 添加一个具有 10 个神经元的全连接层
model.add(tf.keras.layers.Dense(10,activation=act_func, # 使用指定的激活函数 act_func
kernel_initializer='glorot_uniform', # 权重初始化方法为 Glorot 初始化
kernel_regularizer=tf.keras.regularizers.l2(0.0), # 正则化项为 L2 正则化
input_shape=(X_train.shape[1],) # 输入数据的形状由 X_train.shape[1] 来确定
)
)
model.add(tf.keras.layers.Dense(2,activation=act_func,
kernel_initializer='glorot_uniform'))
model.add(tf.keras.layers.Dense(10,activation=act_func,
kernel_initializer='glorot_uniform'))
# 添加一个具有与输入数据维度相同的输出层(输出层不需要指定激活函数)
model.add(tf.keras.layers.Dense(X_train.shape[1],
kernel_initializer='glorot_uniform'))
model.compile(loss='mse',optimizer='adam') # 设置编译器
print(model.summary())
tf.keras.utils.plot_model(model, show_shapes=True)
return model
# 返回构建好的模型对象
model = tf.keras.Sequential()
model = AutoEncoder_build(model=model,X_train=X_train, act_func='elu')
三、训练模型
1.添加噪声到训练数据 X_train 中,通过将随机生成的服从正态分布的噪声乘以因子 factor,然后与 X_train 相加得到加噪后的训练数据 X_train_noise。np.clip 函数将 X_train_noise 中小于 0 的值设为 0,大于 1 的值设为 1,以保证数据在 [0, 1] 范围内。
2.使用加噪后的训练数据 X_train_noise 和原始训练数据 X_train 进行模型训练。通过调用 model.fit 函数,传入训练数据和相关参数进行训练。其中,batch_size 指定每个批次的样本数,epochs 指定训练的迭代次数,shuffle 参数表示在每个 epoch 结束后是否打乱样本顺序,validation_split 表示从训练数据中划分出一部分作为验证集的比例。verbose 参数设置为 1,表示打印训练过程中的详细信息。
3.定义了一个名为 plot_AE_history 的函数,用于绘制自编码器训练过程的损失曲线。通过使用 plt.plot 函数分别绘制训练损失和验证损失,并设置标签、图例和坐标轴的标签和范围等。
4.这样就完成了自编码器的训练和可视化过程。
def AutoEncoder_main(model,Epochs,BATCH_SIZE,validation_split):
# 添加噪声到训练数据 X_train
factor = 0.5 # 因子
X_train_noise = X_train + factor * np.random.normal(0,1,X_train.shape) # 随机生成的服从正态分布的噪声乘以因子
X_train_noise = np.clip(X_train_noise,0.,1.) # 噪声的训练数据,将 X_train_noise 中小于 0 的值设为 0,大于 1 的值设为 1,以保证数据在 [0, 1] 范围内。
history=model.fit(np.array(X_train_noise),np.array(X_train), # 模型训练
batch_size=BATCH_SIZE, # 每个批次的样本数
epochs=Epochs, # 训练的迭代次数
shuffle=True, # 在每个 epoch 结束后是否打乱样本顺序
validation_split=validation_split, # 从训练数据中划分出一部分作为验证集的比例
verbose = 1) # 打印训练过程中的详细信息
return history
# 绘制自编码器训练过程的损失曲线
def plot_AE_history(history):
plt.plot(history.history['loss'],
'b',
label='Training loss')
plt.plot(history.history['val_loss'],
'r',
label='Validation loss')
plt.legend(loc='upper right')
plt.xlabel('Epochs')
plt.ylabel('Loss, [mse]')
plt.ylim([0,.1])
plt.show()
history = AutoEncoder_main(model=model,Epochs=100,BATCH_SIZE=100,validation_split=0.05)
plot_AE_history(history)
查看误差分布:
X_pred = model.predict(np.array(X_train))
X_pred = pd.DataFrame(X_pred,
columns=X_train.columns)
X_pred.index = X_train.index
scored = pd.DataFrame(index=X_train.index)
scored['Loss_mae'] = np.mean(np.abs(X_pred-X_train), axis = 1)
plt.figure()
sns.distplot(scored['Loss_mae'],
bins = 10,
kde= True,
color = 'blue')
plt.xlim([0.0,.5])
因为该自编码器学习到了“正常数据”的编码格式,所以当一个数据集提供给该自编码器时,它会按照“正常数据”的编码格式去编码和解码。如果解码后的数据集和输入数据集的误差在一定范围内,则表明输入的数据集是“正常的”,否则是“异常的"。所以根据下图,我们知道”正常的数据“编码和解码后的误差分布在0.4以内,所以我们可以认为如果一个新的数据集编码,解码之后的误差超出该范围则为异常数据。
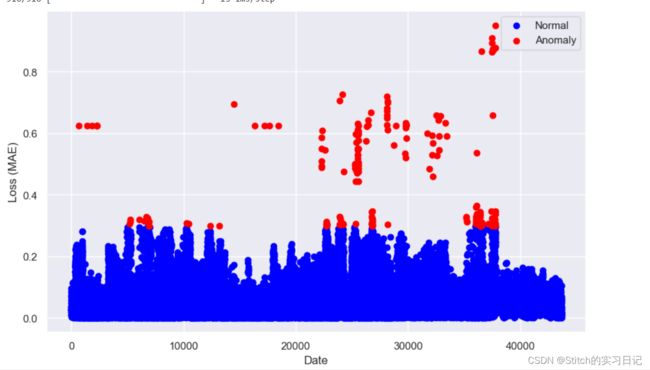
四、测试数据-画图
基于上面的分析,我们可以确定阈值。接下来我们仅仅需要对测试数据集进行预测,然后将预测的结果误差和阈值进行比对,确认是否为正常数据。
X_pred = model.predict(np.array(X_test)) # 对训练集进行预测
X_pred = pd.DataFrame(X_pred, columns=X_test.columns)
X_pred.index = X_test.index
threshod = 0.4
# 计算每个样本的平均绝对误差损失值
scored = pd.DataFrame(index=X_test.index)
scored['Loss_mae'] = np.mean(np.abs(X_pred-X_test), axis=1)
scored['Threshold'] = threshod
scored['Anomaly'] = scored['Loss_mae'] > scored['Threshold'] # 判断是否为异常值
X_pred_train = model.predict(np.array(X_train))
X_pred_train = pd.DataFrame(X_pred_train, columns=X_train.columns)
X_pred_train.index = X_train.index
scored_train = pd.DataFrame(index=X_train.index)
scored_train['Loss_mae'] = np.mean(np.abs(X_pred_train-X_train), axis=1)
scored_train['Threshold'] = threshod
scored_train['Anomaly'] = scored_train['Loss_mae'] > scored_train['Threshold']
# 合并训练集和测试集的异常值检测结果
scored_all = pd.concat([scored_train, scored])
# 绘制异常值和正常值的散点图,横轴为日期,纵轴为损失值(MAE)
anomaly = scored_all[scored_all['Anomaly'] == True]
normal = scored_all[scored_all['Anomaly'] == False]
fig, ax = plt.subplots(figsize=(10, 6))
ax.scatter(normal.index, normal['Loss_mae'], c='blue', label='Normal')
ax.scatter(anomaly.index, anomaly['Loss_mae'], c='red', label='Anomaly')
ax.set_xlabel('Date')
ax.set_ylabel('Loss (MAE)')
ax.legend(loc='upper right')
plt.show()